-
过去几十年,在生物信息领域产出大量基因数据[1-2]。这些基因数据普遍具有样本小、维度高和高噪声等特点[3]。如何处理这些不相关和冗余特征给数据降维带来重大挑战。常见的数据降维包括特征提取[4]和特征选择[5]两类。特征选择由于可以删除无关和冗余特征,同时保留相关原始特征,因此引起许多关注。
在特征选择中主要有数据层面(过滤式方法)和算法层面(包装器方法和嵌入式方法)[6-8]两方面的研究。过滤式特征选择算法凭借其计算成本低、与具体分类器分离及应用领域广等优点,逐渐成为特征选择技术中的研究热点。常见的基于信息论的过滤式特征选择算法包括采用平均冗余策略的特征选择算法(MID[9]、MIQ[9]、JMI[10]和CFR[11]等)和采用“最大最小”极端标准的特征选择算法(CMIM[12]、JMIM[13]和DWUR[14]等)。然而这些算法存在忽视对交互依赖特征相关性和冗余性判断的问题。
因此,本文提出一种利用联合互信息和互信息判断特征与类标签之间相关性和冗余性的特征选择算法(joint feature relevance and redundancy, JFRR)。该算法利用联合互信息计算在已选特征下候选特征与类标签之间的相关性;通过互信息计算已选特征和候选特征的冗余性;通过在9个基准基因数据集的实验对比,该算法(JFRR)优于其他特征选择算法(MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR[15])。
-
设
$ X $ 、$ Y $ 和$ Z $ 是3个离散型变量[16],其中,$ X = \left\{ {{x_1},{x_2}, \cdots ,{x_L}} \right\} $ ,$ Y = \left\{ {{y_1},{y_2}, \cdots ,{y_M}} \right\} $ ,$ Z = \left\{ {{z_1},{z_2}, \cdots ,{z_N}} \right\} $ 。因此,$ X $ 和$ Y $ 之间的互信息定义如下:$$ I\left( {X;Y} \right) = \sum\limits_{i = 1}^L {\sum\limits_{j = 1}^M {p\left( {{x_i},{y_i}} \right){{\log }_2}\frac{{p\left( {{x_i},{y_i}} \right)}}{{p\left( {{x_i}} \right)p\left( {{y_i}} \right)}}} } $$ (1) 式中,
$ p\left( {{x_i},{y_i}} \right) $ 指联合分布;$ p\left( {{x_i}} \right) $ 和$ p( {{y_j}} ) $ 指边际分布。同时,
$ X $ 、$ Y $ 和$ Z $ 的条件互信息定义如下:$$ \begin{split} & I\left( {X;Y|Z} \right) = \sum\limits_{t = 1}^N {p\left( {{z_t}} \right)} \sum\limits_{i = 1}^L {\sum\limits_{j = 1}^M {p\left( {{x_i},{y_i}|{z_t}} \right)} } \times \\ & \qquad\qquad {\log _2}\frac{{p\left( {{x_i},{y_i}|{z_t}} \right)}}{{p\left( {{x_i}|{z_t}} \right)p\left( {{y_i}|{z_t}} \right)}} \end{split} $$ (2) 根据文献[13]的定义,联合互信息定义如下:
$$ I\left( {X,Y;Z} \right) = I\left( {Y;Z} \right) + I\left( {X;Z|Y} \right) $$ (3) -
通过以上描述可知,传统的特征选择算法通常使用最小化冗余项和最大化相关项选择特征子集
$ S $ 。但是由此产生如下问题:1) 当已选特征量增加时,冗余项的大小也会随着相关项的增加而增加。这就存在一些冗余特征可能被选中;2) 在冗余项中,只考虑已选特征和候选特征之间互信息的计算,而忽视类标签,可能会造成已选特征和候选特征共享信息,意味着它们之间存在冗余信息。事实上,它们可能与类标签集合$ C $ 之间共享不同信息。以上问题可能会高估某些候选特征的重要性[17-19]。因此需要考虑,如何在已选特征集合
$ S $ 规模不断增加的情况下,解决$ S $ 与类标签集合$ C $ 的相关性,同时解决候选特征$ {f_k} $ 与$ S $ 的冗余性,以及解决在$ S $ 条件下,候选特征$ {f_k} $ 与类标签$ C $ 的相关性的问题。为此,本文提出一种基于信息论的特征选择算法(JFRR)。该算法充分利用了线性累计加和的方式,具体如下:
$$ {J_{{\rm{JFRR}}}} = \sum\limits_{{f_i} \in S} {\left( {I\left( {{f_k},{f_i};C} \right) - I\left( {{f_k};{f_i}} \right)} \right)} $$ (4) 式中,设
$ F $ 是原始特征集合,$ S \subset F $ ;$J(\cdot)$ 代表评估标准;$ {f_i} \in S,{f_k} \in F - S $ 。通过式(4)可知,JFRR算法利用联合互信息和互信息原理充分考虑
$ S $ 与$ C $ 之间的相关性,$ {f_k} $ 与$ S $ 的冗余性以及在$ S $ 条件下,$ {f_k} $ 与$ C $ 之间的相关性。JFRR算法的具体描述如下。输入:原始特征集合
$ F = \left\{ {{f_1},{f_2}, \cdots {f_n}} \right\} $ ,类标签集合$ C $ ,已选特征子集$ S $ ,阈值$ K $ 输出:最优特征子集
$ S $ 1) 初始化:
$S = \varnothing$ ,$ k = 0 $ 2) for k=1 to n
3) 计算每个特征与标签的互信息值
$ I\left( {C;{f_k}} \right) $ 4) End for
5)
$ {J_{{\rm{JFRR}}}}\left( {{f_k}} \right) = \arg \max \left( {I\left( {{f_k};C} \right)} \right) $ 6) Set
$ F \leftarrow F\backslash \left\{ {{f_k}} \right\} $ 7) Set
$ S \leftarrow \left\{ {{f_k}} \right\} $ 8) while
$ k \leqslant K $ 9) for each
$ {f_k} \in F $ do10) 根据式(1),计算
$ {f_k} $ 与$ {f_i} $ 之间冗余$ I\left( {{f_k};{f_i}} \right) $ 的值;11) 根据式(1),计算
$ {f_i} $ 与$ C $ 之间相关性$ I\left( {{f_i};C} \right) $ 的值;12) 根据式(3),计算
$ {f_k} $ 、$ {f_i} $ 与$ C $ 之间联合互信息$ I\left( {{f_k},{f_i};C} \right) $ 的值;13) 根据式(4),更新
${J_{{\rm{JFRR}}}}\left( {{f_k}} \right)$ 的值;14) end for
15) 根据
$ {J_{{\rm{JFRR}}}}\left( {{f_k}} \right) $ 评估标准,寻找最优的候选特征$ {f_k} $ 16) Set
$ F \leftarrow F\backslash \left\{ {{f_k}} \right\} $ 17) Set
$ S \leftarrow \left\{ {{f_k}} \right\} $ 18) k=k+1
19) end while
从式(4)可知,JFRR算法采用前向顺序搜索特征子集。JFRR算法主要分为3部分。第1部分为1)~7),主要是初始化
$ S $ 集合和计数器$ k $ ;将选择出最大的特征$ {f_k} $ 加入$ S $ 集合,同时$ {f_k} $ 变成已选特征$ {f_i} $ 。第2部分为8)~13),分别计算$ I\left( {{f_i};C} \right) $ 、$ I\left( {{f_k};{f_i}} \right) $ 和$ I\left( {{f_k},{f_i};C} \right) $ 的值。第3部分为14)~19),根据式(4)的选择标准选择$ {f_k} $ ,一直循环到用户指定的阈值$ K $ 就停止循环。 -
本节将JFRR与MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR算法进行对比。具体分类器为:决策树(C4.5)和支持向量机(support vector machine, SVM)。本文的实验环境是Intel-i7处理器,16 GB内存,仿真软件是Python2.7。实验数据集选择ASU和UCI基因数据集[9, 20],详细描述见表1。其中,这9个数据集包含不同的样本数、特征数和类数。样本范围为50~569,特征范围为31~9712,类的范围为2~12,数据类型涉及连续型和离散型。采用6折交叉验证方法进行实验验证。为保证实验公平,分别通过分类评价指标fmc(F1_micro)和pcm(Precision_micro)来评价预测性能。
表 1 数据集描述
序号 数据集 样本数 特征数 分类标签数 数据来源 1 lung 203 3 312 5 ASU 2 lung_discrete 73 325 7 ASU 3 lymphoma 96 4 026 9 ASU 4 Carcinom 174 9 182 11 ASU 5 nci9 60 9 712 9 ASU 6 GLIOMA 50 4 434 4 ASU 7 dermatology 358 35 6 UCI 8 wdbc 569 31 2 UCI 9 arrhythmia 416 279 12 UCI -
为了比较JFRR与MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR算法之间的优劣性,将它们所选的特征子集放到同一个分类器(C4.5和SVM)进行比较,特征子集的规模设置为30。表2选择C4.5分类器。表3选择SVM分类器。在表2~表3中,粗体代表该数据集下特征选择算法中最高平均分类预测值。“Wins/Ties/Losses”描述JFRR算法分别与MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR算法之间的优/平/输个数。
-
在表2中,7个特征选择算法的平均fmc精度值分别为82.459%、80.24%、68.122%、75.356%、68.695%、73.047%和77.296%。JFRR算法获得最高fmc值。同时,从WINS/TIES/LOSSES行的统计结果得出JFRR分别优于MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR算法9、9、9、9、8和6次。
表 2 C4.5分类器的平均fmc性能比较
% 数据集 JFRR MID MIQ CMIM JMIM CFR CMI-MRMR lung 87.555 86.546 79.907 84.632 81.804 75.266 90.526 lung_discrete 86.039 80.722 63.346 77.378 63.531 77.718 84.493 lymphoma 89.322 86.672 67.11 86.794 61.462 85.761 87.835 Carcinom 77.595 72.123 58.313 53.895 58.294 50.884 64.932 nci9 75.554 72.605 48.581 72.903 55.971 69.034 46.108 GLIOMA 80.011 79.448 58.68 61.055 58.496 54.448 74.627 dermatology 94.41 93.017 93.298 93.341 94.175 93.572 94.41 wdbc 95.966 95.789 94.738 95.445 94.557 94.38 95.259 arrhythmia 55.677 55.235 49.122 52.762 49.962 56.36 57.473 平均值 82.459 80.24 68.122 75.356 68.695 73.047 77.296 WINS/TIES/LOSSES 9/0/0 9/0/0 9/0/0 9/0/0 8/0/1 6/1/2 表 3 SVM分类器的平均fmc性能比较
% 数据集 JFRR MID MIQ CMIM JMIM CFR CMI-MRMR lung 91.106 90.111 77.344 89.126 84.694 85.184 92.563 lung_discrete 91.906 87.767 66.539 86.272 61.304 83.985 87.49 lymphoma 95.102 95.102 70.741 93.713 65.171 91.959 95.102 Carcinom 88.653 89.693 74.39 74.702 74.39 62.107 87.826 nci9 83.15 79.304 48.168 76.868 53.494 75.737 48.168 GLIOMA 32.165 32.165 32.165 34.248 30.081 34.248 36.331 dermatology 92.432 91.876 91.876 91.867 97.466 92.432 91.876 wdbc 94.91 94.563 90.677 94.38 90.852 94.559 90.333 arrhythmia 59.445 58.746 57.509 57.509 57.509 57.509 58.464 平均值 80.985 79.925 67.712 77.6317 68.329 75.302 76.461 WINS/TIES/LOSSES 6/2/1 8/1/0 8/0/1 8/0/1 7/1/1 6/1/2 在表3中,7个特征选择算法的平均fmc精度值分别为80.985%、79.925%、67.712%、77.6317%、68.329%、75.302%和76.461%。JFRR算法获得最高fmc值。同时,从WINS/TIES/LOSSES行的统计结果得出JFRR分别优于MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR算法6、8、8、8、7和6次。
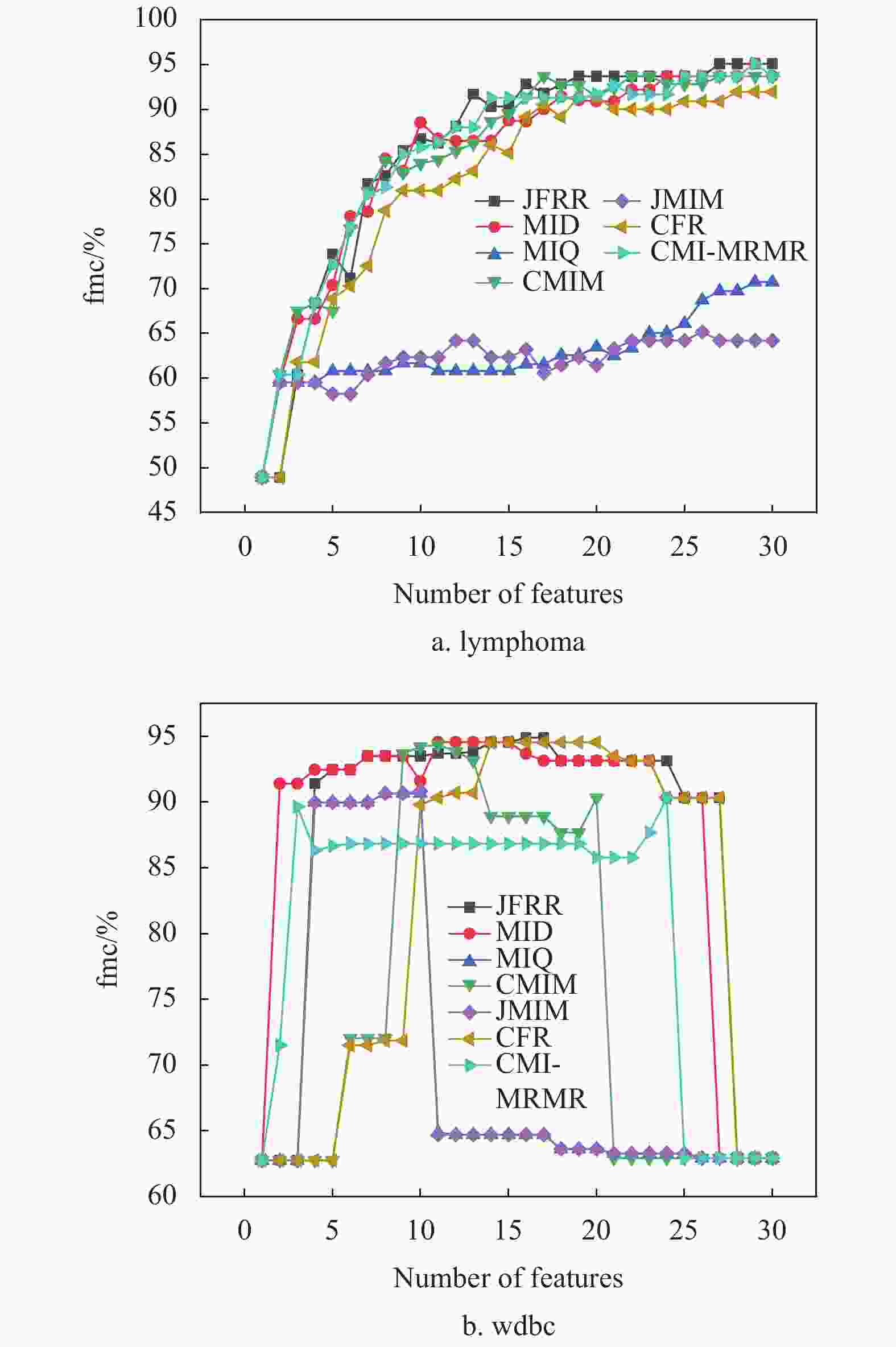
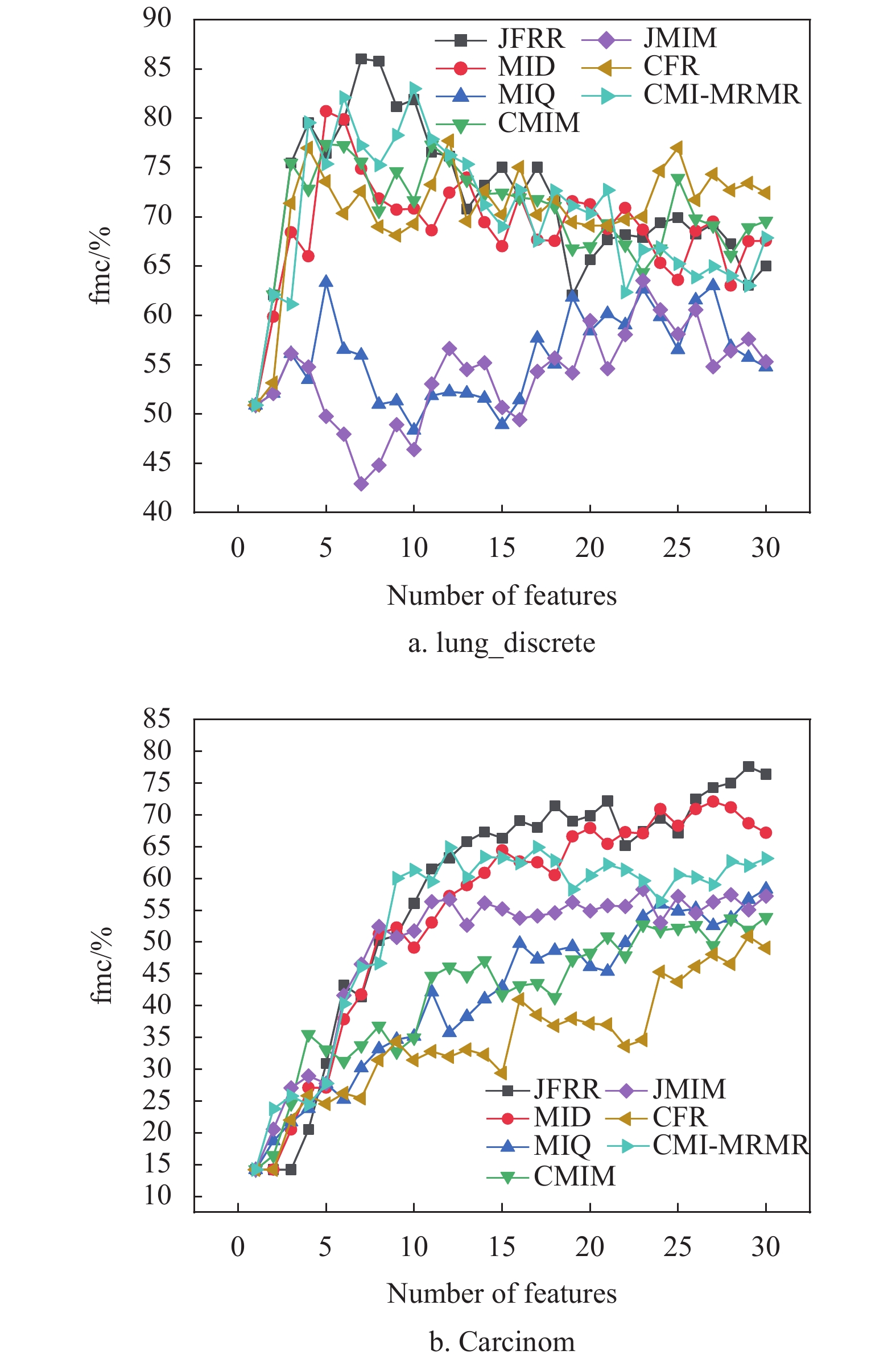
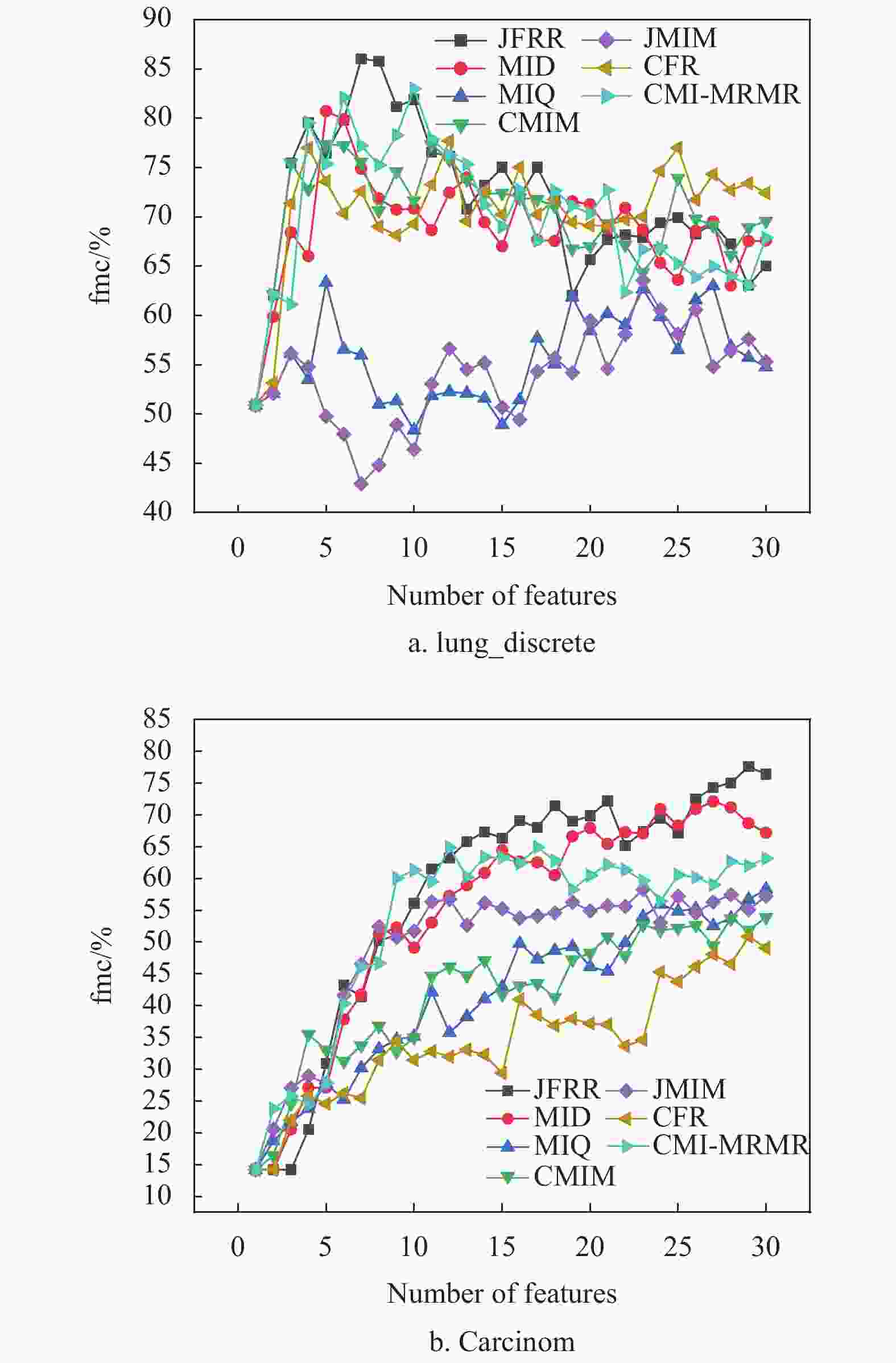
为了进一步比较特征子集对fmc值的影响,图1和图2分别给出部分数据集的fmc性能差异。当数据的维数不断增加时,JFRR算法通过动态调整特征间的相关性和冗余性提升了特征子集的数据质量。图1和图2的实验结果显示,JFRR算法对分类提升的效果明显。并且,JFRR明显优于MID、CMIM、MIQ、JMIM、CFR和CMI-MRMR。
图1是C4.5在高维数据集上的性能比较。在图1a中,JFRR算法的分类fmc值为86.039%,是7种分类算法中最高的,分别比MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR高出5.317%、22.693%、8.661%、22.508%、8.321%和1.546%。在图1b中,JFRR算法的分类fmc值为77.595%,也是7种分类算法中最高的,分别比MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR高出5.472%、19.282%、23.7%、19.301%、26.711%和12.663%。图2是SVM在高维数据集上的性能比较。在图2a中,JFRR算法的分类fmc值为95.102%,是7种分类算法中最高的,分别比MID、MIQ、CMIM、JMIM、 CFR和CMI-MRMR高出0.0%、24.361%、1.389%、29.931%和3.143%和0.0%。在图2b中,JFRR算法的分类fmc值为94.91%,是7种分类算法中最高的,分别比MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR高出0.347%、4.233%、0.53%、4.058%、0.351%和4.577%。
-
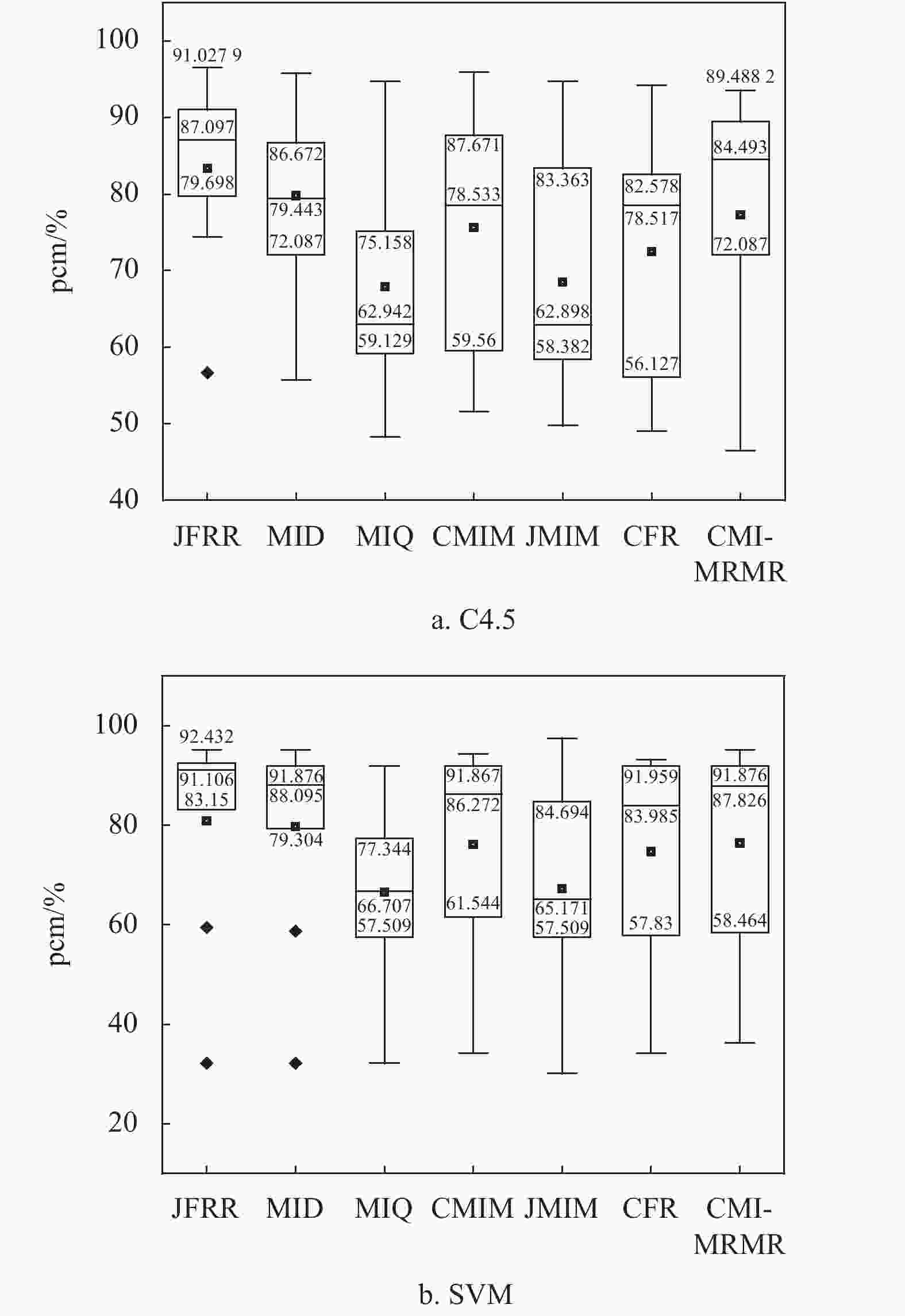
图3为pcm盒图。从图3a中可以得出,在C4.5分类器的pcm盒图中,使用JFRR算法选择出的特征集合在五位数(最小值、四分位数(第25个百分位数)、中位数、四分位数(第75个百分位数)和最大值)中体现出的分类效果都是最优。同时,从图3b中也可以得出,在SVM分类器的pcm盒图中,使用JFRR算法选择出的特征集合在五位数(最小值、四分位数(第25个百分位数)、中位数和四分位数(第75个百分位数))中体现出的分类效果都是最优的效果。
综上,不同分类器表现出的分类结果不尽相同。但是,JFRR算法在fmc和pcm的评价指标值在大多数数据集上都是最好。从C4.5和SVM分类器表现结果中可知,C4.5分类性能明显优于SVM分类性能。
-
计算特征选择算法的运行时间也是衡量特征选择算法重要性的标准之一。JFRR、MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR算法在9个数据集上进行特征排序后得出的运行时间如表4所示。可以看出,JFRR算法的运行时间在可接受的范围之内。
-
本节分析JFRR与MID、CMIM、MIQ、JMIM、CFR 和CMI-MRMR之间在交互特征依赖相关性和冗余性的差异。从表5可以得出,与JFRR相比,MID、MIQ、CMIM和CFR将
$ I\left( {{f_k};C} \right) $ 定义为衡量特征相关性的标准。CMI-MRMR将$ I\left( {{f_i},C|{f_k}} \right) $ 定义为衡量特征相关性的标准。只有JFRR和JMIM将$ I({f_k},{f_i};C) $ 定义为衡量交互特征依赖性动态变化标准。但是,JMIM算法却忽视特征冗余性变化。因此,得出JFRR与其他特征选择算法差异明显。表 4 不同特征选择算法运行时间比较
s 数据集 JFRR MID MIQ CMIM JMIM CFR CMI-MRMR lung 95.568 118.655 57.357 127.739 109.277 126.454 882.251 lung_discrete 2.718 0.969 1.0 2.796 2.721 2.781 31.682 lymphoma 37.508 9.497 9.763 27.825 27.308 27.966 326.731 Carcinom 198.758 88.56 100.252 212.276 369.935 369.935 2298.744 nci9 76.264 27.323 25.375 51.558 50.038 48.722 25.375 GLIOMA 46.543 20.548 22.712 38.615 70.993 65.942 353.598 dermatology 0.868 0.31 0.318 0.55 0.551 0.554 6.811 wdbc 1.434 0.598 0.591 1.31 1.311 1.285 14.378 arrhythmia 15.985 5.952 8.217 19.022 23.048 16.727 214.663 平均值 52.85 30.268 25.065 53.521 72.798 73.374 461.581 表 5 算法比较
算法 考虑特征之间的交互相关性变化 特征冗余性 MID $ I\left( {{f_k};C} \right) $ 是 CMIM $ I\left( {{f_k};C} \right) $ 是 MIQ $ I\left( {{f_k};C} \right) $ 是 JMIM $ I({f_k},{f_i};C) $ 否 CFR $ I\left( {{f_k};C} \right) $ 是 JFRR $ I({f_k},{f_i};C) $ 是 CMI-MRMR $ I\left( {{f_i},C|{f_k}} \right) $ 是 -
随着基因数据中高维特征数据的不断增多,特征间的关系变得越来越复杂(包含大量无关特征和冗余特征)。而传统的特征选择算法往往忽视特征间的相关性和冗余性之间的联系。本文提出一种基于联合互信息的JFRR算法。该算法利用互信息和联合互信息间的关系动态分析和调整特征间以及特征与类标签间的相关信息和冗余信息,从而达到删除无关特征和冗余特征的目的,以此提高特征子集的数据质量。为了全面验证JFRR算法的有效性,实验在9个基因数据集上进行。分别通过使用分类器(C4.5和SVM)和分类准确率指标(fmc和pcm)全面评估所选特征子集的质量。实验结果证明JFRR明显优于MID、MIQ、CMIM、JMIM、CFR和CMI-MRMR等6种特征选择算法。
但在一些基因数据中,JFRR算法仍旧存在选择出的特征子集不理想的情况。未来的工作将进一步研究和改进互信息和联合互信息的关系,并以此优化JFRR算法,同时在更广泛的基因数据集中对算法进行验证,以此提高分类预测精度。
An Algorithm for Cross-Dependent Feature Selection of Genetic Data
-
摘要: 特征选择是生物信息领域中数据预处理阶段必不可少的步骤。传统特征选择算法忽视了特征之间的依赖相关性和冗余性,因此提出一种联合互信息的特征选择算法(JFRR)。该算法利用互信息计算特征之间的冗余值,并利用联合互信息分别计算已选特征集合、候选特征及类标签之间的相关性。将JFRR与其他6个特征选择算法在2个分类器上,使用9个不同基因数据集,进行分类准确率指标(Precision_micro和F1_micro)验证。实验结果表明,该算法能有效提高分类精度。Abstract: Feature selection is an essential step in the data preprocessing phase in the field of bioinformatics. Traditional feature selection algorithms ignore the problems of dependency relevance and redundancy between features. This paper proposes a joint feature relevance and redundancy (JFRR) algorithm for feature selection. The algorithm uses mutual information to calculate the redundancy values between features and applies joint mutual information to compute the relevance among the set of selected features, candidate features and class labels. Finally, JFRR is validated with the other six feature selection algorithms on two classifiers using nine different gene datasets with classification accuracy metrics (Precision_micro and F1_micro). The experimental results show that the JFRR method can effectively improve classification accuracy.
-
Key words:
- classification /
- feature selection /
- joint mutual information /
- mutual information /
- relevance
-
表 1 数据集描述
序号 数据集 样本数 特征数 分类标签数 数据来源 1 lung 203 3 312 5 ASU 2 lung_discrete 73 325 7 ASU 3 lymphoma 96 4 026 9 ASU 4 Carcinom 174 9 182 11 ASU 5 nci9 60 9 712 9 ASU 6 GLIOMA 50 4 434 4 ASU 7 dermatology 358 35 6 UCI 8 wdbc 569 31 2 UCI 9 arrhythmia 416 279 12 UCI 表 2 C4.5分类器的平均fmc性能比较
% 数据集 JFRR MID MIQ CMIM JMIM CFR CMI-MRMR lung 87.555 86.546 79.907 84.632 81.804 75.266 90.526 lung_discrete 86.039 80.722 63.346 77.378 63.531 77.718 84.493 lymphoma 89.322 86.672 67.11 86.794 61.462 85.761 87.835 Carcinom 77.595 72.123 58.313 53.895 58.294 50.884 64.932 nci9 75.554 72.605 48.581 72.903 55.971 69.034 46.108 GLIOMA 80.011 79.448 58.68 61.055 58.496 54.448 74.627 dermatology 94.41 93.017 93.298 93.341 94.175 93.572 94.41 wdbc 95.966 95.789 94.738 95.445 94.557 94.38 95.259 arrhythmia 55.677 55.235 49.122 52.762 49.962 56.36 57.473 平均值 82.459 80.24 68.122 75.356 68.695 73.047 77.296 WINS/TIES/LOSSES 9/0/0 9/0/0 9/0/0 9/0/0 8/0/1 6/1/2 表 3 SVM分类器的平均fmc性能比较
% 数据集 JFRR MID MIQ CMIM JMIM CFR CMI-MRMR lung 91.106 90.111 77.344 89.126 84.694 85.184 92.563 lung_discrete 91.906 87.767 66.539 86.272 61.304 83.985 87.49 lymphoma 95.102 95.102 70.741 93.713 65.171 91.959 95.102 Carcinom 88.653 89.693 74.39 74.702 74.39 62.107 87.826 nci9 83.15 79.304 48.168 76.868 53.494 75.737 48.168 GLIOMA 32.165 32.165 32.165 34.248 30.081 34.248 36.331 dermatology 92.432 91.876 91.876 91.867 97.466 92.432 91.876 wdbc 94.91 94.563 90.677 94.38 90.852 94.559 90.333 arrhythmia 59.445 58.746 57.509 57.509 57.509 57.509 58.464 平均值 80.985 79.925 67.712 77.6317 68.329 75.302 76.461 WINS/TIES/LOSSES 6/2/1 8/1/0 8/0/1 8/0/1 7/1/1 6/1/2 表 4 不同特征选择算法运行时间比较
s 数据集 JFRR MID MIQ CMIM JMIM CFR CMI-MRMR lung 95.568 118.655 57.357 127.739 109.277 126.454 882.251 lung_discrete 2.718 0.969 1.0 2.796 2.721 2.781 31.682 lymphoma 37.508 9.497 9.763 27.825 27.308 27.966 326.731 Carcinom 198.758 88.56 100.252 212.276 369.935 369.935 2298.744 nci9 76.264 27.323 25.375 51.558 50.038 48.722 25.375 GLIOMA 46.543 20.548 22.712 38.615 70.993 65.942 353.598 dermatology 0.868 0.31 0.318 0.55 0.551 0.554 6.811 wdbc 1.434 0.598 0.591 1.31 1.311 1.285 14.378 arrhythmia 15.985 5.952 8.217 19.022 23.048 16.727 214.663 平均值 52.85 30.268 25.065 53.521 72.798 73.374 461.581 表 5 算法比较
算法 考虑特征之间的交互相关性变化 特征冗余性 MID $ I\left( {{f_k};C} \right) $ 是 CMIM $ I\left( {{f_k};C} \right) $ 是 MIQ $ I\left( {{f_k};C} \right) $ 是 JMIM $ I({f_k},{f_i};C) $ 否 CFR $ I\left( {{f_k};C} \right) $ 是 JFRR $ I({f_k},{f_i};C) $ 是 CMI-MRMR $ I\left( {{f_i},C|{f_k}} \right) $ 是 -
[1] DABBA A, ABDELKAMEL T, SAMY M, et al. Gene selection and classification of microarray data method based on mutual information and moth flame algorithm[J]. Expert Systems with Applications, 2021, 166: 114012. doi: 10.1016/j.eswa.2020.114012 [2] HAMBALI M A, OLADELE T O, ADEWOLE K S. Microarray cancer feature selection: Review, challenges and research directions[J]. International Journal of Cognitive Computing in Engineering, 2020, 1: 78-97. doi: 10.1016/j.ijcce.2020.11.001 [3] 王翔, 胡学钢. 高维小样本分类问题中特征选择研究综述[J]. 计算机应用, 2017, 37(9): 2433-2438. doi: 10.11772/j.issn.1001-9081.2017.09.2433 WANG X, HU X G. Overview on feature selection in high-dimensional and small-sample-size classification[J]. Journal of Computer Applications, 2017, 37(9): 2433-2438. doi: 10.11772/j.issn.1001-9081.2017.09.2433 [4] WANG X, LIU J, CHENG Y, et al. Dual hypergraph regularized PCA for biclustering of tumor gene expression data[J]. IEEE Transactions on Knowledge and Data Engineering, 2019, 31(12): 2292-2303. doi: 10.1109/TKDE.2018.2874881 [5] LIU H, GREGORY D. A semi-parallel framework for greedy information-theoretic feature selection[J]. Information Sciences, 2019, 492: 13-28. doi: 10.1016/j.ins.2019.03.075 [6] CAI J, LUO J W, WANG S L, et al. Feature selection in machine learning: A new perspective[J]. Neurocomputing, 2018, 300: 70-79. doi: 10.1016/j.neucom.2017.11.077 [7] LEE C Y, CAI J Y. LASSO variable selection in data envelopment analysis with small datasets[J]. Omega, 2020, 91: 102019. doi: 10.1016/j.omega.2018.12.008 [8] GAO L Y, WU W G. Relevance assignation feature selection method based on mutual information for machine learning[J]. Knowledge-Based Systems, 2020, 209: 106439. doi: 10.1016/j.knosys.2020.106439 [9] 谢娟英, 王明钊, 周颖, 等. 非平衡基因数据的差异表达基因选择算法研究[J]. 计算机学报, 2019, 42(6): 1232-1251. doi: 10.11897/SP.J.1016.2019.01232 XIE J Y, WANG M Z, ZHOU Y, et al. Differential expression gene selection algorithms for unbalanced gene datasets[J]. Chinese Journal of Computers, 2019, 42(6): 1232-1251. doi: 10.11897/SP.J.1016.2019.01232 [10] MACEDO F, OLIVEIRA M R, PACHECO A, et al. Theoretical foundations of forward feature selection methods based on mutual information[J]. Neurocomputing, 2019, 325: 67-89. doi: 10.1016/j.neucom.2018.09.077 [11] GAO W F, HU L, ZHANG P, et al. Feature selection considering the composition of feature relevancy[J]. Pattern Recognition Letters, 2018, 112: 70-74. doi: 10.1016/j.patrec.2018.06.005 [12] BROWN G, POCOCK A, ZHAO M J, et al. Conditional likelihood maximisation: A unifying framework for information theoretic feature selection[J]. The Journal of Machine Learning Research, 2012, 13: 27-66. [13] BENNASAR M, HICKS Y, SETCHI R. Feature selection using joint mutual information maximisation[J]. Expert Systems with Applications, 2015, 42(22): 8520-8532. doi: 10.1016/j.eswa.2015.07.007 [14] 肖利军, 郭继昌, 顾翔元. 一种采用冗余性动态权重的特征选择算法[J]. 西安电子科技大学学报, 2019, 46(5): 155-161. XIAO L J, GUO J C, GU X Y. Algorithm for selection of features based on dynamic weights using redundancy[J]. Journal of XiDian University. 2019, 46(5): 155-161. [15] GU X Y, GUO J C, XIAO L J, et al. Conditional mutual information-based feature selection algorithm for maximal relevance minimal redundancy[J]. Applied Intelligence, 2022, 52(2): 1436-1447. doi: 10.1007/s10489-021-02412-4 [16] MEYER P E, SCHRETTER C, BONTEMPI G. Information-Theoretic feature selection in microarray data using variable complementarity[J]. IEEE Journal of Selected Topics in Signal Processing, 2008, 2(3): 261-274. doi: 10.1109/JSTSP.2008.923858 [17] ZHANG P, GAO W F. Feature selection considering uncertainty change ratio of the class label[J]. Applied Soft Computing, 2020, 95: 106537. doi: 10.1016/j.asoc.2020.106537 [18] CHE J X, YANG Y L, LI L, et al. Maximum relevance minimum common redundancy feature selection for nonlinear data[J]. Information Sciences, 2017, 409-410: 68-86. doi: 10.1016/j.ins.2017.05.013 [19] ZHANG Y S, ZHANG Q, CHEN Z J, et al. Feature assessment and ranking for classification with nonlinear sparse representation and approximate dependence analysis[J]. Decision Support Systems, 2019, 122: 113064. doi: 10.1016/j.dss.2019.05.004 [20] 谢娟英, 丁丽娟, 王明钊. 基于谱聚类的无监督特征选择算法[J]. 软件学报, 2020, 31(4): 1009-1024. doi: 10.13328/j.cnki.jos.005927 XIE J Y, DING L J, WANG M Z. Spectral clustering based unsupervised feature selection algorithms[J]. Journal of Software, 2020, 31(4): 1009-1024. doi: 10.13328/j.cnki.jos.005927 -

 ISSN
ISSN 



 下载:
下载: