-
PD-1是程序性细胞死亡受体1(programmed cell death protein 1, CD279)的英文简写;而PD-L1(programmed cell death 1 ligand 1, CD274)则是该受体的一型配体。PD-1属CD28家族,主要表达在T细胞上。PD-L1属于B7家族,在黑色素瘤等几十种人类肿瘤组织及肿瘤细胞上高表达。文献[1- 2]的研究表明,阻断PD-1/PD-L1信号通路可激活肿瘤抗原特异性T细胞,对于恶性肿瘤具有较好的治疗作用。因此,PD-1和PD-L1成为了目前肿瘤免疫治疗的热门靶点,在国内外掀起了PD-1/PD-L1通路抑制剂研发的热潮。
迄今为止,针对PD-1靶点的抗体药物纳武单抗(nivolumab)、帕姆单抗(pembrolizumab)和西米普利单抗(cemiplimab)以及针对PD-L1靶点的抗体药物阿替利珠单抗(atezolizumab)、阿维鲁单抗(avelumab)和度伐利尤单抗(durvalumab)已获得美国食品药品监督管理局(FDA)批准上市[3-7],还有许多其他抗体药物正处于临床试验阶段[8]。前期研究表明,虽然阻断PD-1/PD-L1通路的抗体药物对于癌症有一定的治疗效果[9],但存在个体差异大(只对大约20%的患者产生持久反应[10])、易引发不恰当的免疫反应[11]等问题。此外,治疗性抗体开发难度大,生产成本较高[12],同时抗体疗法本身存在一些固有的缺陷,如器官或肿瘤渗透性差、口服生物利用度差等。相较于抗体药物,多肽药物在分子质量水平上与小分子化学药物相近,在生物活性上与蛋白质药物相近,集蛋白质药物与传统化学药物的优点于一身。此外,多肽药物分子还拥有结构小、易改造、易合成等优点。但是,多肽稳定性差、半衰期短,这成为了多肽开发成药物得到应用的重大限制。但目前已有多种长效多肽药物方法如改变构型、化学修饰和融合蛋白技术等可用来延长多肽药物在体内的半衰期[13],因此,有望开发出治疗恶性肿瘤的多肽药物。
高内涵和高通量的表面展示技术(surface display)是一种常用的筛选多肽配体的方法,其中的噬菌体展示技术(phage display)[14]在筛选候选多肽方面具有耗时少、操作简单等优势。在噬菌体展示淘选实验中,先将蛋白质等靶标(target)[15-16]固定,与噬菌体展示文库一起温育,经过几轮选择和增殖的生物淘选(biopanning)[17],得到与靶标特异性结合的噬菌体,最终通过测序确定展示的外源多肽序列。在传统的噬菌体展示实验中,经过几轮生物淘选后,随机挑选出几十或几百个噬菌体克隆进行第一代DNA测序。由于噬菌体展示文库的多样性,低通量的第一代DNA测序仅分析了库容的九牛一毛(<0.01%)。近10年间,高通量测序逐渐应用在噬菌体展示领域,形成的二代噬菌体展示技术(next-generation phage display, NGPD)可一次性分析几百万条序列,保留了文库的原始多样性,并能淘选出代表性不足的克隆,这些克隆在传统噬菌体展示淘选实验中大多丢失[18-20]。而采用二代噬菌体展示技术筛选靶标特异性结合肽,需要的淘选实验轮数更少,可加快发现与开发多肽配体的节奏。此外,二代噬菌体展示技术还能有效抑制靶标无关肽出现在最终的淘选结果中[21]。目前,已报道了一些借助传统噬菌体展示淘选得到的阻断PD-1/PD-L1相互作用的PD-1结合肽与PD-L1结合肽[11, 22-25]。然而,通过检索生物淘选数据库BDB[26-27],发现还未有研究者采用二代噬菌体展示技术淘选PD-1结合肽。
本研究为了获得更多新型的PD-1结合肽,采用重组人PD-1蛋白为靶标,分别淘选Ph.D.-7和Ph.D.-12噬菌体展示文库,获得了400条潜在的PD-1结合肽。然后,对获得的PD-1结合肽数据集进行了聚类分析、氨基酸组成与基于位置的氨基酸偏好性分析,发现了一些PD-1结合肽的序列组成特性。通过模拟位点分析,发现模拟位点残基与PD-L1上的真实表位残基有一定的重叠,揭示了淘选出的PD-1结合肽可能模拟了PD-L1蛋白的部分特性。本文结果将对计算机辅助设计PD-1结合肽有一定的指导意义。淘选出的PD-1结合肽可作为阻断PD-1/PD-L1相互作用的多肽药物开发的候选多肽。
-
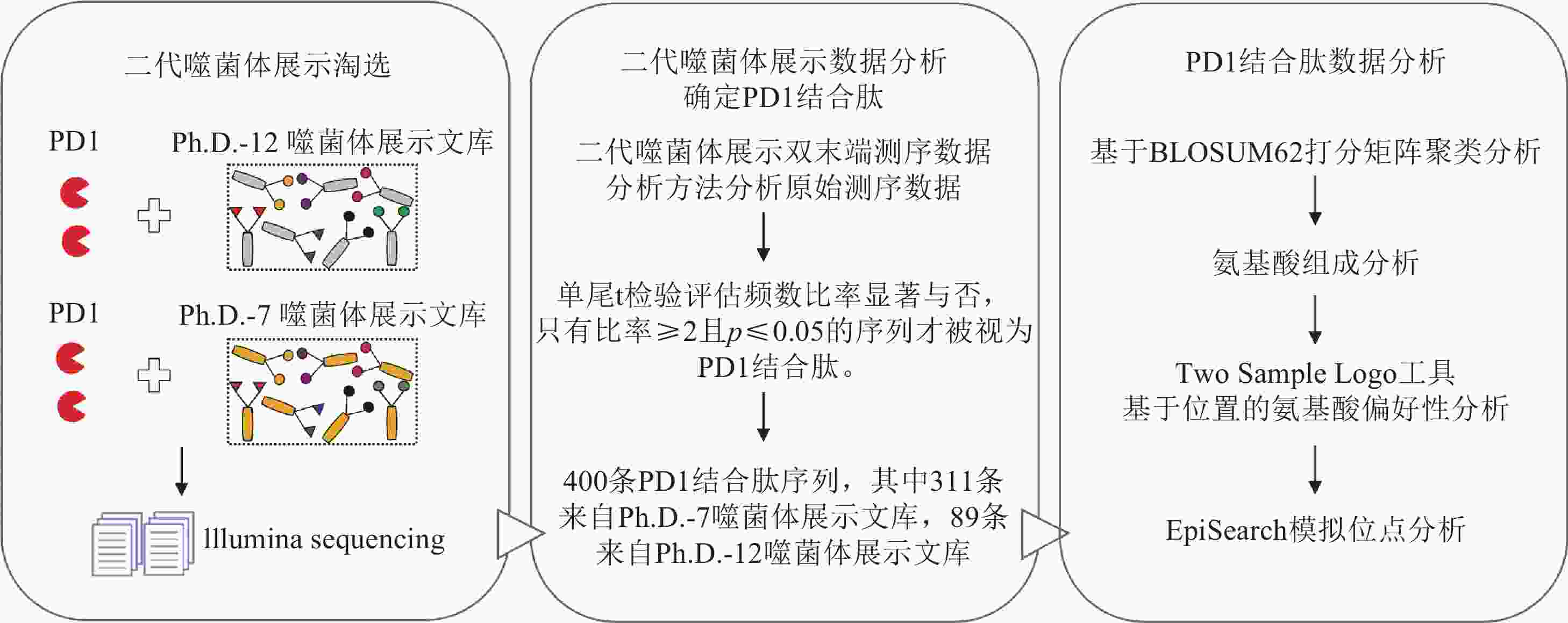
本文采用重组人PD-1蛋白(北京义翘神州科技有限公司)分别淘选Ph.D.-.7和Ph.D.-12噬菌体展示文库(New England Biolabs),获得PD-1结合肽。按照Ph.D.TM Phage Display Libraries Instruction Manual中“Solution-phase Panning with Affinity Bead Capture”的实验流程进行两轮噬菌体展示淘选。在第二轮PD-1淘选实验(R2-PD)时,同时进行了2个对照淘选实验。在第一个对照淘选实验中,将经过第一轮PD-1淘选(R1-PD)富集得到的噬菌体克隆与Dynabeads(Cat# 10-103-D, Invitrogen)进行孵育(R2-DB);在第二个对照淘选实验中,将第一轮淘选后富集的噬菌体克隆与无关的抗FLAG M2单克隆抗体(Cat# F3165, Sigma-Aldrich)进行孵育(R2-UF)。随后,将第1轮之前(R0)的文库、两轮淘选(R1和R2)后的文库,送二代测序。
-
测序完成后,采用二代噬菌体展示双末端测序数据分析方法分析原始测序数据[28]。使用单尾t检验评估多肽在R2-PD淘选实验中的拷贝数比率显著与否,只有序列在R2-PD淘选实验中的拷贝数相较于各个对照样本中的拷贝数比率≥2且p≤0.05才被视为PD-1结合肽。
-
BLOSUM62是生物信息学中应用最广的氨基酸替换打分矩阵[29],常用于序列对比。本研究根据BLOSUM62打分矩阵分别对来自Ph.D.-7噬菌体展示文库的PD-1结合肽和来自Ph.D.-12噬菌体展示文库的PD-1结合肽进行聚类分析。
-
使用Two Sample Logo(TSL)工具来分析多肽序列中特定位置的氨基酸偏好[30]。该工具需要固定长度的多肽序列作为输入,因此分别使用来自Ph.D.-7噬菌体展示文库的PD-1结合肽和非PD-1结合肽以及来自Ph.D.-12噬菌体展示文库的PD-1结合肽和非PD-1结合肽来产生TSL。
-
采用EpiSearch[31]分别对聚类后的每种类别的PD-1结合肽进行构象性表位分析,将PD-1结合肽映射到PD-L1晶体结构(PDBID: 4ZQK,链A)[32]的表面,然后对每种类别中每条肽的映射结果取并集,作为该类别下PD-1识别的PD-L1上的模拟位点。最后对每种类别PD-1结合肽映射结果取并集,作为PD-1识别的PD-L1上的模拟位点。将上述得到的模拟位点与PD-L1上的真实表位进行比较。
-
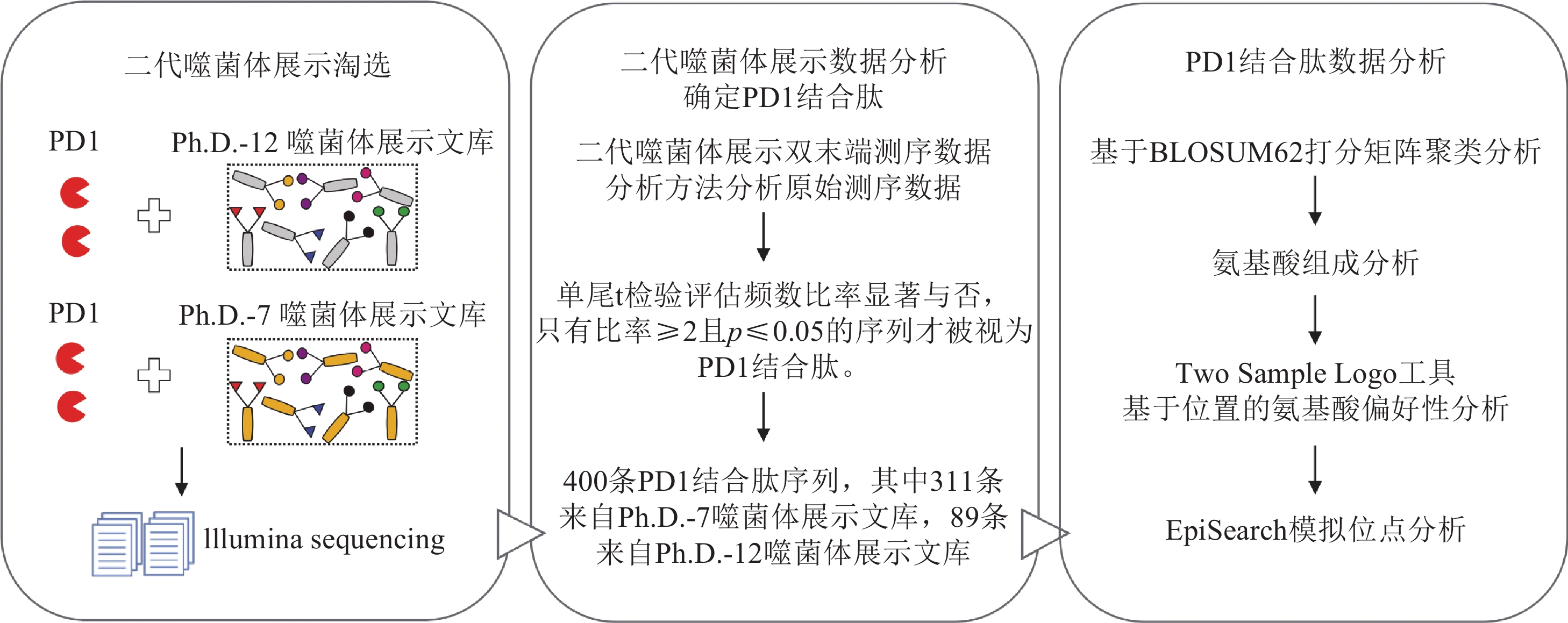
本研究的实验流程如图1所示,首先进行了二代噬菌体展示淘选实验,采用重组人PD-1蛋白为靶标,分别淘选Ph.D.-.7和Ph.D.-12噬菌体展示文库,两轮淘选后获得高度结合PD-1的噬菌体克隆,随后进行Illumina高通量测序。第二阶段主要是确定PD-1结合肽,如果多肽序列在第二轮PD-1淘选后的文库中的拷贝数显著高于各个对照样本中的拷贝数,则认为该多肽为PD-1结合肽。第三阶段主要是对PD-1结合肽数据集进行聚类、氨基酸组成、基于位置的氨基酸偏好性分析以及模拟位点分析。
-
经过两轮的噬菌体展示淘选实验与二代噬菌体展示双末端测序数据分析,最终确定了400条PD-1结合肽序列,其中311条来自Ph.D.-7噬菌体展示文库,89条来自Ph.D.-12噬菌体展示文库。这些多肽在R2-PD中高度富集,而在R0、R1-PD和对照实验R2-DB及R2-UF中拷贝数较低。
随机从余下的Ph.D.-7多肽数据中挑选311条多肽作为非PD-1结合肽,从Ph.D.-12多肽数据中挑选89条多肽作为非PD-1结合肽用于后续特定位置的氨基酸偏好分析。
-
根据BLOSUM62打分矩阵进行聚类分析,来自Ph.D.-7噬菌体展示文库的311条PD-1结合肽聚成了9个类,来自Ph.D.-12噬菌体展示文库的89条PD-1结合肽聚成了4个类。
-
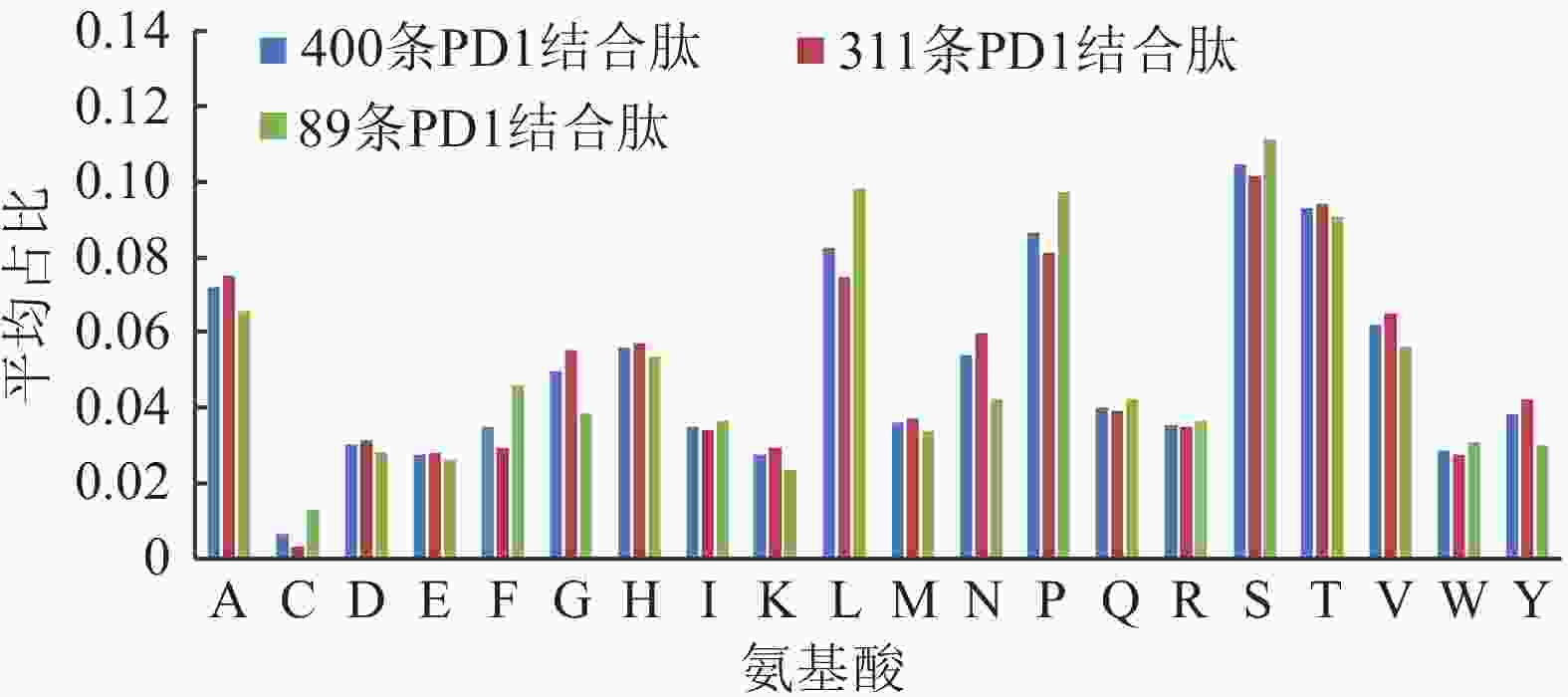
PD-1结合肽的氨基酸组成如图2所示,成分差异清晰可见。分别对3个PD-1结合肽数据集进行了分析,相比其他氨基酸,丙氨酸A、亮氨酸L、脯氨酸P、丝氨酸S和苏氨酸T在PD-1结合肽中含量较高,这表明这些氨基酸在与PD-1结合中起着至关重要的作用。
-
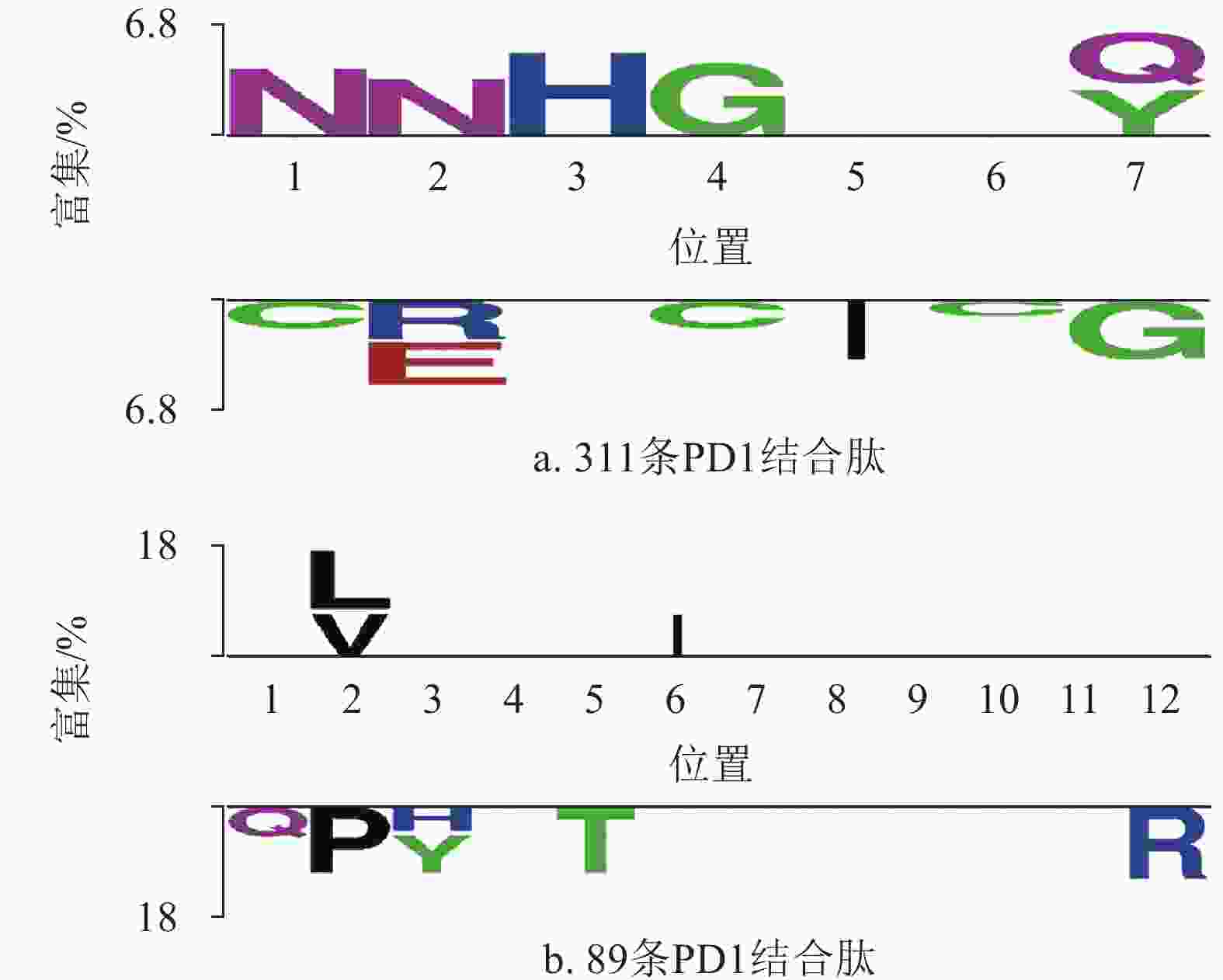
PD-1结合肽数据集基于位置的氨基酸偏好性采用Two Sample Logo软件分析,氨基酸偏好性趋势如图3所示。图3a表明,天冬酰胺N偏向于出现在311条PD-1结合肽的前两个位置,组氨酸H和甘氨酸G分别偏向于出现在第3个和第4个位置,谷氨酰胺Q和酪氨酸Y更倾向于出现在第7个位置。图3b表明,对于来自Ph.D.-12噬菌体展示文库的89条PD-1结合肽,亮氨酸L和缬氨酸V偏好第2个位置,异亮氨酸倾向于出现在第6个位置。
-
使用EpiSearch将PD-1结合肽映射到PD-L1晶体结构(PDBID:3B5H,链A)的表面,分别将来自Ph.D.-12噬菌体展示文库的89条PD-1结合肽和来自Ph.D.-7噬菌体展示文库的311条PD-1结合肽按聚类结果进行分析。311条源自Ph.D.-7噬菌体展示文库的PD-1结合肽分成9个类进行模拟位点分析,EpiSearch均未给出结果,其参数设置为Patch Size = 10,Area cutoff = 5,Accuracy cutoff = 3。如表1所示,来自Ph.D.-12噬菌体展示文库的89条PD-1结合肽分成4个类进行模拟位点分析,在EpiSearch默认参数下,只有Cluster 1与Cluster 2类得到了模拟位点结果;对于Cluster 3与Cluster 4,EpiSearch未给出结果。表中加粗显示的残基同时出现在真实的PD-L1上的表位残基与模拟位点残基中,比较得到的模拟位点残基与PD-L1上的真实表位残基,发现两者有9个位点残基是一致的。上述结果表明,基于噬菌体展示淘选数据的模拟位点分析有一定的可靠性。同时,虽然Cluster 1和Cluster 2按照聚类分析聚成了两类,但是模拟位点分析得到的构象性表位残基是完全一致的,而且模拟位点残基与PD-L1上的真实表位残基具有较高的一致性,因此推断Cluster 1和Cluster 2中的PD-1结合肽可能模拟了PD-L1蛋白的部分特性,这些多肽可作为后期药物开发的候选多肽。此外,根据模拟位点分析结果,Cluster 3 和 Cluster 4 中的 PD-1 结合肽以及来自 Ph.D.-7 噬菌体展示文库的 311 条 PD-1 结合肽的结合位点与真实的表位残基可能不一致,所以这些多肽可能对于后期药物开发的意义不大。
表 1 来自Ph.D.-12噬菌体展示文库的89条PD-1结合肽的模拟位点分析结果
类别名称 模拟位点残基 表位残基个数 Cluster 1 (16 peptides) F19, T20, K41, P43, V44, E45, L48, D49, L50, A51, A52, I54, Y56, F67, H69, S93, M115, S117, Y118, G119, G120, A121, D122 23 Cluster 2 (24 peptides) F19, T20, K41, P43, V44, E45, L48, D49, L50, A51, A52, I54, Y56, F67, H69, S93, M115, S117, Y118, G119, G120, A121, D122 23 Cluster 3 (28 peptides) NA NA Cluster 4 (21 peptides) NA NA 真实的PD-L1上的表位残基
(PDBsum: PDBID: 4ZQK)F19, V23, D26, I54, Y56, E58, Q66, R113, M115, S117, G119, G120, A121, D122, Y123, K124, R125 17 真实表位与模拟位点的交集 F19, I54, Y56, M115, S117, G119, G120, A121, D122 9 -
PD-1结合肽是治疗癌症的潜在治疗药物。本研究通过二代噬菌体展示淘选获得的PD-1结合肽有望开发成阻断PD-1/PD-L1相互作用的候选多肽药物。基于BLOSUM62打分矩阵进行了聚类分析,淘选出的400条PD-1结合肽聚成了13类。对PD-1结合肽数据集进行了氨基酸组成与基于位置的氨基酸偏好性分析,发现丙氨酸A、亮氨酸L、脯氨酸P、丝氨酸S和苏氨酸T在与PD-1结合中起着非常重要的作用。且PD-1结合肽的氨基酸组成有一定的位置偏好性。模拟位点分析揭示了淘选出的PD-1结合肽可能模拟了PD-L1蛋白的部分特性。希望上述结果对PD-1结合肽的计算设计有一定的指导意义。
本研究仍然存在不足之处,未对淘选出的潜在PD-1结合肽进行体外与体内实验,因此未评估能否结合PD-1、能否阻断PD-1/PD-L1相互作用。在后续的工作中,将进一步开展实验验证,并基于机器学习构建PD-1结合肽预测工具,填补快速获取PD-1结合肽计算工具的空白。
Identification of PD-1 Binding Peptides by Next-Generation Phage Display and Mimotope Analysis
-
摘要: 阻断PD-1/PD-L1相互作用可以激活肿瘤浸润性T细胞并恢复其抗肿瘤活性,对于恶性肿瘤具有较好的治疗作用。尽管靶向PD-1/PD-L1通路的抗体药物对癌症有一定的疗效,但现有抗体药物存在生产成本高、个体差异大、引发不恰当的免疫反应等问题,而且还伴随着不可避免的缺陷,如器官或肿瘤渗透性差、口服生物利用度差等,因此迫切需要寻求多肽抑制剂等来弥补当前PD-1/PD-L1相互作用抗体阻断剂的缺点。该文以重组人PD-1蛋白为靶标,首次采用二代噬菌体展示技术淘选Ph.D.-7和Ph.D.-12噬菌体展示文库,获得了400条潜在的PD-1结合肽同时基于BLOSUM62打分矩阵对PD-1结合肽数据集进行了聚类分析。最后,对PD-1结合肽数据集进行了氨基酸组成、基于位置的氨基酸偏好性分析以及模拟位点分析。该文获得的PD-1结合肽有望开发成阻断PD-1/PD-L1相互作用的候选多肽药物,研究结果对于PD-1结合肽的计算设计有一定的指导意义。
-
关键词:
- 氨基酸组成与偏好 /
- 模拟位点分析 /
- 二代噬菌体展示 /
- PD-1结合肽 /
- PD-1/PD-L1相互作用
Abstract: Blocking the interaction of PD-1/PD-L1, which has a preferable therapeutic effect on malignant tumors, can activate tumor-infiltrating T cells and restore their anti-tumor activities. Although antibody drugs targeting the PD-1/PD-L1 pathway have shown certain efficacy in treating cancer patients, existing antibody drugs have problems such as high production costs, large individual differences, and inducing inappropriate immune responses and unavoidable defects such as poor organ or tumor permeability, inferior oral bioavailability, etc. Therefore, peptide inhibitors are urgently needed to make up for the shortcomings of current PD-1/PD-L1 interaction antibody blockers. In this study, we took the recombinant human PD-1 protein as the target and used next-generation phage display to pan the Ph.D.-7 and Ph.D.-12 phage display libraries for the first time, and isolated 400 potential PD-1 binding peptides. We then clustered the PD-1 binding peptide dataset based on the BLOSUM62 scoring matrix. Finally, amino acid composition, position-based amino acid preference and mimotope analysis were performed on the PD-1 binding peptide dataset. The PD-1 binding peptides obtained by NGPD biopanning in this study are expected to be developed into candidate peptide drugs that block the interaction of PD-1/PD-L1. The research results have certain guiding significance for computationally designing PD-1 binding peptides. -
表 1 来自Ph.D.-12噬菌体展示文库的89条PD-1结合肽的模拟位点分析结果
类别名称 模拟位点残基 表位残基个数 Cluster 1 (16 peptides) F19, T20, K41, P43, V44, E45, L48, D49, L50, A51, A52, I54, Y56, F67, H69, S93, M115, S117, Y118, G119, G120, A121, D122 23 Cluster 2 (24 peptides) F19, T20, K41, P43, V44, E45, L48, D49, L50, A51, A52, I54, Y56, F67, H69, S93, M115, S117, Y118, G119, G120, A121, D122 23 Cluster 3 (28 peptides) NA NA Cluster 4 (21 peptides) NA NA 真实的PD-L1上的表位残基
(PDBsum: PDBID: 4ZQK)F19, V23, D26, I54, Y56, E58, Q66, R113, M115, S117, G119, G120, A121, D122, Y123, K124, R125 17 真实表位与模拟位点的交集 F19, I54, Y56, M115, S117, G119, G120, A121, D122 9 -
[1] WOLCHOK J D, CHAN T A. Cancer: Antitumour immunity gets a boost[J]. Nature, 2014, 515(7528): 496-498. doi: 10.1038/515496a [2] TUMEH P C, HARVIEW C L, YEARLEY J H, et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance[J]. Nature, 2014, 515(7528): 568-571. doi: 10.1038/nature13954 [3] GARON E B, RIZVI N A, HUI R, et al. Pembrolizumab for the treatment of non-small-cell lung cancer[J]. N Engl J Med, 2015, 372(21): 2018-2028. doi: 10.1056/NEJMoa1501824 [4] HOOS A. Development of immuno-oncology drugs—From CTLA4 to PD1 to the next generations[J]. Nat Rev Drug Discov, 2016, 15(4): 235-247. doi: 10.1038/nrd.2015.35 [5] BANG Y J, RUIZ E Y, CUTSEM E V, et al. Phase III, randomised trial of avelumab versus physician's choice of chemotherapy as third-line treatment of patients with advanced gastric or gastro-oesophageal junction cancer: Primary analysis of JAVELIN Gastric 300[J]. Ann Oncol, 2018, 29(10): 2052-2060. doi: 10.1093/annonc/mdy264 [6] POSTOW M A, CHESNEY J, PAVLICK A C, et al. Nivolumab and ipilimumab versus ipilimumab in untreated melanoma[J]. N Engl J Med, 2015, 372(21): 2006-2017. doi: 10.1056/NEJMoa1414428 [7] TANG J, YU J X , HUBBARD-LUCEY V M, et al. Trial watch: The clinical trial landscape for PD1/PDL1 immune checkpoint inhibitors[J]. Nat Rev Drug Discov, 2018, 17(12): 854-855. doi: 10.1038/nrd.2018.210 [8] LIN X, LU X, LUO G, et al. Progress in PD-1/PD-L1 pathway inhibitors: From biomacromolecules to small molecules[J]. Eur J Med Chem, 2020, 186: 111876. doi: 10.1016/j.ejmech.2019.111876 [9] HAMANISHI J, MANDAI M, MATSUMURA N, et al. PD-1/PD-L1 blockade in cancer treatment: Perspectives and issues[J]. Int J Clin Oncol, 2016, 21(3): 462-473. doi: 10.1007/s10147-016-0959-z [10] YANG C Y, LIN M W, CHANG Y L, et al. Programmed cell death-ligand 1 expression is associated with a favourable immune microenvironment and better overall survival in stage I pulmonary squamous cell carcinoma[J]. Eur J Cancer, 2016, 57: 91-103. doi: 10.1016/j.ejca.2015.12.033 [11] FISHMAN J A, HOGAN J I, MAUS M V. Inflammatory and infectious syndromes associated with cancer immunotherapies[J]. Clin Infect Dis, 2019, 69(6): 909-920. doi: 10.1093/cid/ciy1025 [12] ANTHONYMACKITZ D, 任丽萍, 谢诗扬, 等. 治疗性抗体可开发性评估研究进展[J]. 电子科技大学学报, 2021, 50(3): 476-480. ANTHONYMACKITZ D, REN L, XIE S, et al. Progress in research on evaluation of developability of therapeutic antibody[J]. Journal of University of Electronic Science and Technology of China, 2021, 50(3): 476-480. [13] CAVACO M, CASTANHO M, NEVES V. Peptibodies: An elegant solution for a long-standing problem[J]. Peptide Science, 2018, 110(1): e23095. [14] SMITH G P. Filamentous fusion phage: Novel expression vectors that display cloned antigens on the virion surface[J]. Science, 1985, 228(4705): 1315-1317. doi: 10.1126/science.4001944 [15] HE B, MAO C, RU B, et al. Epitope mapping of metuximab on CD147 using phage display and molecular docking[J]. Comput Math Methods Med, 2013, 2013: 983829. [16] ZHANG Y, HE B, LIU K, et al. A novel peptide specifically binding to VEGF receptor suppresses angiogenesis in vitro and in vivo[J]. Signal Transduction and Targeted Therapy, 2017, 2: 17010. doi: 10.1038/sigtrans.2017.10 [17] PANDE J, SZEWCZYK M M, GROVER A K. Phage display: Concept, innovations, applications and future[J]. Biotechnol Adv, 2010, 28(6): 849-858. doi: 10.1016/j.biotechadv.2010.07.004 [18] JUDS C, SCHMIDT J, WELLER M G, et al. Combining phage display and next-generation sequencing for materials sciences: A case study on probing polypropylene surfaces[J]. J Am Chem Soc, 2020, 142(24): 10624-10628. doi: 10.1021/jacs.0c03482 [19] ASAR M C, FRANCO A, SOENDERGAARD M. Phage display selection, identification, and characterization of novel pancreatic cancer targeting peptides[J]. Biomolecules, 2020, 10(5): 714. doi: 10.3390/biom10050714 [20] PLEIKO K, POSNOGRAJEVA K, HAUGAS M, et al. In vivo phage display: Identification of organ-specific peptides using deep sequencing and differential profiling across tissues[J]. Nucleic Acids Res, 2021, 49(7): 38. doi: 10.1093/nar/gkaa1279 [21] MATOCHKO W L, LI C S, TANG S K, et al. Prospective identification of parasitic sequences in phage display screens[J]. Nucleic Acids Res, 2014, 42(3): 1784-1798. doi: 10.1093/nar/gkt1104 [22] ZHAI W, ZHOU X, ZHAI M, et al. Blocking of the PD-1/PD-L1 interaction by a novel cyclic peptide inhibitor for cancer immunotherapy[J]. Sci China Life Sci, 2021, 64(4): 548-562. doi: 10.1007/s11427-020-1740-8 [23] LI W, ZHU X, ZHOU X, et al. An orally available PD-1/PD-L1 blocking peptide OPBP-1-loaded trimethyl chitosan hydrogel for cancer immunotherapy[J]. J Control Release, 2021, 334: 376-388. doi: 10.1016/j.jconrel.2021.04.036 [24] LIU H, ZHAO Z, ZHANG L, et al. Discovery of low-molecular weight anti-PD-L1 peptides for cancer immunotherapy[J]. J Immunother Cancer, 2019, 7(1): 270. doi: 10.1186/s40425-019-0705-y [25] TOOYSERKANI R, RASAEE M J, BANDEHPOUR M, et al. Novel anti-PD-L1 peptide selected from combinatorial phage library inhibits tumor cell growth and restores T-cell activity[J]. J Drug Target, 2021, 29(7): 771-782. doi: 10.1080/1061186X.2021.1879087 [26] HE B, CHAI G, DUAN Y, et al. BDB: Biopanning data bank[J]. Nucleic Acids Res, 2016, 44(D1): 1127-1132. doi: 10.1093/nar/gkv1100 [27] HE B, JIANG L, DUAN Y, et al. Biopanning data bank 2018: Hugging next generation phage display[J]. Database the Journal of Biological Databases & Curation, 2018, DOI: 10.1093/database/bay032. [28] HE B, TJHUNG K F, BENNETT N J, et al. Compositional bias in naive and chemically-modified phage-displayed libraries uncovered by paired-end deep sequencing[J]. Sci Rep, 2018, 8(1): 1214. doi: 10.1038/s41598-018-19439-2 [29] PEARSON W R. Selecting the right similarity-scoring matrix[J]. Curr Protoc Bioinformatics, 2013, 43: 351-359. [30] VACIC V, IAKOUCHEVA L M, RADIVOJAC P. Two sample logo: A graphical representation of the differences between two sets of sequence alignments[J]. Bioinformatics, 2006, 22(12): 1536-1537. doi: 10.1093/bioinformatics/btl151 [31] NEGI S S, BRAUN W. Automated detection of conformational epitopes using phage display peptide sequences[J]. Bioinform Biol Insights, 2009, 3: 71-81. [32] ZAK K M, KITEL R, PRZETOCKA S, et al. Structure of the complex of human programmed death 1, PD-1, and its ligand PD-l1[J]. Structure, 2015, 23(12): 2341-2348. doi: 10.1016/j.str.2015.09.010 -

 ISSN
ISSN 



 下载:
下载: