-
结直肠癌是世界上第三大癌症,居于因癌症死亡的第二位。在我国,结直肠癌居肿瘤发病和死因的第二、五位,且呈现发病率不断上升和发病低龄化的特点[1-2]。结直肠癌早期症状不明显,约50%~60%的结直肠癌患者初诊时就已出现转移,导致了该疾病整体预后不良[3]。研究表明,癌细胞与肿瘤微环境之间的相互作用在肿瘤进展中起着重要作用。其中,癌相关成纤维细胞(cancer-associated fibroblasts, CAFs) 和血管内皮细胞(vascular endothelial cells, VECs)可促进肿瘤细胞生长、侵袭和转移,与多种癌症的不良预后有关[4-5]。肿瘤微环境中的免疫细胞浸润,如肿瘤相关巨噬细胞(tumor associated macrophages, TAMs)和肿瘤浸润淋巴细胞(TILs),也与患者的生存有关[6-7]。免疫疗法作为最有前途的方法已被广泛应用于癌症治疗[8]。因此,探索结直肠癌免疫浸润的预后相关性,寻找新的免疫相关治疗靶点,是结直肠癌早期诊断和治疗的迫切需要。
抑制素βA(inhibitors beta A, INHBA)属于转化生长因子-β(TGF-β)超家族成员,既可以通过同型二聚形成激活素A,也可以通过异型二聚形成抑制素[9-10]。激活素A和抑制素是两种密切相关的糖蛋白,具有相反的生物学效应,广泛参与机体的生殖和发育过程[11]。据报道,INHBA作为激活素A和抑制素的亚单位,在多种癌症中具有促癌或抗癌作用。如在肺癌[12]、胃癌[13]、食管癌[14]和尿路上皮癌[15]等恶性肿瘤中,高水平的INHBA促进肿瘤的侵袭和转移,并与患者的不良预后相关。相反,在弥漫性大B细胞淋巴瘤中INHBA表达明显下调,并发挥肿瘤抑制作用[16]。本文前期研究发现INHBA在结直肠癌的肿瘤芽中高表达,而肿瘤芽是评估结直肠癌预后的指标之一。肿瘤芽的数量越多,肿瘤越容易转移,预后也越差[17]。本文通过数据库挖掘的方法全面分析结直肠癌中INHBA基因的表达和启动子甲基化水平,及其表达与结直肠癌患者预后以及肿瘤微环境中免疫浸润水平、CAFs和VECs浸润水平的相关性。
-
Oncomine是目前世界上最大的癌基因芯片数据库和整合数据挖掘平台,涵盖715个数据集和86 733例样本[18]。本文研究利用Oncomine分析不同类型癌症和对应正常组织中INHBA的表达水平。检索阈值设置依据如下:P值为0.001,fold change为2.0,基因排名前10%,数据类型来自mRNA。
-
GEPIA是一个用于分析TCGA和GTEx项目中9 736个肿瘤样本和8 587个正常样本的RNA测序表达数据的公共数据库[19]。本研究通过GEPIA分析不同类型癌症和对应正常组织中INHBA的表达情况,以及INHBA表达水平与结直肠癌的临床病理参数的相关性。另外,借助GEPIA在线工具的单基因分析相关模块分析INHBA在结直肠癌组织和正常组织中表达水平与免疫细胞标记物表达水平的相关性。
-
LinkedOmics是一个公开的平台,包括来自所有32种TCGA癌症类型和10个临床蛋白质组学肿瘤分析联盟(CPTAC)癌症队列的多组学数据[20]。本研究应用LinkedOmics的LinkFinder模块分析INHBA的表达水平与结直肠癌临床病理参数的相关性。
-
UALCAN是一个分析癌症组学数据的综合网络资源[21]。本研究运用UALCAN分析INHBA基因启动子甲基化水平。
-
PrognoScan是一个用于基因预后价值meta分析的数据库[22]。本研究通过PrognoScan评估结直肠癌中INHBA表达与生存时间的关系。Cox P值和95%置信区间的HRs自动计算,阈值P设为0.05。
-
TIMER是一个全面的用于系统分析不同类型癌症免疫浸润的资源[23]。本研究通过TIMER分析了INHBA表达与6种免疫细胞(B细胞、CD8+ T细胞、CD4+ T细胞、巨噬细胞、中性粒细胞、树突状细胞)、CAFs和VECs浸润水平的相关性。在这个分析中,肿瘤纯度是一个主要的混杂因素,因此本文对相关分析做了纯度校正,同时,通过基因相关模块评估了INBHA表达与肿瘤浸润免疫细胞、CAFs和VECs标记物之间的关系。结果采用log2 TPM显示基因表达量,Spearman相关系数和P值自动计算。
-
对Oncomine基因表达结果进行P值、t检验、fold change和基因rank显示。采用Welch’s检验和Dunnett-Tukey-Kramer检验研究INHBA表达与结直肠癌临床病理特征的相关性。PrognoScan的生存结果以危险比(HRs)显示,95%置信区间和基于log-rank检验的P或Cox P值。用Spearman相关性评价基因表达的相关性,P<0.05认为有统计学意义,相关系数Cor>0.1认为是相关的。
-
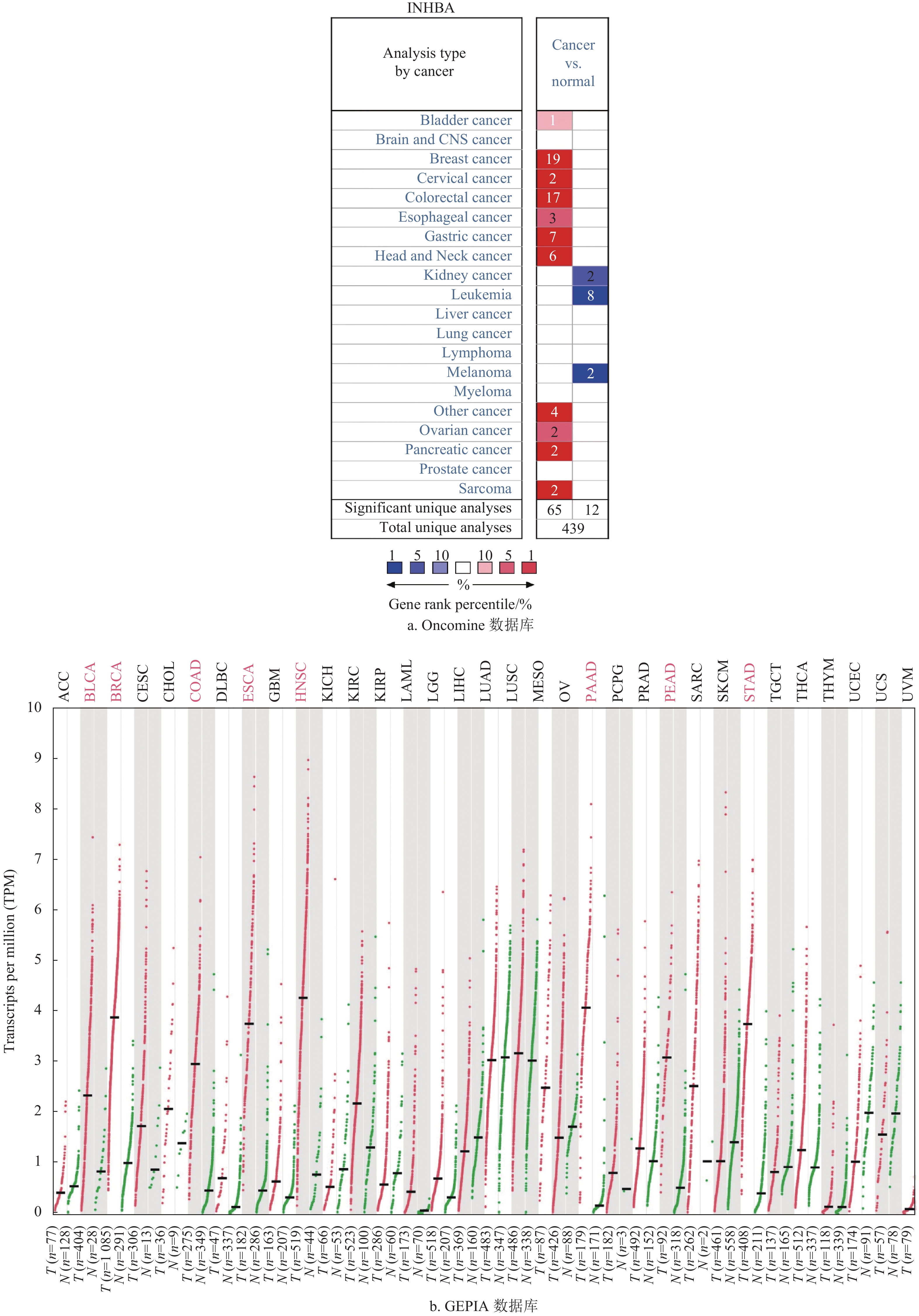
利用Oncomine数据库分析INHBA在不同癌症类型中的表达情况,如图1a所示。结果显示,INHBA在膀胱癌、乳腺癌、宫颈癌、结直肠癌、食管癌、胃癌、头颈癌、卵巢癌、胰腺癌和肉瘤中表达高于正常组织,而在肾癌、白血病和黑色素瘤中表达低于正常组织。通过GEPIA进一步测定TCGA和GTEx项目RNA-seq数据中不同癌症类型的INHBA表达情况,如图1b所示。结果显示,INHBA在膀胱移行细胞癌(BLCA)、乳腺浸润癌(BRCA)、结肠腺癌(COAD)、食管癌(ESCA)、头颈部鳞状细胞癌(HNSC)、胰腺癌(PAAD)、直肠腺癌(READ)和胃腺癌(STAD)中相对于正常癌旁组织呈高表达。综上,INHBA在多种恶性肿瘤中表达上调。
-
在Oncomine数据库中共找到17项INHBA在结直肠癌中表达增高的数据集,0项表达降低的数据集,如图1a。GEPIA数据库同样显示INHBA在结肠癌和直肠癌中的表达水平都显著高于正常肠黏膜组织(P<0.05),如图2a。通过UALCAN数据库分析INHBA基因启动子的甲基化水平。结果显示,与正常肠黏膜相比,INHBA基因启动子的甲基化水平在结肠腺癌和直肠腺癌中都显著降低,如图2b。这提示INHBA在结直肠癌中表达上调很可能与其基因启动子甲基化水平降低有关。接着分别利用GEPIA和LinkedOmics数据库分析INHBA在结直肠癌不同临床分期中的表达情况,结果都显示INHBA的表达与临床分期呈显著正相关。INHBA在Ⅱ、Ⅲ、Ⅳ期的表达明显高于Ⅰ期,如图2c和2d所示。另外,INHBA的表达与结直肠癌的T和N分期也有明显相关性,INHBA在T1、T2、T3、T4期中的表达量明显高于其在Tis期中的表达,如图2e所示,INHBA在N1和N2期中的表达量明显高于其在N0期中的表达,如图2f所示。这些结果提示INHBA参与促进结直肠癌的生长和转移。
-
利用PrognoScan数据库分析了INHBA在结直肠癌患者中的预后价值。结果显示,在采用204926_at和210511_s_at探针的GSE17536数据表中,INHBA高表达组患者总生存期(overall survival, OS)、疾病特异性生存期(disease-specific survival, DSS)和无疾病生存期(disease-free survival, DFS)较INHBA低表达组患者都显著降低,如图3a~图3f所示。在采用204926_at和210511_s_at探针的GSE14333数据表中,INHBA高表达组患者DFS较INHBA低表达组患者也显著降低,如图3g~图3h所示。在采用210511_s_at探针的GSE14333数据表中,INHBA高表达组患者DSS较INHBA低表达组患者同样明显降低,如图3i所示。这表明INHBA高表达与结直肠癌患者预后不良相关。
-
通过TIMER数据库分析得到INHBA表达与结肠癌和直肠癌免疫细胞浸润的相关性结果如图4a和图4b所示,可看出INHBA表达水平与细胞纯度有明显相关性。通过纯度校正后,结肠腺癌和直肠腺癌中INHBA表达水平与CD4+T细胞(Cor=0.469,P=2.14×10−23;Cor=0.301,P=3.25×10−4)、巨噬细胞(macrophages)(Cor=0.547,P=6.46×10−33;Cor=0.493,P=7.23×10−10)、中性粒细胞(neutrophils)(Cor=0.581,P=1.43×10−37;Cor=0.406,P=8.02×10−7)、树突状细胞(dendritic cells, DCs)(Cor=0.576,P=7.42×10−37;Cor=0.458,P=1.43×10−8)的浸润水平都呈显著正相关。这表明INHBA在增加结直肠癌微环境中免疫细胞特别是巨噬细胞的浸润方面发挥了重要作用。
同时,利用TIMER数据库分析得出INHBA表达水平与结肠腺癌和结肠腺癌中单核/巨噬细胞(单核细胞、TAMs、M2巨噬细胞)的常见标志物表达显著相关,而与M1巨噬细胞的常见标志物表达微弱相关,如表1所示。通过GEPIA数据库进一步验证发现,正常组织中INHBA表达与单核细胞、TAMs、M1和M2巨噬细胞的常见标志物表达之间的相关性很低,而结直肠癌中INHBA表达与单核细胞、TAMs和M2巨噬细胞免疫标记物表达显著相关如表2。综上,INHBA在结直肠癌中参与调控巨噬细胞的招募和M1/M2极化。
表 1 TIMER中INHBA与单核/巨噬细胞标志物的相关性分析
Description Gene markers COAD READ None Purity None Purity Cor P Cor P Cor P Cor P Monocyte CD115(CSF1R) 0.654 *** 0.610 *** 0.626 *** 0.541 *** CD16(FCGR3A) 0.657 *** 0.628 *** 0.651 *** 0.570 *** CD86 0.643 *** 0.598 *** 0.611 *** 0.524 *** TAM CD11b(ITGAM) 0.664 *** 0.622 *** 0.663 *** 0.595 *** CCL2 0.647 *** 0.590 *** 0.691 *** 0.633 *** CD68 0.538 *** 0.495 *** 0.470 *** 0.400 *** CD80 0.517 *** 0.479 *** 0.469 *** 0.371 *** M1 iNOS(NOS2) −0.087 0.064 −0.120 0.015 −0.145 0.063 −0.164 0.054 IRF5 0.291 *** 0.305 *** 0.283 ** 0.296 ** CXCL10 0.360 *** 0.300 *** 0.337 *** 0.215 0.011 ROS1 0.107 0.021 0.076 0.124 0.150 0.054 0.085 0.318 M2 CD163 0.668 *** 0.630 *** 0.669 *** 0.609 *** VSIG4 0.603 *** 0.547 *** 0.517 *** 0.453 *** MS4A4A 0.577 *** 0.523 *** 0.600 *** 0.529 *** CD206(MRC1) 0.493 *** 0.441 *** 0.501 *** 0.414 *** None, correlation without adjustment; Purity, correlation adjusted for tumor purity; Cor value of Spearman’s correlation. *P < 0.01; **P < 0.001; ***P < 0.0001. 表 2 GEPIA中INHBA与单核/巨噬细胞标志物的相关性分析
Description Gene markers COAD+READ Normal Tumor Cor P Cor P Monocyte CD115(CSF1R) 0.026 0.86 0.73 *** CD16(FCGR3A) 0.00016 1 0.74 *** CD86 0.15 0.28 0.72 *** TAM CD11b(ITGAM) 0.43 * 0.77 *** CCL2 0.53 *** 0.75 *** CD68 −0.2 0.17 0.58 *** CD80 0.091 0.53 0.61 *** M1 macrophage iNOS(NOS2) −0.11 0.44 −0.058 0.27 IRF5 −0.17 0.24 0.35 *** CXCL10 0.054 0.71 0.43 *** ROS1 −0.17 0.23 0.23 *** M2 macrophage CD163 0.18 0.21 0.71 *** VSIG4 0.019 0.89 0.71 *** MS4A4A 0.078 0.59 0.71 *** CD206(MRC1) 0.23 0.098 0.69 *** Tumor, correlation analysis in tumor tissue of TCGA. Normal, correlation analysis in normal colon and rectal tissue of TCGA; Cor value of Spearman’s correlation. *P < 0.01; **P < 0.001; ***P < 0.0001. -
通过TIMER数据库分析得到结肠腺癌和直肠腺癌中INHBA表达水平与CAFs(EPIC计算法:Cor=0.907,P=1.1×10−104;Cor=0.898,P=1.97×10−33。MCP-Counter计算法:Cor=0.892,P=1.84×10−96;Cor=0.833,P=1.48×10−24)和VECs(EPIC计算法:Cor=0.629,P=9.38×10−32;Cor=0.547,P=2.07×10−8。MCP-Counter计算法:Cor=0.731,P=2.44×10−47;Cor=0.712,P=2.69×10−15)的浸润水平都呈显著正相关,如图5所示。同样,利用TIMER数据库分析发现INHBA表达水平与CAFs和VECs的常见标志物表达都高度相关,如表3所示。这些结果都表明INHBA在增加结直肠癌肿瘤微环境中CAFs和VECs的浸润方面也发挥了重要作用。
表 3 TIMER中INHBA与CAFs、VECs标记物的相关性分析
Description Gene marker COAD READ None Purity None Purity Cor P Cor P Cor P Cor P CAF α-SMA(ACTA2) 0.761 *** 0.729 *** 0.724 *** 0.670 *** FAP 0.775 *** 0.761 *** 0.736 *** 0.726 *** Tenascin-C(TNC) 0.809 *** 0.778 *** 0.764 *** 0.707 *** Periostin(POSTN) 0.774 *** 0.748 *** 0.816 *** 0.774 *** PDGFRA 0.608 *** 0.568 *** 0.688 *** 0.616 *** PDGFRB 0.871 *** 0.858 *** 0.885 *** 0.863 *** THY1 0.808 *** 0.781 *** 0.814 *** 0.767 *** Podoplanin(PDPN) 0.777 *** 0.743 *** 0.784 *** 0.723 *** Integrin β1(ITGB1) 0.619 *** 0.594 *** 0.704 *** 0.679 *** Caveolin-1(CAV1) 0.715 *** 0.667 *** 0.767 *** 0.702 *** VEC CD105(ENG) 0.686 *** 0.638 *** 0.641 *** 0.570 *** CD62E(SELE) 0.635 *** 0.604 *** 0.585 *** 0.504 *** CD106(VCAM1) 0.700 *** 0.658 *** 0.762 *** 0.706 *** CD146(MCAM) 0.701 *** 0.661 *** 0.717 *** 0.661 *** None, correlation without adjustment; Purity, correlation adjusted for tumor purity; Cor value of Spearman’s correlation. *P < 0.01; **P < 0.001; ***P < 0.0001. -
近年来,尽管结直肠癌的早期诊断(肠镜检查)和治疗方面取得了一定的进步,但术后的局部复发和转移仍然是结直肠癌患者的主要死因[24]。转移是恶性肿瘤的主要特征之一,肿瘤一旦发生转移,病人的生存率就会急剧下降[25]。因此,积极寻找诊断和治疗结直肠癌的潜在靶点,对于早期干预结直肠癌和改善结直肠癌患者的预后具有重要的临床意义。
TGF-β信号通路在肿瘤发生发展中是一把双刃剑,早期通过抑制细胞生长从而抑制肿瘤发生,在肿瘤发展的后期可以诱导上皮−间质转化等机制促进肿瘤的侵袭和转移[26]。INHBA作为TGF-β超家族的成员,在不同类型癌症中也具有促癌或抑癌的双重作用。
本文应用生物信息学全面分析了INHBA在结直肠癌中的表达及临床意义。Oncomine和GEPIA在线分析数据表明,INHBA在结直肠癌中的表达明显上调。GEPIA和LinkedOmics数据库分析发现INHBA的表达与结直肠癌临床分期、T和N分期具有显著的正相关性,这就提示INHBA参与促进结直肠癌的生长和转移。在机制方面,通过UALCAN数据库分析发现在结直肠癌组织中INHBA基因启动子甲基化程度明显降低,由于启动子甲基化水平与基因表达呈负相关,这就解释了结直肠癌中该基因表达上调的原因。进一步利用PrognoScan数据库进行生存分析发现,INHBA高表达的结直肠癌患者OS、DSS和DMFS都明显较差。这些结果表明INHBA是结直肠癌预后相关的生物标志物。
肿瘤微环境在肿瘤的发生发展过程中起着重要作用[27]。本文通过TIMER数据库分析发现INHBA表达水平与CD4+T细胞、巨噬细胞、中性粒细胞、树突状细胞的免疫浸润水平都有显著的正相关性,表明INHBA有可能参与结直肠癌细胞的免疫浸润过程。其中,肿瘤微环境中存在最多的炎性细胞是巨噬细胞(肿瘤相关巨噬细胞)。不同微环境中TAMs可以极化为经典活化型(M1 型)和替代活化型(M2 型)两种不同的亚群,其中M1型可促进肿瘤免疫、抑制肿瘤发生;M2 型可以引发免疫逃逸,刺激血管生成,促进肿瘤的生长与转移[28]。TIMER和GEPIA在线分析发现INHBA表达与单核细胞、TAMs和M2 型巨噬细胞的常见标志物表达呈显著正相关,而与M1 型巨噬细胞的常见标志物表达相关性很低,如表3所示,这表明INHBA可能通过调节巨噬细胞M1/M2极化,进而影响TAMs功能并参与结直肠癌的进展。研究表明,M2 型TAMs不但参与多种恶性肿瘤生物行为,并且与肿瘤患者的不良预后密切相关[29]。因此,本研究可为结直肠癌免疫治疗提供新思路和新靶点。除此之外,肿瘤微环境中还存在大量的癌相关成纤维细胞(CAFs)和血管内皮细胞(VECs)。CAFs广泛参与肿瘤的发生发展和转移过程[30]。肿瘤细胞与CAFs和VECs存在相互作用,共同调控肿瘤新生血管形成,且导致不利的临床预后[31]。本文通过TIMER数据库发现结直肠癌中INHBA表达水平与CAFs和VECs的浸润水平呈显著正相关。同时,INHBA表达与CAFs和VECs的常见标志物表达呈显著正相关。其中α-SMA(ACTA2)、FAP、Tenascin-C(TNC)、Periostin(POSTN)、PDGFRA、PDGFRB、THY1、Podoplanin(PDPN)、Integrinβ1(ITGB1)和Caveolin-1(CAV1)被认为是活化的CAFs标记[32]。CD105、CD62E、CD106等是活化VECS的标记[33]。近年来,许多研究表明上皮间质转化(epithelial-mesenchymal transition, EMT)与肿瘤的转移密切相关。其中,TGF-β信号通路是EMT中最为关键的信号通路[34]。作为TGF-β超家族的成员,INHBA表达与上皮间质转化的中心调控因子的表达也呈显著正相关(数据未显示)。这提示INHBA也可通过参与EMT过程促进结直肠癌的转移。
-
综上所述,本研究通过生物信息学的方法发现INHBA在结直肠癌中呈高表达,其高表达与患者的不良预后密切相关。INHBA可能通过参与肿瘤微环境的免疫浸润、CAFs和VECs浸润过程,从而影响结直肠癌的进展。这些研究结果为结直肠癌的诊治提供了潜在的预后指标和治疗靶点。后续将通过基础和临床方面的实验更深入阐明INHBA在结直肠癌发生发展的作用和机制。
Expression and Clinical Significance of INHBA in Colorectal Cancer Based on Bioinformatics Databases
-
摘要: 抑制素βA作为转化生长因子-β(TGF-β)超家族成员,在不同肿瘤中具有促癌或抗癌作用。通过生物信息数据库分析了INHBA在结直肠癌中的表达水平及临床意义。结果显示INHBA在结直肠癌中明显高表达,且其高表达与结直肠癌的临床分期、T分期和N分期呈正相关。INHBA基因启动子的甲基化水平在结肠腺癌和直肠腺癌中都显著降低。生存分析显示,INHBA高表达的结直肠癌患者预后不良。此外,INHBA表达水平与肿瘤微环境中的绝大多数免疫细胞浸润水平呈正相关,尤其是巨噬细胞。同时,INHBA高表达与癌相关成纤维细胞(CAFs)和血管内皮细胞(VECs)高浸润水平也是密切相关。因此,INHBA在结直肠癌中高表达,参与结直肠癌的进展,有望成为预后和治疗的潜在标志物。Abstract: Inhibitors beta A (INHBA), as a member of transforming growth factor-β (TGF-β) superfamily, have pro-cancer or anti-cancer effects in different tumors. This study aims to investigate the expression and clinical significance of INHBA in colorectal cancer through bioinformatics databases. The results showed that INHBA expression was significantly upregulated in colorectal cancer, and its high expression was positively correlated with clinical stages, T stages and N stages of colorectal cancer. The methylation level of INHBA promoter was significantly reduced in both colon and rectal cancer. Survival analysis showed that colorectal cancer patients with high INHBA expression had poor prognosis. Moreover, INHBA expression was positively correlated with the infiltration levels of most immune cells in the tumor microenvironment, especially macrophages. INHBA high expression was also closely related to the high infiltration levels of cancer-associated fibroblasts (CAFs) and vascular endothelial cells (VECs). Taken together, INHBA is highly expressed in colorectal cancer tissues and involved in the development of colorectal cancer, which is expected to be a potential marker for prognosis and treatment.
-
Key words:
- colorectal cancer /
- database /
- INHBA /
- prognosis /
- tumor microenvironment
-
表 1 TIMER中INHBA与单核/巨噬细胞标志物的相关性分析
Description Gene markers COAD READ None Purity None Purity Cor P Cor P Cor P Cor P Monocyte CD115(CSF1R) 0.654 *** 0.610 *** 0.626 *** 0.541 *** CD16(FCGR3A) 0.657 *** 0.628 *** 0.651 *** 0.570 *** CD86 0.643 *** 0.598 *** 0.611 *** 0.524 *** TAM CD11b(ITGAM) 0.664 *** 0.622 *** 0.663 *** 0.595 *** CCL2 0.647 *** 0.590 *** 0.691 *** 0.633 *** CD68 0.538 *** 0.495 *** 0.470 *** 0.400 *** CD80 0.517 *** 0.479 *** 0.469 *** 0.371 *** M1 iNOS(NOS2) −0.087 0.064 −0.120 0.015 −0.145 0.063 −0.164 0.054 IRF5 0.291 *** 0.305 *** 0.283 ** 0.296 ** CXCL10 0.360 *** 0.300 *** 0.337 *** 0.215 0.011 ROS1 0.107 0.021 0.076 0.124 0.150 0.054 0.085 0.318 M2 CD163 0.668 *** 0.630 *** 0.669 *** 0.609 *** VSIG4 0.603 *** 0.547 *** 0.517 *** 0.453 *** MS4A4A 0.577 *** 0.523 *** 0.600 *** 0.529 *** CD206(MRC1) 0.493 *** 0.441 *** 0.501 *** 0.414 *** None, correlation without adjustment; Purity, correlation adjusted for tumor purity; Cor value of Spearman’s correlation. *P < 0.01; **P < 0.001; ***P < 0.0001. 表 2 GEPIA中INHBA与单核/巨噬细胞标志物的相关性分析
Description Gene markers COAD+READ Normal Tumor Cor P Cor P Monocyte CD115(CSF1R) 0.026 0.86 0.73 *** CD16(FCGR3A) 0.00016 1 0.74 *** CD86 0.15 0.28 0.72 *** TAM CD11b(ITGAM) 0.43 * 0.77 *** CCL2 0.53 *** 0.75 *** CD68 −0.2 0.17 0.58 *** CD80 0.091 0.53 0.61 *** M1 macrophage iNOS(NOS2) −0.11 0.44 −0.058 0.27 IRF5 −0.17 0.24 0.35 *** CXCL10 0.054 0.71 0.43 *** ROS1 −0.17 0.23 0.23 *** M2 macrophage CD163 0.18 0.21 0.71 *** VSIG4 0.019 0.89 0.71 *** MS4A4A 0.078 0.59 0.71 *** CD206(MRC1) 0.23 0.098 0.69 *** Tumor, correlation analysis in tumor tissue of TCGA. Normal, correlation analysis in normal colon and rectal tissue of TCGA; Cor value of Spearman’s correlation. *P < 0.01; **P < 0.001; ***P < 0.0001. 表 3 TIMER中INHBA与CAFs、VECs标记物的相关性分析
Description Gene marker COAD READ None Purity None Purity Cor P Cor P Cor P Cor P CAF α-SMA(ACTA2) 0.761 *** 0.729 *** 0.724 *** 0.670 *** FAP 0.775 *** 0.761 *** 0.736 *** 0.726 *** Tenascin-C(TNC) 0.809 *** 0.778 *** 0.764 *** 0.707 *** Periostin(POSTN) 0.774 *** 0.748 *** 0.816 *** 0.774 *** PDGFRA 0.608 *** 0.568 *** 0.688 *** 0.616 *** PDGFRB 0.871 *** 0.858 *** 0.885 *** 0.863 *** THY1 0.808 *** 0.781 *** 0.814 *** 0.767 *** Podoplanin(PDPN) 0.777 *** 0.743 *** 0.784 *** 0.723 *** Integrin β1(ITGB1) 0.619 *** 0.594 *** 0.704 *** 0.679 *** Caveolin-1(CAV1) 0.715 *** 0.667 *** 0.767 *** 0.702 *** VEC CD105(ENG) 0.686 *** 0.638 *** 0.641 *** 0.570 *** CD62E(SELE) 0.635 *** 0.604 *** 0.585 *** 0.504 *** CD106(VCAM1) 0.700 *** 0.658 *** 0.762 *** 0.706 *** CD146(MCAM) 0.701 *** 0.661 *** 0.717 *** 0.661 *** None, correlation without adjustment; Purity, correlation adjusted for tumor purity; Cor value of Spearman’s correlation. *P < 0.01; **P < 0.001; ***P < 0.0001. -
[1] SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2021, 71(3): 209-249. doi: 10.3322/caac.21660 [2] SIEGEL R L, MILLER K D, FEDEWA S A, et al. Colorectal cancer statistics, 2017[J]. CA: A Cancer Journal for Clinicians, 2017, 67(3): 177-193. doi: 10.3322/caac.21395 [3] FERLAY J, STELIAROVA-FOUCHER E, LORTET-TIEULENT J, et al. Cancer incidence and mortality patterns in Europe: Estimates for 40 countries in 2012[J]. European Journal of Cancer, 2013, 49(6): 1374-1403. doi: 10.1016/j.ejca.2012.12.027 [4] ZHOU J, WANG X H, ZHAO Y X, et al. Cancer-Associated fibroblasts correlate with tumor-associated macrophages infiltration and lymphatic metastasis in triple negative breast cancer patients[J]. Journal of Cancer, 2018, 9(24): 4635-4641. doi: 10.7150/jca.28583 [5] LIU S C, QIN T T, LIU Z J, et al. Anlotinib alters tumor immune microenvironment by downregulating PD-L1 expression on vascular endothelial cells[J]. Cell Death & Disease, 2020, [6] IGLESIA M D, PARKER J S, HOADLEY K A, et al. Genomic analysis of immune cell infiltrates across 11 tumor types[J]. Journal of the National Cancer Institute, 2016, [7] DENKERT C, VON MINCKWITZ G, DARB-ESFAHANI S, et al. Tumour-Infiltrating lymphocytes and prognosis in different subtypes of breast cancer: A pooled analysis of 3771 patients treated with neoadjuvant therapy[J]. The Lancet Oncology, 2018, 19(1): 40-50. doi: 10.1016/S1470-2045(17)30904-X [8] GANESH K, STADLER Z K, CERCEK A, et al. Immunotherapy in colorectal cancer: Rationale, challenges and potential[J]. Nature Reviews Gastroenterology & Hepatology, 2019, 16(6): 361-375. [9] MURATA M, ETO Y, SHIBAI H, et al. Erythroid differentiation factor is encoded by the same mRNA as that of the inhibin beta A chain[J]. PNAS, 1988, 85(8): 2434-2438. doi: 10.1073/pnas.85.8.2434 [10] BROWN C W, HOUSTON-HAWKINS D E, WOODRUFF T K, et al. Insertion of Inhbb into the Inhba locus rescues the Inhba-null phenotype and reveals new activin functions[J]. Nature Genetics, 2000, 25(4): 453-457. doi: 10.1038/78161 [11] CHEN Y G, WANG Q, LIN S L, et al. Activin signaling and its role in regulation of cell proliferation, apoptosis, and carcinogenesis[J]. Experimental Biology and Medicine (Maywood), 2006, 231(5): 534-544. doi: 10.1177/153537020623100507 [12] SEDER C W, HARTOJO W, LIN L, et al. Upregulated INHBA expression may promote cell proliferation and is associated with poor survival in lung adenocarcinoma[J]. Neoplasia, 2009, 11(4): 388-396. doi: 10.1593/neo.81582 [13] WANG Q, WEN Y G, LI D P, et al. Upregulated INHBA expression is associated with poor survival in gastric cancer[J]. Medical Oncology, 2012, 29(1): 77-83. doi: 10.1007/s12032-010-9766-y [14] SEDER C W, HARTOJO W, LIN L, et al. INHBA overexpression promotes cell proliferation and may be epigenetically regulated in esophageal adenocarcinoma[J]. Journal of Thoracic Oncology, 2009, 4(4): 455-462. doi: 10.1097/JTO.0b013e31819c791a [15] LEE H Y, LI C C, HUANG C N, et al. INHBA overexpression indicates poor prognosis in urothelial carcinoma of urinary bladder and upper tract[J]. Journal of Surgical Oncology, 2005, 111(4): 414-422. [16] JIANG L, SI T, YU M, et al. The tumor suppressive role of inhibin betaA in diffuse large B-cell lymphoma[J]. Leukemia & Lymphoma, 2018, 59(5): 1202-1212. [17] LI H, ZHONG A J LI S, et al. The integrated pathway of TGFβ/Snail with TNFα/NFκB may facilitate the tumor-stroma interaction in the EMT process and colorectal cancer prognosis[J]. Scientific Reports, 2017, 7(1): 4915. doi: 10.1038/s41598-017-05280-6 [18] RHODES D R, KALYANA-SUNDARAM S, MAHAVISNO V, et al. Oncomine 3.0: Genes, pathways, and networks in a collection of 18, 000 cancer gene expression profiles[J]. Neoplasia, 2007, 9(2): 166-180. doi: 10.1593/neo.07112 [19] TANG Z F, LI C W, KANG B X, et al. GEPIA: A web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Research, 2017, 45(W1): W98-W102. doi: 10.1093/nar/gkx247 [20] VASAIKAR S V, STRAUB P, WANG J, et al. LinkedOmics: Analyzing multi-omics data within and across 32 cancer types[J]. Nucleic Acids Research, 2018, 46(D1): 956-963. doi: 10.1093/nar/gkx1090 [21] CHANDRASHEKAR D S, BASHEL B, BALASUBRAMANYA S A H, et al. UALCAN: A portal for facilitating tumor subgroup gene expression and survival analyses[J]. Neoplasia, 2017, 19(8): 649-658. doi: 10.1016/j.neo.2017.05.002 [22] MIZUNO H, KITADA K, NAKAI K, et al. PrognoScan: A new database for meta-analysis of the prognostic value of genes[J]. BMC Medical Genomics, 2009, [23] LI T W, FAN J Y, WANG B B, et al. TIMER: A web server for comprehensive analysis of tumor-infiltrating immune cells[J]. Cancer Research, 2017, 77: e108-e110. doi: 10.1158/0008-5472.CAN-17-0307 [24] TANAKA A, SADAHIRO S, SUZUKI T, et al. Comparisons of rigid proctoscopy, flexible colonoscopy, and digital rectal examination for determining the localization of rectal cancers[J]. Diseases of the Colon and Rectum, 2018, 61(2): 202-206. doi: 10.1097/DCR.0000000000000906 [25] HAMIDI H, IVASKA J. Every step of the way: Integrins in cancer progression and metastasis[J]. Nature Review Cancer, 2018, 18(9): 533-548. doi: 10.1038/s41568-018-0038-z [26] SEOANE J, GOMIS R R. TGF-Beta family signaling in tumor suppression and cancer progression[J]. Cold Spring Harbor Perspectives in Biology, 2017, 9(12): a022277. doi: 10.1101/cshperspect.a022277 [27] HINSHAW D C, SHEVDE L A. The tumor microenvironment innately modulates cancer progression[J]. Cancer Research, 2019, 79(18): 4557-4566. doi: 10.1158/0008-5472.CAN-18-3962 [28] WU K Y, LIN K J, LI X Y, et al. Redefining tumor-associated macrophage subpopulations and functions in the tumor microenvironment[J]. Frontiers in Immunology, 2020, 11: 1731. doi: 10.3389/fimmu.2020.01731 [29] LAN J Q, SUN L, XU F, et al. M2 macrophage-derived exosomes promote cell migration and invasion in colon cancer[J]. Cancer Research, 2019, 79(1): 146-158. doi: 10.1158/0008-5472.CAN-18-0014 [30] FREEMAN P, MIELGO A. Cancer-Associated fibroblast mediated inhibition of CD8+ cytotoxic T cell accumulation in tumours: mechanisms and therapeutics opportunities[J]. Cancers (Basel), 2020, 12(9): 2687. doi: 10.3390/cancers12092687 [31] MAO Y, KELLER E T, GARFIELD D H, et al. Stromal cells in tumor microenvironment and breast cancer[J]. Cancer Metastasis, 2013, 32(1-2): 303-315. doi: 10.1007/s10555-012-9415-3 [32] KANZAKI R, PIETRAS K. Heterogeneity of cancer- associated fibroblasts: Opportunities for precision medicine[J]. Cancer Science, 2020, 111(8): 2708-2717. doi: 10.1111/cas.14537 [33] TROPEA M M, HARPER B J A, GRANINGER G M, et al. Isolation of a circulating CD45-, CD34dim cell population and validation of their endothelial phenotype[J]. Thrombosis and Haemostasis, 2014, 112(4): 770-780. [34] HAO Y, BAKER D, DIJKE P T. TGF-Beta-Mediated Epithelial-Mesenchymal transition and cancer metastasis[J]. International Journal of Molecular Sciences, 2019, 20(11): 2767. doi: 10.3390/ijms20112767 -

 ISSN
ISSN 




 下载:
下载: