-
肿瘤微环境(tumor microenvironment, TME)是指肿瘤所生存的细胞环境,主要包括肿瘤细胞、成纤维细胞、间充质细胞、血液和淋巴管,以及各种肿瘤浸润免疫细胞与相关趋化因子和细胞因子等[1]。如此数量众多且各不相同的细胞以及细胞外基质在各种层面和尺度上相互调控和交流,共同塑造TME[2]。TME是肿瘤生长发育的温床,探究TME中发生的各种生物学过程和相互作用对于肿瘤的发展机制以及临床诊疗研究至关重要[3]。
TME中存在各种先天免疫细胞(巨噬细胞、树突状细胞、先天淋巴细胞和NK细胞等)以及适应性免疫细胞(T细胞和B细胞等)[4]。当肿瘤微环境与这些免疫细胞的功能及信号交流有关时,又可被称为肿瘤免疫微环境(tumor immune microenvironment, TIME)[5]。癌症的发展和演进受TIME中免疫细胞组分的影响并受宿主免疫系统的控制。文献[6-7]研究表明,TIME中不同免疫细胞的比例及某些免疫系统相关生物标志物可用于癌症检测、预后及治疗反应的评估。此外,TIME中还包含多种潜在的癌症治疗靶点[8-10],如CTLA-4和PD-1/PD-L1等免疫检查点阻断相关的靶点是目前肿瘤靶向治疗的热点[11-12]。同时,癌细胞与TIME中免疫细胞之间的串扰会产生促进肿瘤生长和转移的环境,对于肿瘤的发生和演进非常重要。如在TIME中,肿瘤细胞表面的配体PD-L1与T细胞表面受体的PD-1之间的互作,使得肿瘤细胞免疫抑制信号传递到T细胞内部,抑制T细胞免疫功能,从而阻止免疫系统攻击肿瘤细胞,产生免疫逃逸[13]。而针对PD1/PD-L1的免疫抑制剂已是目前肿瘤免疫治疗最热的靶点[14]。但在TIME中,除了肿瘤与免疫细胞之间的通讯交流,不同的免疫细胞间的交流同样对肿瘤的发展起重要作用。文献[15]研究表明,细胞外空间中免疫细胞和基质细胞的募集、成功激活和重编程是TIME中免疫细胞相互调控和交流的结果。如M2巨噬细胞可通过分泌TGF-β以及IL10抑制CD8+ T细胞功能,促进肿瘤细胞的免疫逃逸[16]。嗜酸性粒细胞可通过分泌CXCL9、CXCL10及CCL5募集和激活T细胞,通过分泌IL-6、IL-12和CXCL10募集NK细胞,并诱导M1极化[17]。NK细胞可通过自分泌和旁分泌PGE和TGF-β等细胞因子的方式调节自身的肿瘤免疫杀伤功能[18]。因此,进一步探究TIME中免疫细胞间的信号通讯网络对于肿瘤演进机制以及开发新的肿瘤免疫治疗手段具有重要价值。
最近,快速发展的单细胞RNA测序(single cell RNA sequencing, scRNA-seq)技术可在单细胞水平上精确表征肿瘤组成,提供对肿瘤的异质性和基因表达的高分辨率景观,是剖析TIME的有力工具[19]。目前已有大量研究利用scRNA-seq技术探究各种癌症的TIME中的细胞景观,为解析TIME与肿瘤发生发展机制提供了重要线索[20-24]。但此类研究主要关注肿瘤细胞与免疫细胞间的信号通讯,而TIME中不同免疫细胞间的信号通讯网络探讨较少。因此,为深入挖掘TIME中免疫细胞间的通讯网络,本文收集多套TIME的单细胞测序数据,并结合最新的细胞间通讯预测算法和工具[25-26],尝试解析TIME中不同免疫细胞间的信号通讯网络,以揭示肿瘤在免疫微环境中的协调和发展机制,并进一步为肿瘤的临床诊断和治疗提供新的线索。
-
本文共收集8套肿瘤样本及3套正常样本的CD45+细胞的测序数据,数据集相关信息如表1所示。肿瘤数据包括两套肝细胞肝癌(hepatocellular carcinoma, HCC)、两套头颈癌(head and neck squamous cell carcinoma, HNSCC)、一套黑色素瘤 (melanoma)、一套乳腺癌 (breast carcinoma, BRCA)、一套透明细胞肾癌(clear cell renal carcinoma, ccRCC)以及一套透明细胞肾癌类器官(clear cell renal carcinoma organoid, ccRCCO)的数据。正常样本数据包括两套肝脏组织以及一套乳腺组织数据。以上数据涵盖了10X以及smart-seq2两种测序技术平台。
-
首先从Gene Expression Omnibus (GEO)数据库下载11套数据集的count数据及meta数据,使用Seurat3.0默认参数对所有数据初步过滤,要求每个细胞内检测到的线粒体基因比例小于10%,利用log2[TPM/10+1]对基因表达值进行标准化。
-
使用Garnett (Version: 0.1.20)算法对细胞的身份进行识别[27]。研究7种免疫细胞,包括树突状细胞(dendritic cell, DC)、自然杀伤细胞(natural killer cells, NK)、单核/巨噬细胞(monocytes/macrophages,mono/macro)、B细胞(B cells)、T细胞 (T cells)及其两个亚型:CD4 T细胞(CD4 T cells)与CD8 T细胞(CD8 T cells)。不同免疫细胞的细胞标志物(marker)信息如表2所示。部分Garnett算法未成功注释的unkown细胞在后续的分析中被过滤掉。
表 1 CD45+细胞scRNA-seq数据集
样本 来源 测序平台 组织类型 细胞数目 肿瘤 GSE111360 10X ccRCC 17 237 GSE111360 10X ccRCCO 33 578 GSE114725 10X Breast Tumor 14 291 GSE139324 10X HNSCC(HPV+) 18 864 GSE139324 10X HNSCC(HPV-) 25 667 GSE140228 10X HCC 12 367 GSE140228 SMART-Seq2 HCC 1 482 GSE72056 SMART-Seq2 melanoma 1 827 正常 GSE114725 10X 乳腺 3 095 GSE140228 10X 肝脏 9 869 GSE140228 SMART-Seq2 肝脏 1 180 表 2 7种免疫细胞的标志物
细胞 标志物 NK cells NCAM1, FCGR3A Monocytes CD14, FCGR1A, CD68, S100A12 B cells CD19, MS4A1, CD79A T cells CD3D, CD3E, CD3G CD4 T cells CD4, FOXP3, IL2RA, IL7R CD8 T cells CD8A, CD8B Dendritic cells IL3RA, CD1C, BATF3, THBD, CD209 -
使用CellCall算法推测TIME中不同免疫细胞间通讯关系[25]。CellCall算法通过整合配体−受体(ligand-receptor, L-R)互作的表达和L-R互作下游转录因子(transcription factor, TF)的激活程度来推断细胞间通讯关系。同时,CellCall还嵌入了一个通路激活分析算法来识别不同细胞间通讯所涉及的关键信号转导通路。在使用CellCall算法推测细胞间通讯之前,本文排除了在特定细胞类型的少于10%的细胞中表达的基因。
-
为了筛选在肿瘤与正常样本中高变异的差异通讯关系,本文首先计算每条通讯关系在肿瘤与正常样本间的差异倍数(fold change, FC);然后使用R包“statmod”拟合不同通讯关系的FC值方差的广义线性模型。广义线性模型是线性模型在研究响应值的非正态分布以及非线性模型的线性转化时的一种发展,用以筛选在不同细胞间高变异的信号通讯关系。其原理为利用广义的线性模型拟合所有通讯关系在不同的FC均值情况下的期望方差,若某细胞间通讯关系的实际方差值显著高于期望方差,即高于期望方差的1.5倍,则该通讯关系为高变异的信号通讯关系。
-
泛癌表达谱数据来自于TCGA数据库,包括33种癌症的表达谱数据以及临床资料信息。使用R包“survival”中的Kaplan-Meier、log-rank检验和单变量Cox回归评估TF表达与生存时间之间的关系。使用Metascape对差异基因进行功能富集分析[28]。使用Wilcoxon秩和检验评估正常与肿瘤样本中细胞间通讯强度的差异(P<0.05)。
-
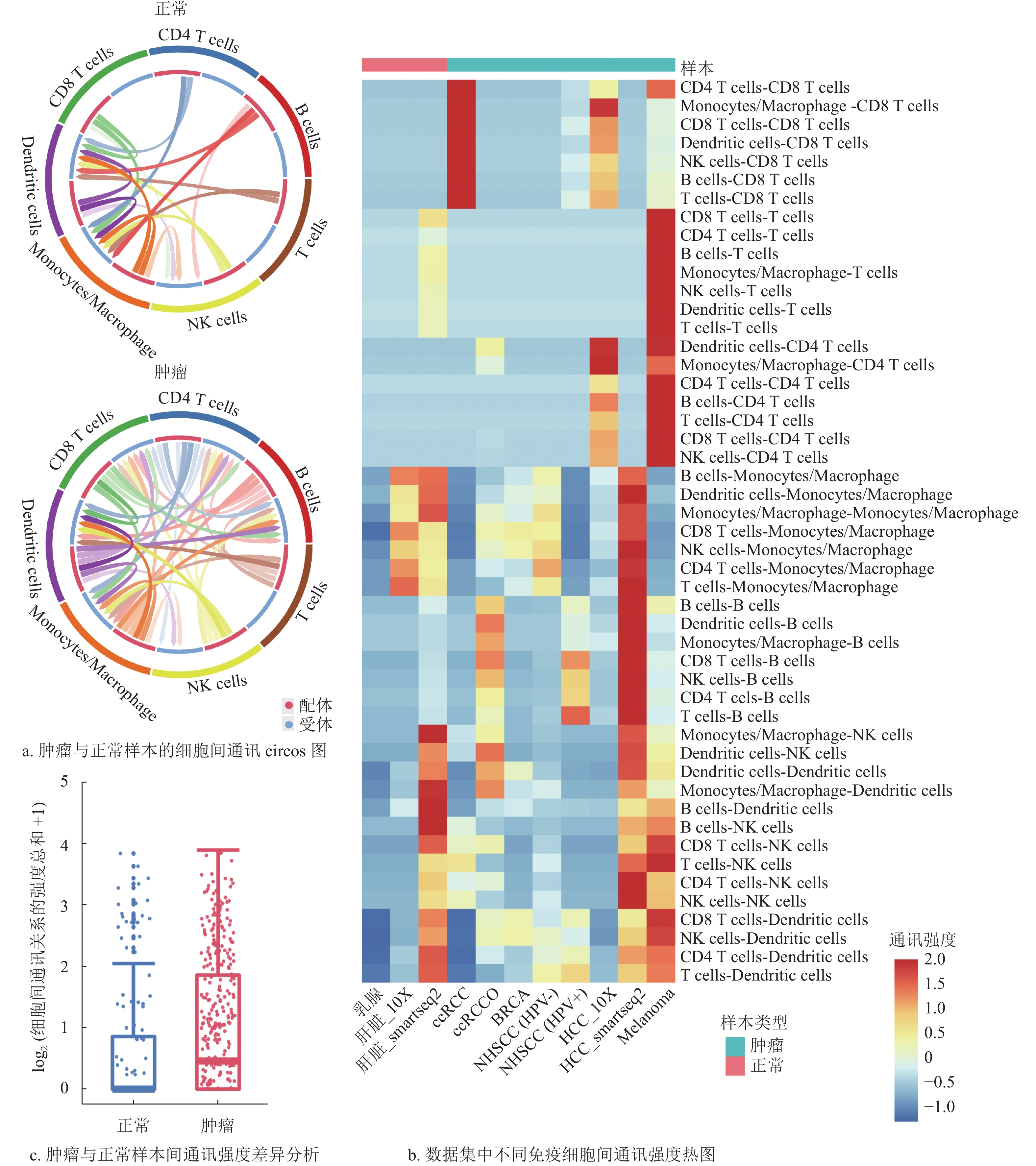
为探究肿瘤与正常样本TIME中免疫细胞间的整体景观以及差异,本文使用CellCall算法分别对11套TIME样本进行细胞间通讯预测。结果如图1a和1b所示,7种免疫细胞在正常样本和肿瘤样本中均存在大量的细胞间通讯关系。正常样本与肿瘤样本的整体细胞间通讯强度的比较分析如图1c所示,发现肿瘤样本中不同免疫细胞间存在更强的细胞间通讯(P=2.6×10−5)。可见,在免疫微环境中不同免疫细胞之间存在大量高度复杂的交流和通讯网络,但相对来说,在肿瘤样本中免疫细胞间的交流更加频繁与密切。因此,进一步探究肿瘤中特异以及缺失的细胞间通讯关系,对于理解肿瘤在机体内的发生发展过程及临床诊断治疗研究具有重要价值。
-
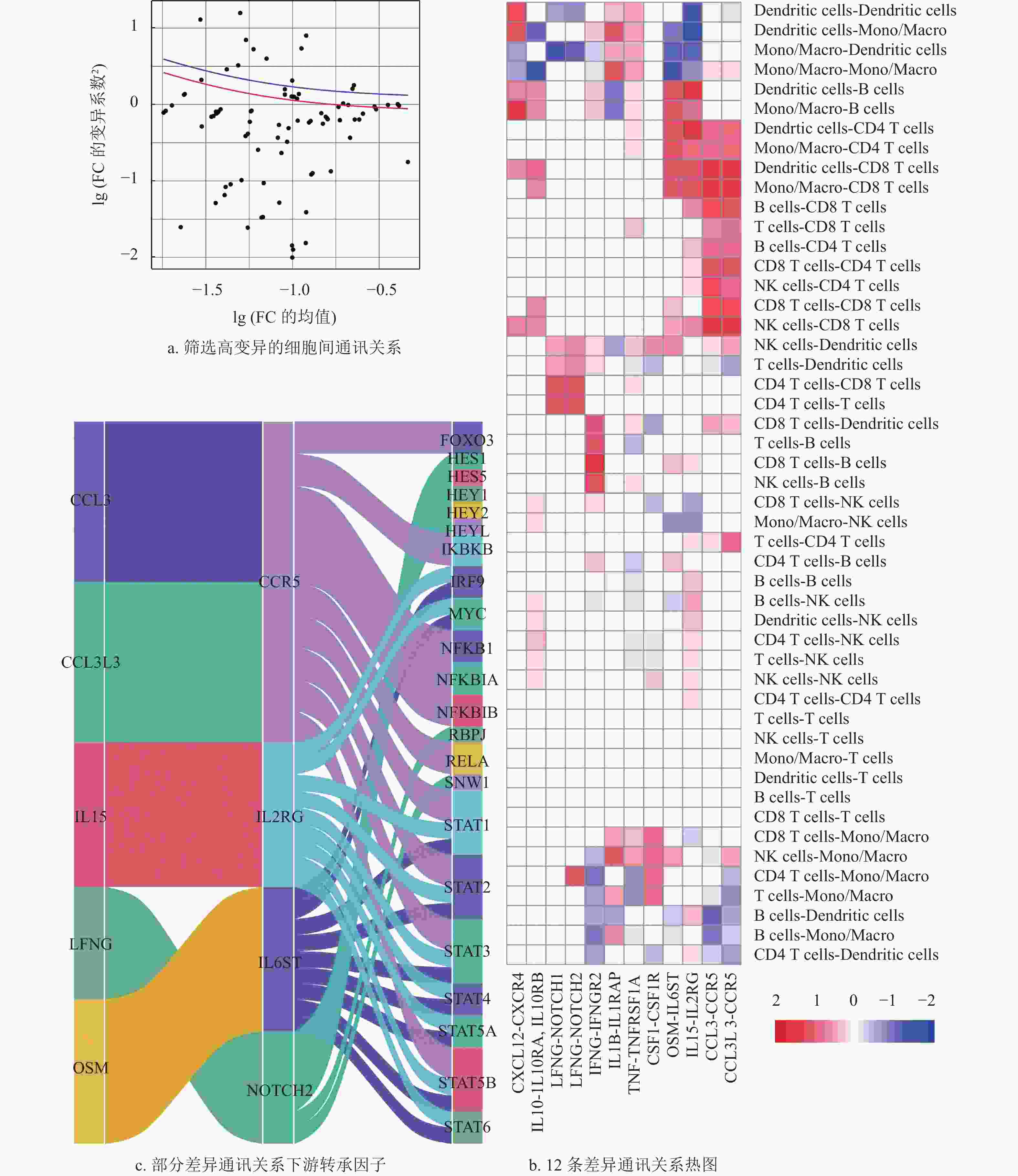
为进一步筛选肿瘤与正常样本间不同免疫细胞之间显著差异的细胞间通讯关系,本文通过对每条细胞间通讯关系在肿瘤与正常样本中的FC值去拟合广义线性模型曲线,筛选高变异的细胞间通讯关系,如图2a所示。得到了12条在肿瘤和正常样本间显著存在差异的细胞间通讯关系,其结果如图2b所示,主要包括各种趋化因子−趋化因子受体信号(CCL3/CCL3L3-CCR5、CXCL12-CXCR4)、白介素−白介素受体信号(IL15-IL2RG、IL1B-IL1RAP、IL10-IL10RA、IL10RB) 以及Notch 通路信号(LFNG-NOTCH1/NOTCH2)。已有大量文献证明这些细胞间通讯信号关系与肿瘤免疫显著相关[29]。如CXCL12-CXCR4可介导浆DC细胞的肿瘤浸润和TREG细胞归巢至骨髓微环境,并参与肿瘤细胞的增殖、转移及肿瘤血管生成[30-33]。LFNG- NOTCH信号可介导T细胞发育失调,导致肿瘤微环境中T/B细胞比例改变,影响肿瘤免疫过程[34-35]。
进一步探究部分差异通讯关系下游存在的转录因子,如图2c所示,发现这些通讯关系下游的转录因子主要包括NFKB家族(NFKB1、NFKBIA与NFKBIB) 以及STAT 家族(STAT1、STAT2、STAT3、STAT4、STAT5A、STAT5B与STAT6)及其他肿瘤相关转录因子。这些转录因子在免疫细胞的分化与肿瘤发展密切相关[36-37]。
-
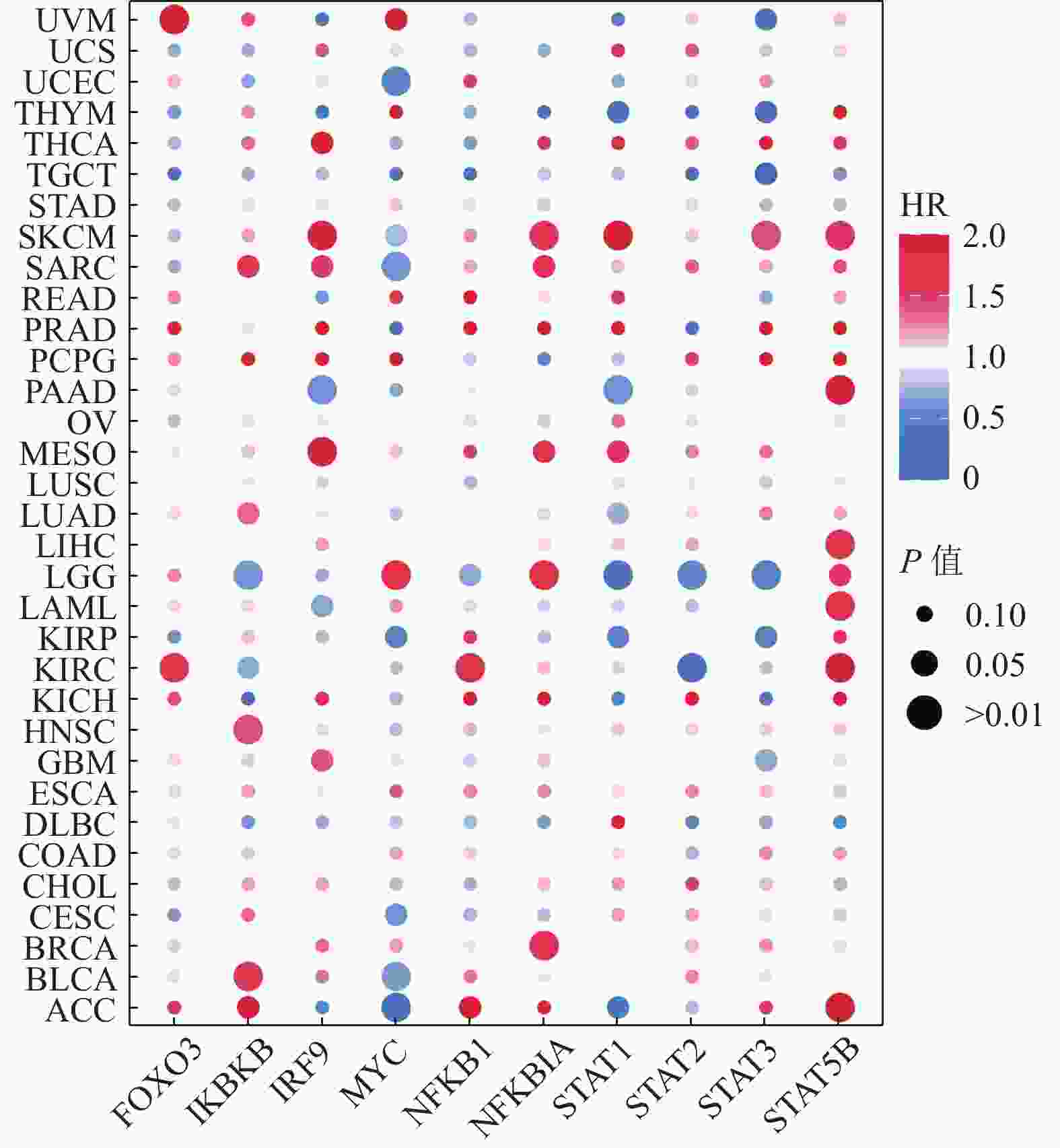
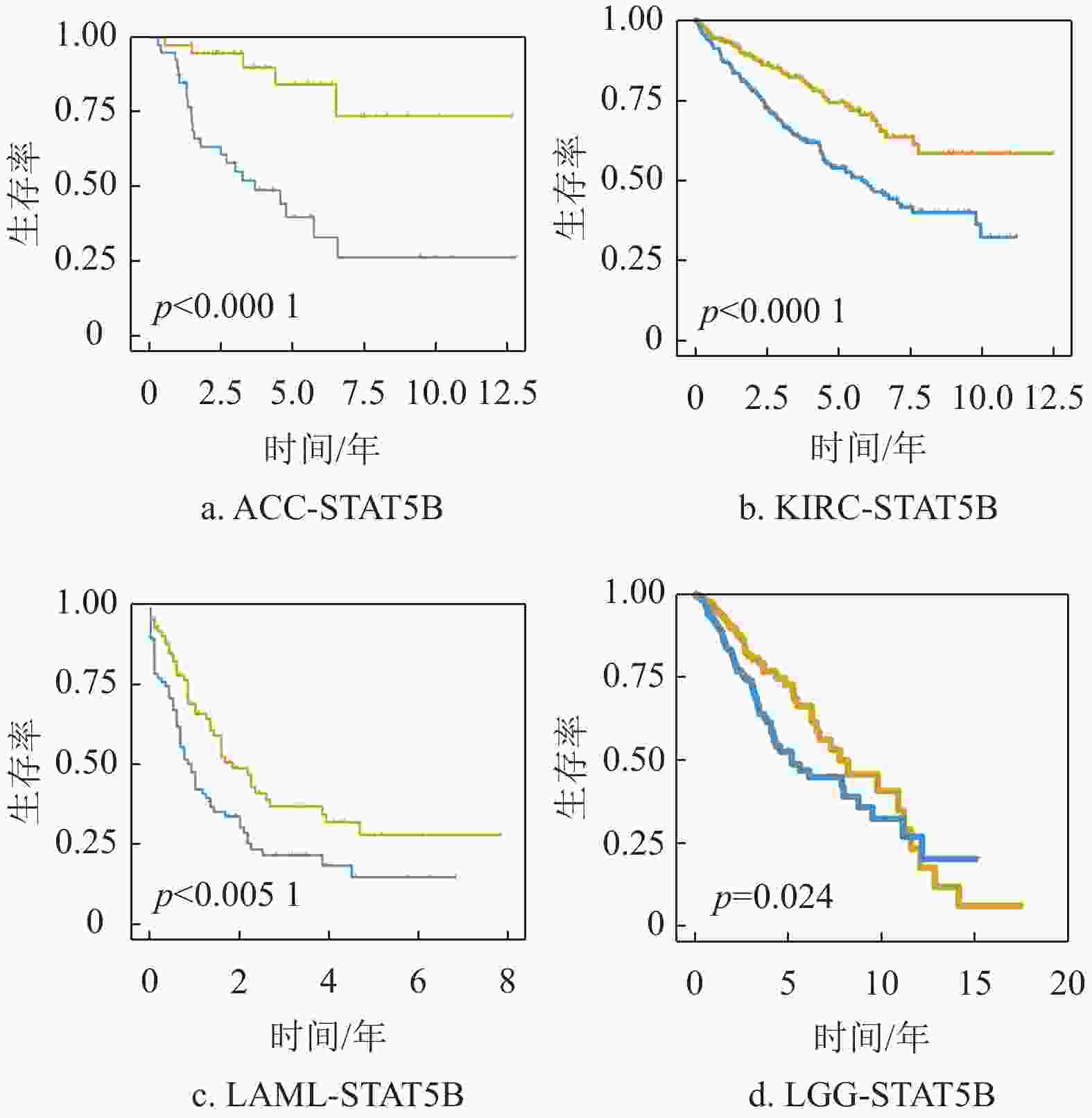
为进一步明确相关细胞间通讯信号关系与肿瘤预后的关系,本文分析了这些差异细胞间通讯下游的10个关键转录因子与不同肿瘤的预后相关性。通过对TCGA数据库中33种肿瘤进行预后分析发现,这些转录因子在部分肿瘤中呈现显著的预后相关性,如图3所示。一些转录因子在不同的肿瘤中存在相反的预后情况,如IKBKB的表达在HNSC、BLCA及ACC等肿瘤中是显著的风险因素,在LGG中却是显著的保护因素。但是,一些转录因子在大多数肿瘤中的预后却趋向于一致,如STAT5B在ACC、KIRC、LAML及PAAD等肿瘤中均是显著的风险因素,如图4所示。以上结果提示,这些差异的细胞间通讯关系以及下游转录因子与TIME的发展相关,值得深入研究以揭示肿瘤在免疫微环境中的协调和发展机制,并进一步为肿瘤的临床诊断和治疗提供线索。
-
在TIME中,除了肿瘤细胞与免疫细胞之间的交流之外,不同免疫细胞间的信息交流对与肿瘤的发生发展也具有重要作用[16]。本文通过收集多种肿瘤组织CD45+细胞的单细胞测序数据,推测不同免疫细胞间存在的细胞间通讯关系,并与正常样本进行比较。结果发现,免疫细胞间通讯在肿瘤样本中更加频繁,且该通讯关系多涉及各种趋化因子−趋化因子受体信号、白介素−白介素受体信号以及Notch信号通路。此外,转录因子与肿瘤生存期的生存分析发现部分转录因子与多种肿瘤的预后显著相关。因此,本文构建的肿瘤免疫微环境中不同免疫细胞间的全局通讯景观,有助于理解各免疫细胞在肿瘤发生发展中的通讯关系及表明相关转录因子在肿瘤预后的辅助提示作用。
本文仍然存在不足之处。首先,肿瘤与正常样本来源于多种不同组织,为非配对样本。同时,由于细胞数量的限制,本文只注释了7种重要的免疫细胞,还有更多的免疫细胞及亚型未得到有效识别。此外,筛选出的差异通讯信号关系,未进行进一步的实验验证。在后续研究中,将收集更全面和丰富的TIME及配对样本的单细胞测序数据,尝试识别更多种免疫细胞及亚型,探究其相互间通讯景观,深入研究其机制机理。
Deciphering the Landscape of Intercellular Communication Among Immune Cells in Tumor Immune Microenvironment
-
摘要: 肿瘤免疫微环境(TIME)内细胞间通讯为肿瘤发生发展提供了重要的微环境,但目前单细胞相关研究多关注免疫细胞与肿瘤之间的通讯,对不同免疫细胞间的信号交流研究较少。由此收集11套CD45+细胞的单细胞测序数据,推测出TIME中不同免疫细胞间的信号通讯网络。结果发现,肿瘤样本中免疫细胞间的交流更加频繁与密切,进一步分析得到12条与肿瘤相关的差异信号通讯关系,主要包括各种趋化因子−趋化因子受体信号、白介素−白介素受体信号以及Notch通路信号。这些通讯信号关系及其下游转录因子与肿瘤发生发展密切相关。此外,发现部分关键转录因子与各种肿瘤的预后显著相关。最后,通过构建TIME中不同免疫细胞间存在的细胞间通讯全局景观,挖掘出肿瘤中特异及缺失的免疫细胞间通讯关系,为理解TIME中免疫系统调控及肿瘤发展机制提供了线索,也为肿瘤的临床诊疗研究提供了新的设计思路。Abstract: The intercellular communication in tumor immune microenvironment (TIME) provides an important niche for tumorigenesis and tumor progression. Most of single cell level studies focus on the crosstalk between tumor cells and immune cells, lacking of research on the intercellular communication among immune cells. Hence, in this study, we collected 11 single cell RNA sequencing (scRNA-seq) data of CD45+ cells, and deciphered the intercellular communication network among different immune cells in TIME. The results demonstrated that there were significantly more intercellular communications in tumor samples, and 12 differential intercellular communications were detected between tumor and normal samples, including some chemokine -chemokine receptor signaling, interleukin-interleukin-receptor signaling and notch signaling. Most of these signaling and downstream transcription factors (TFs) have been implicated in tumor progression. Survival analysis of some TFs based on the cancer genome altas (TCGA) pan-cancer data indicated that these identified TFs were significantly associated with the overall survival in patients with different cancers. In summary, this study provides a comprehensive view of the intercellular communication landscape among immune cells in TIME, and identifies some specific intercellular communications involved in tumor immunity. We believe that our study provides valuable clues for understanding the mechanisms of tumor progression in TIME and provides possible diagnostic strategies for the tumor diagnosis and treatment.
-
表 1 CD45+细胞scRNA-seq数据集
样本 来源 测序平台 组织类型 细胞数目 肿瘤 GSE111360 10X ccRCC 17 237 GSE111360 10X ccRCCO 33 578 GSE114725 10X Breast Tumor 14 291 GSE139324 10X HNSCC(HPV+) 18 864 GSE139324 10X HNSCC(HPV-) 25 667 GSE140228 10X HCC 12 367 GSE140228 SMART-Seq2 HCC 1 482 GSE72056 SMART-Seq2 melanoma 1 827 正常 GSE114725 10X 乳腺 3 095 GSE140228 10X 肝脏 9 869 GSE140228 SMART-Seq2 肝脏 1 180 表 2 7种免疫细胞的标志物
细胞 标志物 NK cells NCAM1, FCGR3A Monocytes CD14, FCGR1A, CD68, S100A12 B cells CD19, MS4A1, CD79A T cells CD3D, CD3E, CD3G CD4 T cells CD4, FOXP3, IL2RA, IL7R CD8 T cells CD8A, CD8B Dendritic cells IL3RA, CD1C, BATF3, THBD, CD209 -
[1] FRIDMAN W H, PAGÈS F, SAUTÈS-FRIDMAN C, et al. The immune contexture in human tumours: Impact on clinical outcome[J]. Nat Rev Cancer, 2012, 12(4): 298-306. doi: 10.1038/nrc3245 [2] ANGELL H, GALON J. From the immune contexture to the immunoscore: The role of prognostic and predictive immune markers in cancer[J]. Curr Opin Immunol, 2013, 25(2): 261-267. doi: 10.1016/j.coi.2013.03.004 [3] ARNETH B. Tumor microenvironment[J]. Medicina, 2020, 56(1): 15. [4] GAJEWSKI T F, SCHREIBER H, FU Y X. Innate and adaptive immune cells in the tumor microenvironment[J]. Nat Immunol, 2013, 14(10): 1014-1022. doi: 10.1038/ni.2703 [5] BINNEWIES M, ROBERTS E W, KERSTEN K, et al. Understanding the tumor immune microenvironment (time) for effective therapy[J]. Nat Med, 2018, 24(5): 541-550. doi: 10.1038/s41591-018-0014-x [6] WATNICK R S. The role of the tumor microenvironment in regulating angiogenesis[J]. Cold Spring Harb Perspect Med, 2012, 2(12): a006676. [7] POTTIER C, WHEATHERSPOON A, RONCARATI P, et al. The importance of the tumor microenvironment in the therapeutic management of cancer[J]. Expert Review of Anticancer Therapy, 2015, 15(8): 943-954. doi: 10.1586/14737140.2015.1059279 [8] FRIDMAN W H, MILLER I, SAUTÈS-FRIDMAN C, et al. Therapeutic targeting of the colorectal tumor stroma[J]. Gastroenterology, 2020, 158(2): 303-321. doi: 10.1053/j.gastro.2019.09.045 [9] PETITPREZ F, VANO Y A, BECHT E, et al. Transcriptomic analysis of the tumor microenvironment to guide prognosis and immunotherapies[J]. Cancer Immunol Immunother, 2018, 67(6): 981-988. doi: 10.1007/s00262-017-2058-z [10] FRIDMAN W H, ZITVOGEL L, SAUTÈS-FRIDMAN C, et al. The immune contexture in cancer prognosis and treatment[J]. Nat Rev Clin Oncol, 2017, 14(12): 717-734. doi: 10.1038/nrclinonc.2017.101 [11] PARDOLL D M. The blockade of immune checkpoints in cancer immunotherapy[J]. Nat Rev Cancer, 2012, 12(4): 252-264. doi: 10.1038/nrc3239 [12] TOPALIAN S L, DRAKE C G, PARDOLL D M. Immune checkpoint blockade: A common denominator approach to cancer therapy[J]. Cancer Cell, 2015, 27(4): 450-461. doi: 10.1016/j.ccell.2015.03.001 [13] LEI Q Y, WANG D, SUN K, et al. Resistance mechanisms of anti-pd1/pdl1 therapy in solid tumors[J]. Front Cell Dev Biol, 2020, 8: 672. doi: 10.3389/fcell.2020.00672 [14] STENEHJEM D D, TRAN D, NKRUMAH M A, et al. Pd1/pdl1 inhibitors for the treatment of advanced urothelial bladder cancer[J]. Onco Targets Ther, 2018, 11: 5973-5989. doi: 10.2147/OTT.S135157 [15] KORNEEV K V, ATRETKHANY K N, DRUTSKAYA M S, et al. Tlr-Signaling and proinflammatory cytokines as drivers of tumorigenesis[J]. Cytokine, 2017, 89: 127-135. doi: 10.1016/j.cyto.2016.01.021 [16] ANFRAY C, UMMARINO A, ANDÓN F T, et al. Current strategies to target tumor-associated-macrophages to improve anti-tumor immune responses[J]. Cells, 2020, 9(1): 46. [17] SIMON S C S, UTIKAL J, UMANSKY V. Opposing roles of eosinophils in cancer[J]. Cancer Immunology Immunotherapy, 2019, 68(5): 823-833. doi: 10.1007/s00262-018-2255-4 [18] DEVISSER K E, EICHTEN A, COUSSENS L M. Paradoxical roles of the immune system during cancer development[J]. Nat Rev Cancer, 2006, 6(1): 24-37. doi: 10.1038/nrc1782 [19] KAMINSKA B, OCHOCKA N, SEGIT P. Single-cell omics in dissecting immune microenvironment of malignant gliomas-challenges and perspectives[J]. Cells, 2021, 10(9): 2264. [20] ZHANG Q M, HE Y, LUO N, et al. Landscape and dynamics of single immune cells in hepatocellular carcinoma[J]. Cell, 2019, 179(4): 829-845. doi: 10.1016/j.cell.2019.10.003 [21] AZIZI E, CARR A J, PLITAS G, et al. Single-cell map of diverse immune phenotypes in the breast tumor microenvironment[J]. Cell, 2018, 174(5): 1293-1308. doi: 10.1016/j.cell.2018.05.060 [22] TIROSH I, IZAR B, PRAKADAN S M, et al. Dissecting the multicellular ecosystem of metastatic melanoma by single-cell rna-seq[J]. Science, 2016, 352(6282): 189-196. doi: 10.1126/science.aad0501 [23] LAVIN Y, KOBAYASHI S, LEADER A, et al. Innate immune landscape in early lung adenocarcinoma by paired single-cell analyses[J]. Cell, 2017, 169(4): 750-765. doi: 10.1016/j.cell.2017.04.014 [24] CILLO A R, KÜRTEN C H L, TABIB T, et al. Immune landscape of viral- and carcinogen-driven head and neck cancer[J]. Immunity, 2020, 52(1): 183-199. doi: 10.1016/j.immuni.2019.11.014 [25] ZHANG Y, LIU T Y, HU X S, et al. Cellcall: Integrating paired ligand-receptor and transcription factor activities for cell-cell communication[J]. Nucleic Acids Research, 2021, 49(15): 8520-8534. doi: 10.1093/nar/gkab638 [26] ZHANG Y, LIU T Y, WANG J, et al. Cellinker: A platform of ligand-receptor interactions for intercellular communication analysis[J]. Bioinformatics, 2021, 37(14): 2025-2032. doi: 10.1093/bioinformatics/btab036 [27] PLINER H A, SHENDURE J, TRAPNELL C. Supervised classification enables rapid annotation of cell atlases[J]. Nat Methods, 2019, 16(10): 983-986. doi: 10.1038/s41592-019-0535-3 [28] ZHOU Y Y, ZHOU B, PACHE L, et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets[J]. Nature Communications, 2019, 10(1): 1523. doi: 10.1038/s41467-019-09234-6 [29] SHI Y, RIESE D J, SHEN J Z. The role of the cxcl12/cxcr4/cxcr7 chemokine axis in cancer[J]. Frontiers in Pharmacology, 2020, 11(1969): 574667. [30] ZOU L H, BARNETT B, SAFAH H, et al. Bone marrow is a reservoir for cd4+cd25+ regulatory t cells that traffic through cxcl12/cxcr4 signals[J]. Cancer Res, 2004, 64(22): 8451-8455. doi: 10.1158/0008-5472.CAN-04-1987 [31] ZHAO E D, WANG L, DAI J L, et al. Regulatory t cells in the bone marrow microenvironment in patients with prostate cancer[J]. Oncoimmunology, 2012, 1(2): 152-161. doi: 10.4161/onci.1.2.18480 [32] KRYCZEK I, LANGE A, MOTTRAM P, et al. Cxcl12 and vascular endothelial growth factor synergistically induce neoangiogenesis in human ovarian cancers[J]. Cancer Res, 2005, 65(2): 465-472. [33] SCOTTON C J, WILSON J L, MILLIKEN D, et al. Epithelial cancer cell migration: A role for chemokine receptors?[J]. Cancer Res, 2001, 61(13): 4961-4965. [34] YUAN J S, TAN J B, VISAN I, et al. Lunatic fringe prolongs delta/notch-induced self-renewal of committed αβ t-cell progenitors[J]. Blood, 2011, 117(4): 1184-1195. doi: 10.1182/blood-2010-07-296616 [35] SONG Y, KUMAR V, WEI H X, et al. Lunatic, manic, and radical fringe each promote t and b cell development[J]. J Immunol, 2016, 196(1): 232-243. doi: 10.4049/jimmunol.1402421 [36] PALICELLI A, CROCI S, BISAGNI A, et al. What do we have to know about pd-l1 expression in prostate cancer? A systematic literature review. Part 3: Pd-l1, intracellular signaling pathways and tumor microenvironment[J]. International Journal of Molecular Sciences, 2021, 22(22): 12330. [37] YU H, PARDOLL D, JOVE R. Stats in cancer inflammation and immunity: A leading role for stat3[J]. Nature Reviews Cancer, 2009, 9(11): 798-809. doi: 10.1038/nrc2734 -

 ISSN
ISSN 




 下载:
下载: