-
医学影像检查可以有效地辅助医生进行临床诊断。与传统的医学诊断方法相比,医学影像可以无损伤地检查人体的内部结构。由文献[1]可知,医学影像分割是医学影像分析领域中的重要步骤,医学影像分割可以辅助医生定位感兴趣目标的位置,如器官和病变组织。医学影像的获取方式(如计算机断层扫描成像CT,磁共振成像MRI和超声波成像)的不同以及几何维度的不同会导致身体内部器官和病变位置在显示上的形变[2],这增加了医学影像分割的困难程度。一般来说,不同的放射学家对同一医学影像的分割标注会产生差异,甚至同一放射学家对同一医学影像的多次分割标注也会产生差异,导致了分割结果的不可复制性。全自动分割算法可以减少分割医学影像所需要的时间,并产生更加一致的分割掩膜[3-4]。
卷积网络模型AlexNet[5]在ImageNet[6]图像分类竞赛中夺得了冠军,从而引发了深度卷积神经网络的研究热潮。与使用人工设计特征的传统机器学习算法相比,深度卷积神经网络可以通过一定量的标记数据自动学习到更加优秀的多层次特征。深度卷积神经网络在解决各种计算机视觉任务上取得了极大的成功,如图片分类任务[5,7-8]、目标检测任务[9-10]和语义分割任务[11-12]等。值得关注的是,深度卷积神经网络在医学影像分析上同样展示了其优越的性能[13-14]。深度学习的成功主要基于两点:大量的带有标签的训练数据和网络的固有能力。为了提升网络在不同数据集上的分类性能,很多研究学者注重于构建新的网络架构,如AlexNet[5]、VGGNet[8]、Efficientnet [15]等。同时,为了衡量神经网络的性能,部分学者也尝试构建了几个大型数据集,如ImageNet[6]和Open Image[16],这两个数据集都包含数百万张图片,且每张图片都包含有固定的标签。然而,具有标签的医学影像数据集是很难获取的,对于放射科医生而言,手动标注医学影像既浪费时间又耗费精力。
和医学影像分割标签的获取难度相比,医学影像检测标签的人工标注难度就要低很多。医学影像分割任务是一种像素级的分类任务,所需要的标签也是像素级的。与医学影像分割任务不一样的是,医学影像检测任务不需要对医学影像进行逐像素的分类,只需要确定感兴趣对象的边界,一般地,感兴趣目标的位置可以通过左上角和右下角两个像素点进行确定。简而言之,和人工像素级标注医学影像相比,检测边界盒的手动标注大大减少了放射科医师的工作量。
鉴于具有检测标签的医学影像数据更加容易获取,而医学影像分割任务是医学影像分析领域中的关键步骤,本文提出了基于目标检测的医学影像分割算法,该算法降低了分割算法对像素级标签的依赖程度,是一种弱监督的语义分割算法。该算法由4个步骤组成:1)医学影像预处理,包括但不限于随机翻转、随机旋转、随机裁剪、图像正则化;2)将预处理好的医学影像输入到目标检测网络中,得到感兴趣目标的图像块;3)以图像块的关键区域作为参考对象,该图像块作为搜索对象,由孪生网络计算图像块中所有像素点被分类为关键像素点的概率;4)根据概率图和预先设定好的概率阈值得到分割结果。
-
对医学影像中的一些关键目标进行分割可以辅助临床医生做出准确的诊断。早期的医学影像分割算法通常依赖于传统的机器学习技术和手动设计的特征,这些方法取得了不错的效果。但由于特征的设计过于繁琐复杂,图像分割仍然是一项挑战性的任务。深度网络成功从图像中提取到多层次的特征表示,从而成为医学影像分析领域的热门课题之一。
-
目标检测任务旨在找出图像或视频中的感兴趣对象,并同时执行分类任务和定位任务。基于卷积神经网络的目标检测算法可以分为一阶段的目标检测算法和二阶段的目标检测算法。二阶段的目标检测算法起始于RCNN[17],RCNN算法首先使用selective search[18]选择一组目标检测框,将这些图像输入到卷积神经网络提取特征,然后将这些特征输入到分类器中判断检测框中是否存在待检测的目标,并进一步对这些目标进行分类。基于这个基础,各种改进版随之提出,如Faster RCNN[19]、Mask RCNN[20]、Grid RCNN[21]、Libra RCNN[22]等。作为一阶段目标检测算法的代表,YOLO[23]和SSD[24]的检测速度有了很大的提高。YOLO的思想就是将图像划分为多个网格,然后预测每个网格里的待检测目标的边界框。SSD有多个不同的检测分支,可以同时检测多个尺度的目标,对小目标的检测效果很好。综合考虑检测速度和检测精度,本文使用SSD目标检测网络作为基础网络。
孪生网络最早由文献[25]提出,用于验证银行支票上的签名和预留签名是否一致。文献[26]对孪生网络做了改进,将卷积神经网络引入孪生网络。文献[27]探索了一种学习孪生神经网络的方法,该方法采用了一种独特的结构对输入之间的相似性进行自然排序,在one-shot图片分类任务上取得了优秀的性能。文献[28]提出了一种基于注意力机制的孪生网络,同时学习视频的时空表示及其相似性度量。文献[29]将孪生网络应用到目标跟踪领域,开启了目标跟踪领域的一个新方向[30-31]。医学影像中感兴趣目标的像素点之间的特征具有很强的相似性,而孪生网络的固有特性决定了其强大的匹配能力,所以本文使用孪生网络解决医学影像中感兴趣对象图像块的分割问题(也称为像素分类问题)。
-
随着深度卷积神经网络的发展,卷积模型在医学影像分割任务上获得了极大的成功,有效地提升了医学影像分割的效率。
文献[32]使用多个深度模型来分割神经单元,输入图像中的每个像素的标签从以该像素为中心的方形窗口预测得到。文献[33]设计了一个新的体素分类系统,该系统集成了3个二维卷积神经网络,分别与三维图像的3个平面一一对应,并将其应用于膝关节胫骨和软骨的分割。文献[34]设计了一个多分支的卷积神经网络模型对肺结节进行分割。文献[35]设计了一个级联的深度域适应模型在CT图像中对前列腺进行分割。深度卷积神经网络同样被应用在器官及病变组织的分割任务上。文献[36]提出了一种可以分段训练的网络模型Crossbar-Net。Crossbar-Net网络由水平子模型和垂直子模型构成,使得该网络能够同时从垂直和水平的方向捕获肾肿瘤的全局和局部的外观信息。两个模型相互补充,实现了自我完善直至收敛,该模型被应用到了基于CT图像的肾脏肿瘤分割任务、基于磁共振图像的心脏分割任务和基于X射线的乳腺肿块分割任务。文献[37]提出了一个三阶段的自我引导网络,以完成肾脏肿瘤的分割任务。该网络使用低分辨率网络从下采样的CT图像中粗略的估计肾脏肿瘤的位置,使用全分辨率网络和肿瘤细化网络从全分辨率CT图像中提取肾脏和肿瘤的精确边界。文献[38]训练了一个细胞核分割模型,以实现对肾透明细胞癌的病理图像进行逐像素的分割。
为了降低医学影像分割算法对像素级标签的依赖程度,交互式分割算法同样被应用于解决医学影像分割任务,从而获取到更加精确的分割结果,如引入地标点[39]、边界框[40]和少量关键像素点[41],这些方法都可以指导医学影像分割任务并完善分割结果。
但监督深度分割网络的基础之一是具有像素级标签的大型医学影像数据集,而这些分割标签需要资深的放射科医师手工标注或检查,交互式分割算法也需要一些像素级的注释或人工指导,所以,本文提出了基于目标检测的医学影像分割算法,以减少医学影像分割对于像素级标签的依赖,且无需人工参与指导。
-
本文结合目标检测网络和孪生网络提出了一种新的医学影像分割算法,使用很少的标注信息实现对医学影像数据中感兴趣目标的分割。
-
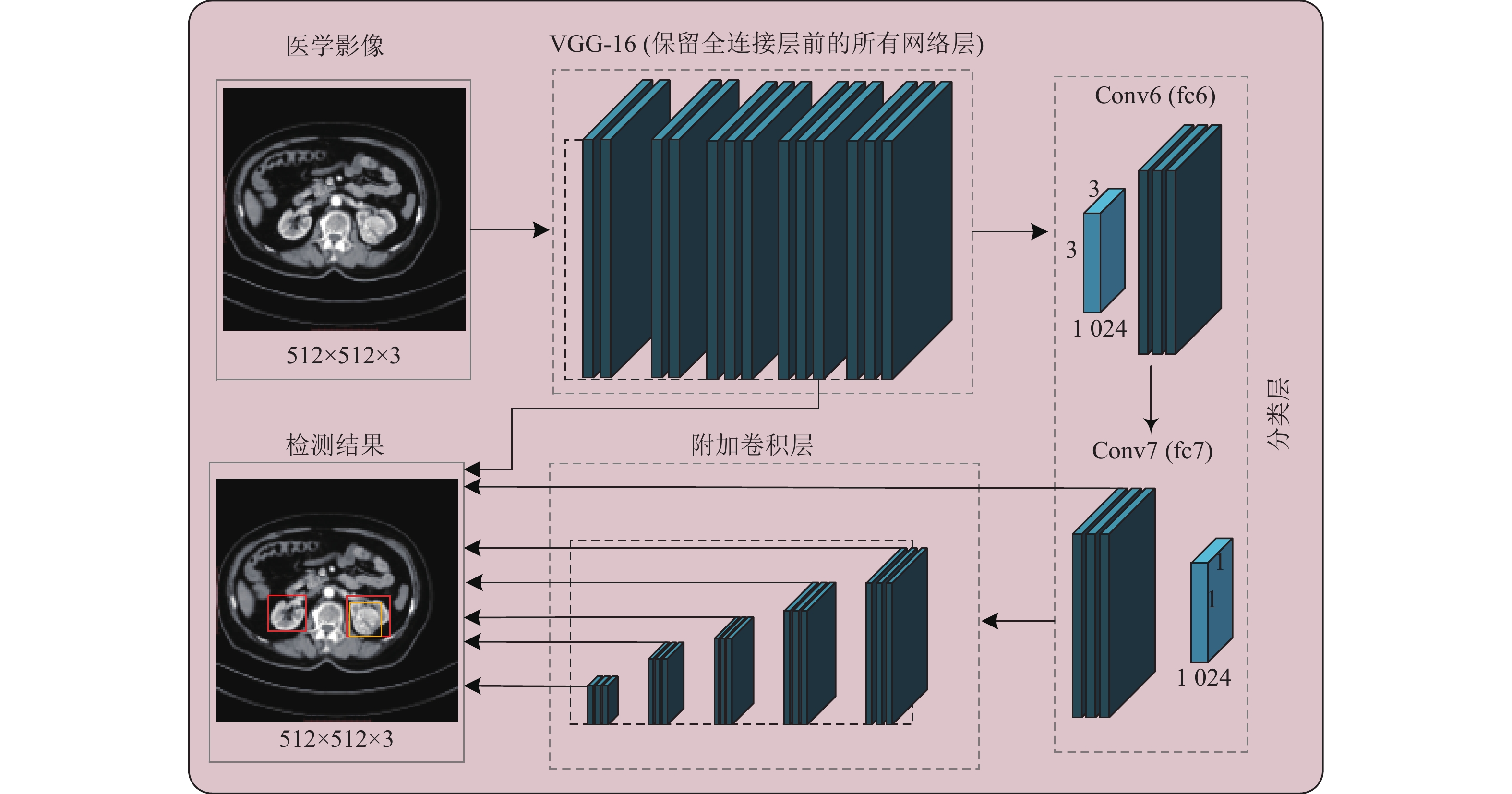
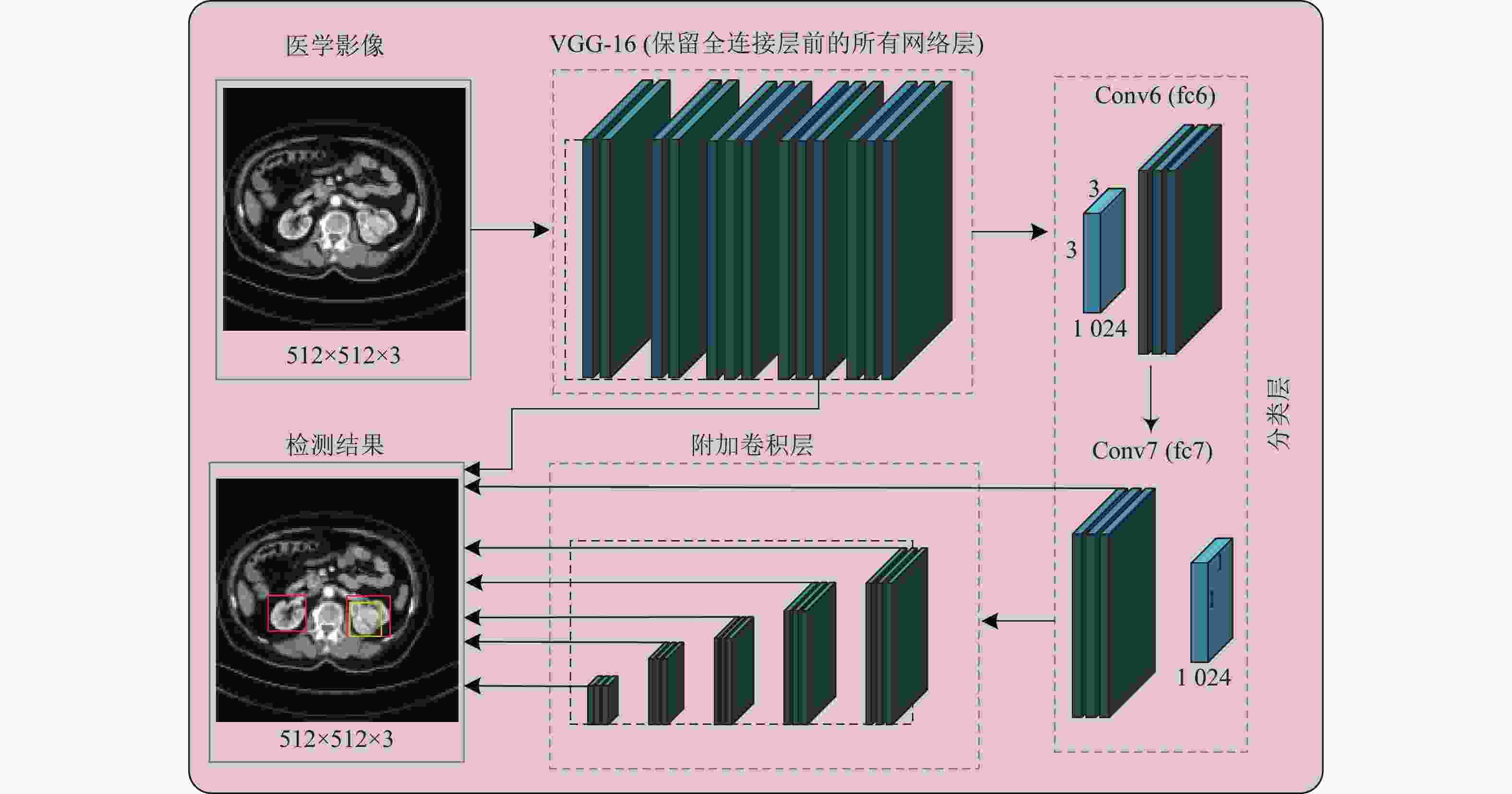
CT影像设备是指利用X射线对人体某一层面进行扫描,最终获得待检测部位的断面信息,即将待检测部位转换为一系列二维图像,感兴趣对象的大小随着层面的不同而发生变化。本文使用文献[24]提出的网络架构SSD作为目标检测网络,该网络在保证检测速度和检测精度的同时,也能很好地对小目标进行检测。SSD的检测流程图如图1所示(图1只对卷积层进行绘制,不包括RELU层和池化层)。
如图1所示,SSD目标检测网络的基础网络是VGG-16,取代VGG-16的全连接层fc6和fc7的是一组辅助卷积层(从conv6到conv12共计7个卷积层)。该模型的输入为512×512大小的医学影像。VGG-16基础网络和新增的卷积层用以获取到不同分辨率的特征图,这些特征图将独立分析输入的医学影像,判断感兴趣的目标是否存在并定位这些目标。本文利用conv4_3、conv7、conv8_2、conv9_2、conv10_2、conv11_2、conv12_2这些大小不同的卷积特征图,并在这些特征图上同时进行交叉熵分类和位置回归,其中深层特征提取层检测医学影像上的大目标,而浅层特征提取层侧重于检测小目标。最后通过非极大值抑制(NMS)过滤冗余的边界框以得到最终的检测结果。
SSD目标检测网络的损失函数为定位损失和分类损失的加权和,总的损失函数为:
$$ L(x,c,l,g) = \frac{1}{N}({L_{{\rm{conf}}}}(x,c) + \gamma {L_{{\rm{loc}}}}(x,l,g)) $$ (1) 式中,
$ L $ 是SSD模型训练过程中总的损失函数;$ {L_{{\rm{conf}}}} $ 表示分类置信度损失函数;$ {L_{{\rm{loc}}}} $ 表示定位损失函数;$ N $ 表示正样本的个数;$ x $ 表示对应预测框的类别信息;$ c $ 表示预测框的置信度;$ l $ 表示预测框的位置信息;$ g $ 表示真实目标框的位置信息;$ \gamma $ 是一个权重参数,用于决定分类置信度损失和定位损失所占的权重关系。 -
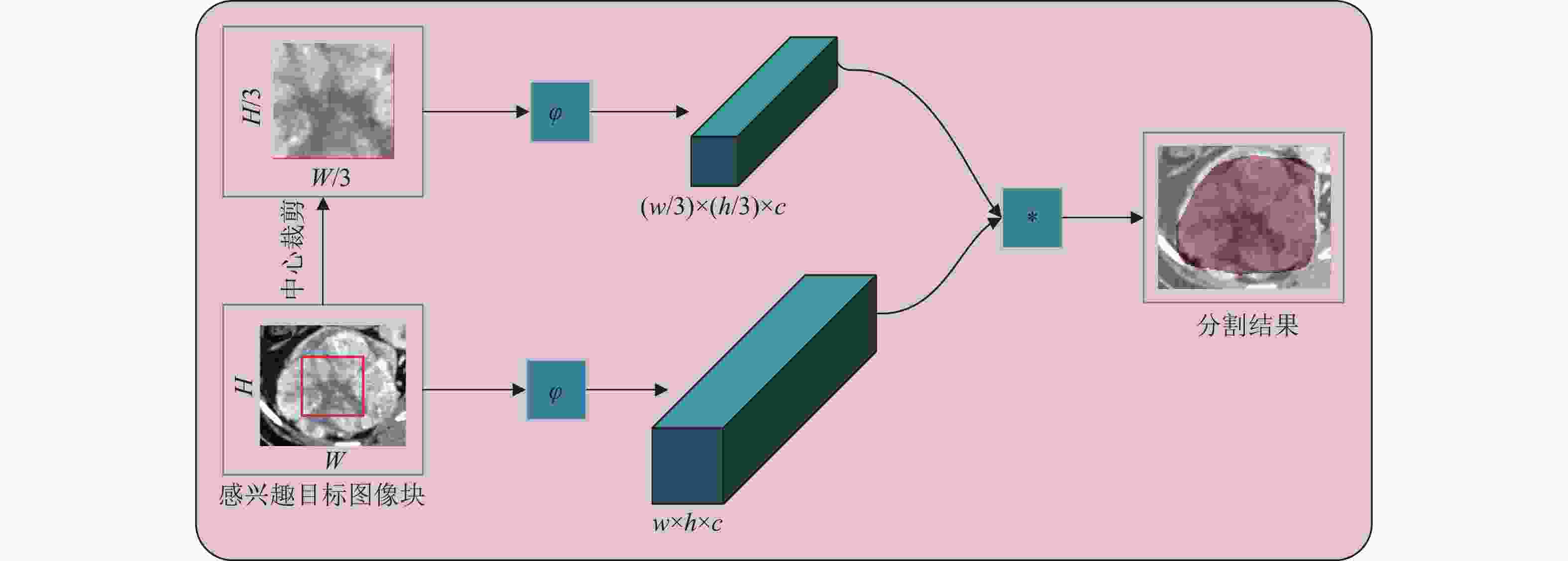
随着卷积神经网络的发展,孪生网络的应用也越来越广泛[26-31]。本文尝试将孪生网络应用到医学影像分割任务中,具体的流程图如图2所示。
根据第2.1小节的描述,SSD目标检测模型可以判断医学影像中感兴趣目标的存在并精确定位这些目标,即SSD目标检测模型的基础网络VGG-16提取的特征可以有效地区分感兴趣的目标和背景。因此,孪生网络使用该基础网络VGG-16作为特征提取器
$ \varphi $ 。如第2.1节所述,SSD模型的输入为
$ 512 \times 512 $ 大小的医学影像,该模型的输出为感兴趣目标的边界盒信息$ ({X_A},{Y_A},{W_A},{H_A}) $ 。搜索对象A是根据边界盒信息对医学影像进行裁剪得到的图像块,大小为$ {W_A} \times {H_A} $ ,示例图像如图2左下角所示。参考图像B一般为图像A的中心区域,大小为$ {W_B} \times {H_B},{W_B} = {W_A}/\alpha , {H_B} = {H_A}/\alpha $ ,当$ \alpha = 3 $ 时,示例图像如图2左上角所示。A和B通过特征提取器$ \varphi $ 得到图像的卷积特征$ {f_A} = \varphi (A) $ 和$ {f_B} = \varphi (B) $ 。孪生网络使用互相关函数$ g $ 来计算图像A上的每个像素点被划分为关键像素点的概率(这个像素点属于感兴趣目标):$$ g(A,B) = \varphi (A) * \varphi (B) $$ (2) 式(2)的输出不是单个的分数,而是在有限网格空间
$ D \in {W_A} \times {H_A} $ 上的一个概率分布图。根据这个概率分布图以及预先设定好的概率阈值$ \beta $ ,以得到感兴趣目标的分割结果:$$ {\rm{seg}} = \left\{ {\begin{array}{*{20}{l}} 1&g(a,b) \geqslant \beta \\ 0&其他 \\ \end{array}} \right. $$ (3) 式中,
$ (a,b) \in D $ ,表示图像A中的一个像素点。当$ {\rm{seg}} = 1 $ 时,这个像素点为关键像素点,即该像素点为图2中分割掩膜下的像素点之一。当$ {\rm{seg}} = 0 $ 时,这个像素点属于背景。 -
本文使用两个公共数据集评估医学影像分割算法,下面简要描述这两个数据集。
肝脏数据集:本文使用的第一个数据集是MICCAI 2017肝肿瘤分割数据集,即LITS17[31]。该数据集包含131名肝细胞癌患者的CT扫描切片,放射科医生分别对CT扫描切片中的肝脏和肿瘤的像素点进行标注。在接下来的实验中,肿瘤分割标签被分类为肝脏分割标签,即数据集被划分为肝脏和背景的二分类问题。此外,本文根据分割标签过滤数据集,得到19159个切片的数据集。每个轴向的CT切片的HU像素阈值设置为[0, 200],即通过窗位的剪切去除噪声和不相关的区域信息,并将所有的切片归一化为[0,1],大部分情况下,CT切片的大小为
$ 512 \times 512 $ ,故调整所有的CT影像切片大小为$ 512 \times 512 $ 。本文将131例肝细胞癌患者划分为训练集和测试集。其中,训练集包含101例患者合14132个切片数据,测试集包含30例患者合4748个切片数据。肾脏数据集:本文使用的第二个公共数据集是Kaggle上的肾脏肿瘤分割数据集,即KITS19[32]。该数据集是由明尼苏达大学医学中心在2010年–2018年期间收集的,包括因肾肿瘤而接受部分肾切除术或根治性肾切除术的患者的CT扫描切片。该数据集共收集了300个肾癌患者的CT切片,其中210个病例用于网络训练和验证,这210例肾癌患者的语义分割掩码由医科学生在放射科医生的监督下进行手工分割,保证了肾脏肿瘤定位的精确性并有效排除肾脏囊肿被标注为肾脏肿瘤的可能性。其余90例肾癌患者用于评估模型的性能,但其语义分割掩码没有公开下载链接,故本文使用验证集评估网络性能。该数据集根据分割标签进行筛选得到39785个切片。每个轴向的CT切片的HU像素阈值设置为包含肾脏的[−200, 500],即通过窗位的剪切去除噪声和不相关的区域信息,并将所有的切片归一化到[0,1],CT切片的大小一般为
$ 512 \times 512 $ ,故调整所有的CT影像切片大小$ 512 \times 512 $ 。根据病例,本文将210例肾癌患者划分为训练集和验证集。其中,训练集包含160例患者合28201个切片数据,验证集包含50例患者合11584个切片数据。 -
使用两个基线模型(UNet[12]和FCN[11])与本文设计的分割模型进行性能比较,用以评估该分割方法的可行性。使用筛子分数(DICE)对分割算法的性能进行评估。筛子分数的计算方法为:
$$ {\rm{Dice}}({S}_{{\rm{GT}}},{S}_{{\rm{DT}}})=\frac{2 ({S}_{{\rm{GT}}}\cap {S}_{{\rm{DT}}})}{|{S}_{{\rm{GT}}}+{S}_{{\rm{DT}}}|} $$ (4) 式中,
$ {S_{{\rm{GT}}}} $ 表示该分割信息由放射科医师手工标注;$ {S_{{\rm{DT}}}} $ 表示该分割信息为分割算法输出的分割结果。由于本文设计的分割算法的基础是目标检测网络,故同样需要对目标检测模型SSD进行评估。一般地,平均准确率AP可以用来评估目标检测网络的性能,该度量的计算为求取精确召回曲线下的面积。AP主要衡量单类别的检测性能,平均准确率均值mAP为AP值在所有类别下的均值。
-
使用深度学习框架pytorch对目标检测网络SSD和孪生网络进行编程实现。目标检测网络SSD在单张英伟达1080Ti显卡上进行训练,需要注意的是,孪生网络的特征提取器为目标检测模型SSD的基础网络VGG-16,所以只需要对目标检测网络进行训练即可。
SSD目标检测网络基于预训练好的VGG-16模型进行训练,VGG-16的预训练在ImageNet数据集上完成。在训练阶段,网络的单次迭代接收的输入数据量为16,共训练200轮,使用Adam优化器[42]进行网络训练,设置初始学习率为0.001,动量参数设置为
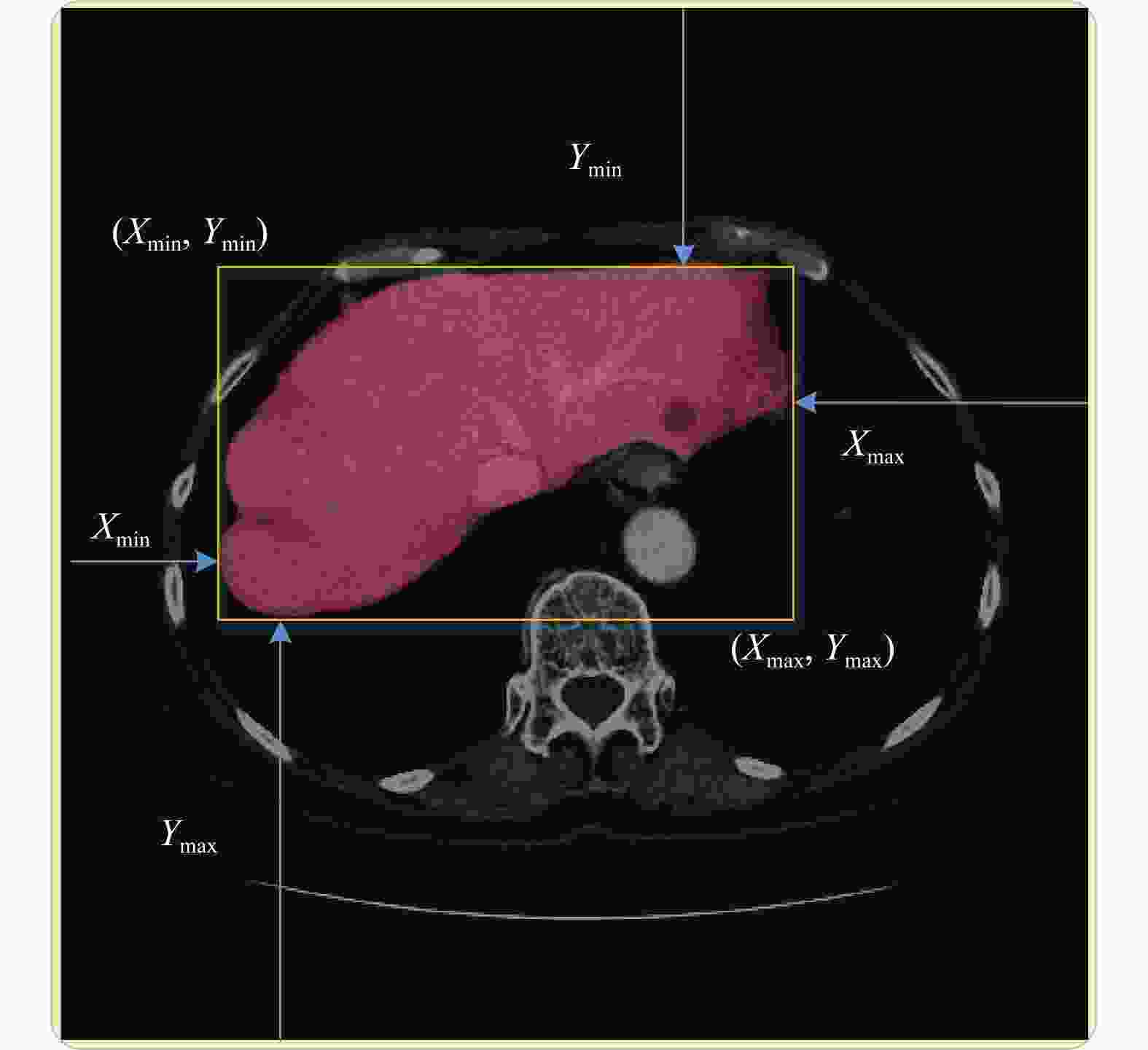
$ {\beta _1} = 0.9,{\beta _2} = 0.999 $ 。LITS17和KITS19所提供的标注都是像素级别的,为了根据分割标签获取检测标签,可以同时从图像的4个边界向分割标签进行靠近,如图3中的箭头所示。
医学影像的大小为
$ W \times H $ ,$ l(x = i,y) $ 表示医学影像的第$ i $ 列,$ l(x,y = j) $ 表示医学影像的第$ j $ 行,那么有$ l(x = 0,y) $ 表示图像的左边界,$ l(x = W,y) $ 表示图像的右边界,$ l(x,y = 0) $ 表示图像的上边界,$ l(x,y = H) $ 表示图像的下边界。该医学影像的边界盒信息$ ({x_{\min }},{y_{\min ,}}{x_{\max }},{y_{\max }}) $ 的计算方式如算法1所示。算法1 根据分割标签求取检测标签
输入:分割标签S,医学影像高度H,医学影像宽度W
输出:检测标签
$ ({x_{\min }},{y_{\min ,}}{x_{\max }},{y_{\max }}) $ $ i \leftarrow 0,j \leftarrow 0 $ while
$ l(x = i,y) \cap S = = {\rm{null}} $ do$ i \leftarrow i + 1 $ end while
$ {x_{\min }} = i - 1 $ while
$ l(x,y = j) \cap S = = {\rm{null}} $ do$ j \leftarrow j + 1 $ end while
$ {y_{\min }} = j - 1 $ $ i \leftarrow W - 1,j \leftarrow H - 1 $ while
$ l(x = i,y) \cap S = = {\rm{null}} $ do$ i \leftarrow i - 1 $ end while
$ {x_{\max }} = i + 1 $ while
$ l(x,y = j) \cap S = = {\rm{null}} $ do$ j \leftarrow j - 1 $ end while
$ {y_{\max }} = j + 1 $ -
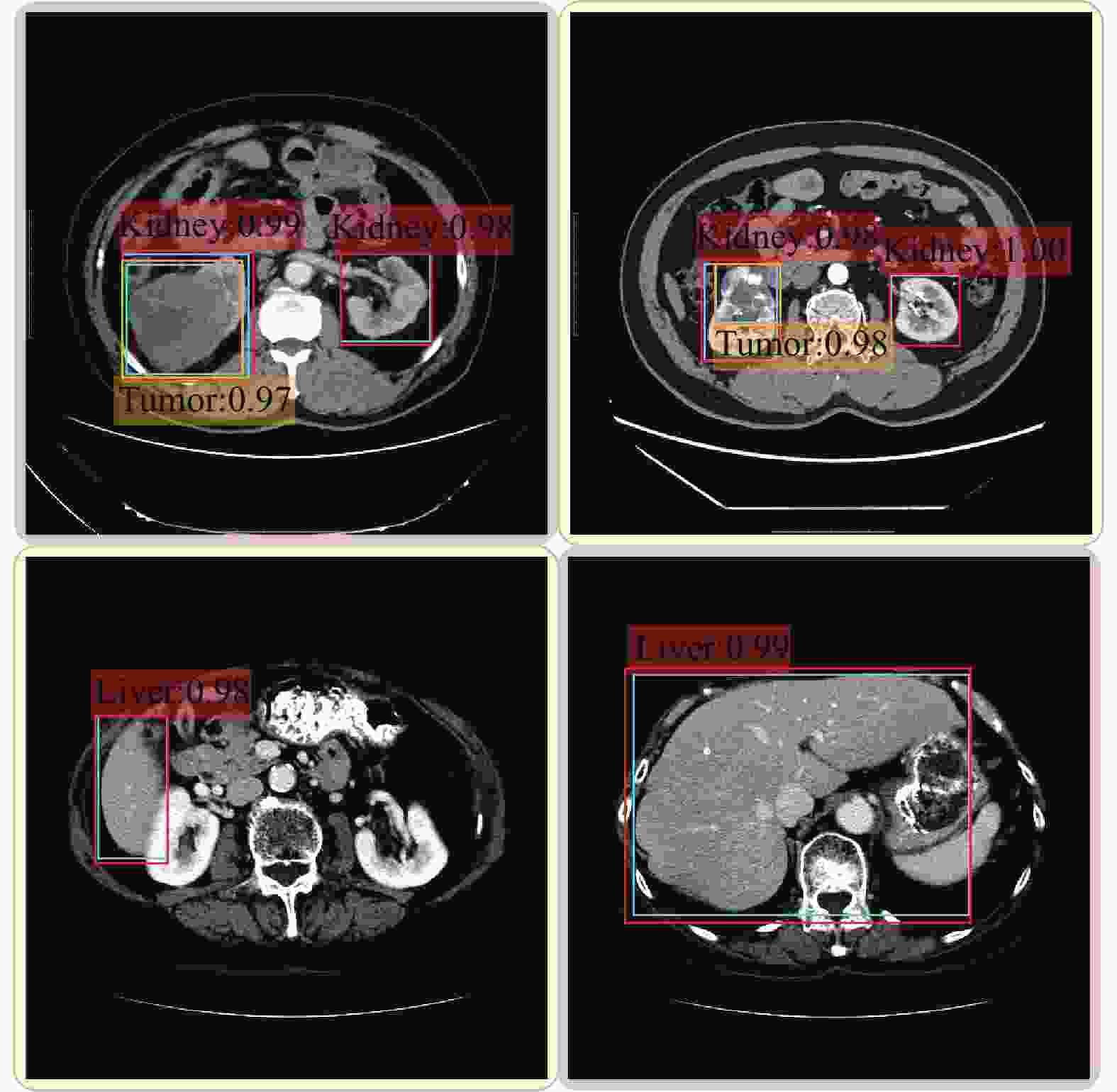
使用两个不同的医学影像数据集分别对SSD检测网络进行训练,将得到的模型分别命名为SSD_Kidney和SSD_Liver。图4为SSD检测模型在两个医学影像数据集上的检测结果示例图,前两个子图为SSD_Kidney在数据集KITS19验证集上的肾脏及肾脏肿瘤的检测结果,后两个子图为SSD_Liver在数据集LITS17验证集上肝脏的检测结果,图中的虚线为检测模型输出的检测结果。表1显示了两个检测模型在数据集上的检测结果,可以看出两个检测模型的器官检测平均准确率都可以达到91%以上,肿瘤检测平均准确率也接近于90%。图4的示例结果和表1的统计结果均可以验证检测模型的性能,这在一定程度上证明SSD检测模型的基础网络VGG-16可以对医学影像中的背景和感兴趣目标进行精准判别。
表 1 不同检测模型的性能比较
模型 器官/病变组织 AP/% SSD_Liver 肝脏 93.87 SSD_Kidney 肾脏 91.68 肾肿瘤 89.32 -
按照SSD模型预测得到的边界盒信息对医学影像进行裁剪,得到感兴趣目标的图像块。在本节中,孪生网络预测图像块中每个像素点被划分为关键像素点的概率,通过概率分布
$ {\rm{seg}} $ 和预先设定好的概率阈值$ \beta $ 得到最终的分割结果。如3.4节所述,SSD模型的基础网络VGG-16可以精准地将感兴趣目标和背景进行区分,因此,第2.2节中所描述的孪生网络使用该VGG-16网络作为特征提取器
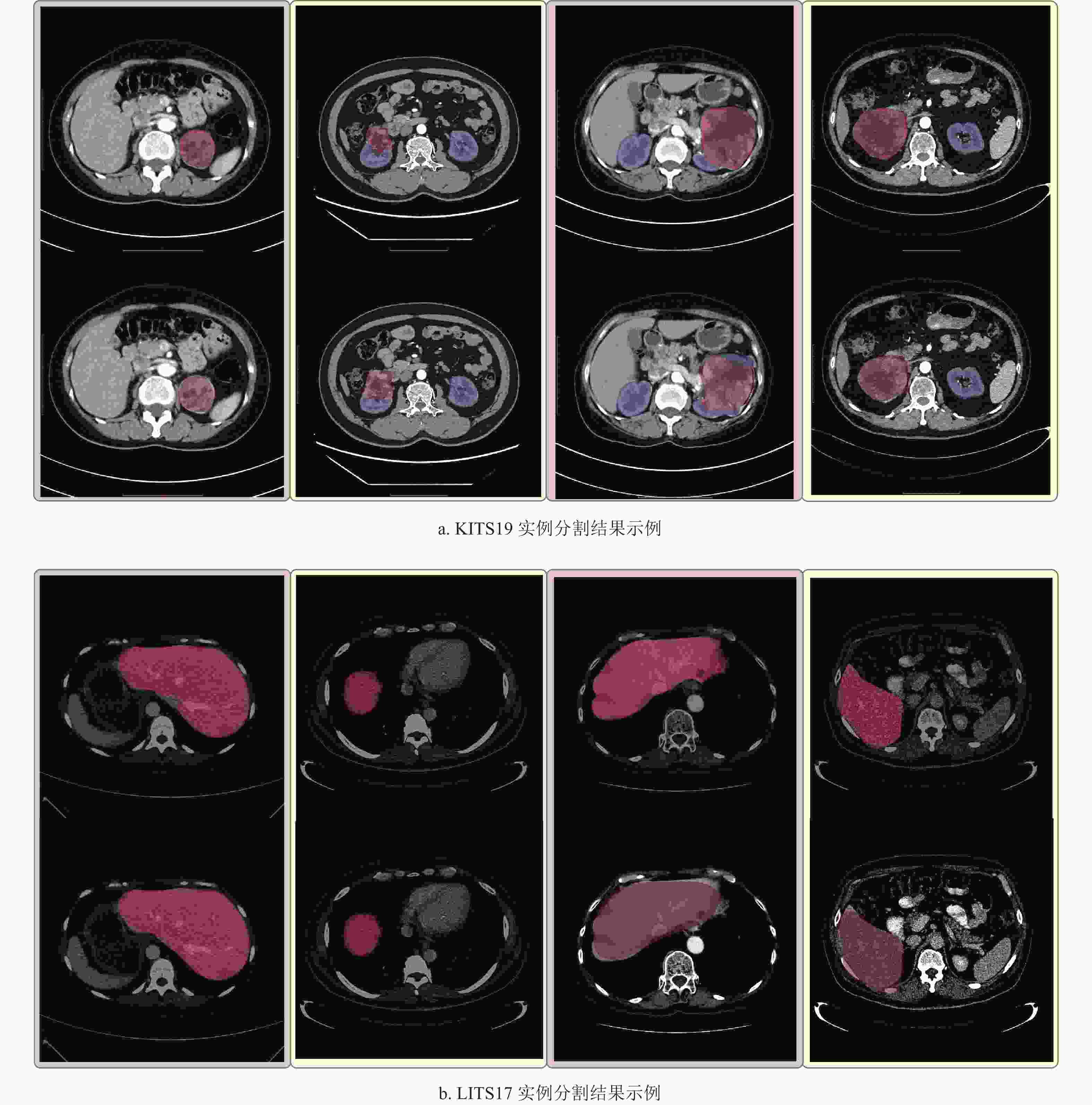
$ \varphi $ 。孪生网络计算每个像素点被分类为关键像素点的概率$ p $ ,当$ p \geqslant \beta $ 时,对应的像素点被分类为关键像素点,从而得到最终的分割掩膜。图5显示了基于目标检测的医学影像分割算法在两个不同数据集上的分割结果,其中,每个子图的第一行的分割掩膜由放射科医师人工标注,第二行的分割掩膜由分割模型输出。表2将本文设计的分割算法与两种常见的二维深度学习分割方法 UNet[12]和 FCN[11]进行比较。根据表2的数据可以发现,本文设计的分割算法的性能随着预定义的概率阈值
$ \beta $ 的变化而发生变化。当$ \beta $ 设置过小时,很多属于背景的像素点被分类为感兴趣目标的关键像素点,导致模型性能急剧下降,当$ \beta = 0.5 $ 时,模型性能至少下降了7%。当$ \beta $ 设置过大时,很多属于感兴趣目标的边界像素点被分类为背景像素点,这也会导致模型性能的下降,当$ \beta = 0.9 $ 时,在肝脏数据集上的模型性能从90.15%下降到了78.27%。当然,$ \alpha $ 参数设置不合理同样会造成模型性能的下降,这主要是因为孪生网络无法通过参考对象获取到精细的感兴趣目标的特征,如表2所示,当$ \alpha = 2 $ 和$ \alpha = 5 $ 时,模型性能都显著降低。当$ \alpha = 3,\beta = 0.7 $ 时,本文设计的分割算法的性能略胜FCN,并极其接近UNet,这表明即使在只存在边界信息的情况下,同样可以对医学影像进行深层次的分析与理解。表 2 不同分割模型的性能比较
模型 参数 LITS17分割结果/% KITS19分割结果/% $ \alpha $ $ \beta $ 肾脏 肿瘤 FCN − − 89.35 93.22 57.85 UNet − − 90.21 95.56 62.35 本文 3 0.5 83.45 87.63 53.36 3 0.7 90.15 94.93 61.93 3 0.9 78.27 83.39 45.99 2 0.7 83.44 87.92 48.21 5 0.7 87.51 91.94 59.33 -
精准的医学影像分割算法可以帮助医生轻松、快速、准确地分析医学影像,有效减轻放射科医师的工作量。随着深度卷积神经网络的发展,一些研究人员尝试使用神经网络来解决医学影像分析任务。带像素级标签的医学影像数据集很难获取,主要原因有:1)为保证医学影像数据集标签的可靠性,需要资深的放射科医师绘制或重新检查标签; 2)与医学影像数量的增加速度相比,能够标记医学影像的放射科医师是有限的; 3) 一般情况下,放射科医师只关注病变器官或组织。因此,本文提出一种基于目标检测的医学影像分割算法,以降低医学影像分割算法对像素级标签的依赖程度。
尽管本文设计的医学影像分割算法的性能提升不是很明显,但其显著降低了医学影像分割算法对于像素级标签的依赖程度,如果使用3D目标检测网络对感兴趣目标进行检测,并使用三维的卷积神经网络作为孪生网络的特征提取器,那么,该算法的性能可以达到进一步提升。在医学影像中,器官位置的分布是比较固定的,所以本文提出的算法在器官分割上可以取得较好的结果;当然,因为病灶位置分布不确定、形状不规则,那么,病灶图像块的中心区域并不一定是关键像素点,这种情况可以考虑加入人工辅助以选择关注像素点所在的区域。
未来会考虑添加更多的医学影像来提升目标检测网络的性能;当然,也可以选择一种更好的目标检测框架来提取更准确的特征。由此可见,本文设计的分割算法还有很大的改进空间。该方法还可用于分析其他医学图像数据集,如肺结节和脑肿瘤的分割。
Medical Image Segmentation Based on Object Detection
-
摘要: 提出了一种基于目标检测的医学影像分割算法。目标检测网络可以从医学影像中获取到感兴趣目标(一般为器官或病变组织)的精确位置信息,根据位置信息对医学影像进行裁剪得到感兴趣目标的图像块。以感兴趣目标图像块的关键区域作为参考对象,以该图像块作为搜索对象,孪生网络可以获取到图像块中的每一个像素点被分类为关键像素点的概率,根据预先设定好的概率阈值可得到感兴趣目标的分割结果。使用肝脏(LITS17)和肾脏(KITS19)两个数据集对上述医学影像分割算法进行评估。实验结果表明该分割方法可以较准确地对医学影像中的感兴趣目标进行分割。Abstract: Abstract This paper proposes a medical image segmentation algorithm based on object detection. With the help of an object detection network, the precise locations of the object of interest (usually an organ or diseased tissue) in medical images are obtained. And the medical images are cropped according to locations obtained by the object detection network to gain the image patches of objects of interest. By taking the key center area of the image patch of objects of interest as the examplar image and this image patch as the search image, the pixel-wise classification of this image patch is achieved by one Siamese network with the help of a pre-defined threshold. Two datasets (LITS17 and KITS19) are used to evaluate our algorithm. And experiments show that the proposed method can accurately segment organs or diseased tissues in medical images.
-
Key words:
- image segmentation /
- medical image /
- object detection /
- Siamese network
-
表 1 不同检测模型的性能比较
模型 器官/病变组织 AP/% SSD_Liver 肝脏 93.87 SSD_Kidney 肾脏 91.68 肾肿瘤 89.32 表 2 不同分割模型的性能比较
模型 参数 LITS17分割结果/% KITS19分割结果/% $ \alpha $ $ \beta $ 肾脏 肿瘤 FCN − − 89.35 93.22 57.85 UNet − − 90.21 95.56 62.35 本文 3 0.5 83.45 87.63 53.36 3 0.7 90.15 94.93 61.93 3 0.9 78.27 83.39 45.99 2 0.7 83.44 87.92 48.21 5 0.7 87.51 91.94 59.33 -
[1] AGGARWAL P, VIG R, BHADORIA S, et al. Role of segmentation in medical imaging: A comparative study[J]. International Journal of Computer Applications, 2011, 29(1): 54-61. doi: 10.5120/3525-4803 [2] INDA M M, BONAVIA R, SEOANE J. Glioblastoma multiforme: A look inside its heterogeneous nature[J]. Cancers, 2014, 6(1): 226-239. doi: 10.3390/cancers6010226 [3] THOMSON D, BOYLAN C, LIPTROT T, et al. Evaluation of an automatic segmentation algorithm for definition of head and neck organs at risk[J]. Radiation Oncology, 2014, 9(1): 1-12. doi: 10.1186/1748-717X-9-1 [4] ERMIŞ E, JUNGO A, POEL R, et al. Fully automated brain resection cavity delineation for radiation target volume definition in glioblastoma patients using deep learning[J]. Radiation oncology, 2020, 15(1): 1-10. doi: 10.1186/s13014-019-1449-z [5] KRIZHEVSKY A, SUTSKEVER I, HINTON G E. Imagenet classification with deep convolutional neural networks[C]//26th Annual Conference on Neural Information Processing Systems. [S.l.]: NIPS, 2012: 1097-1105. [6] DENG J, DONG W, SOCHER R, et al. ImageNet: A large-scale hierarchical image database[C]//2009 IEEE Conference on Computer Vision and Pattern Recognition. New York: IEEE, 2009: 248-255. [7] SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[C]//3rd International Conference on Learning Representations, ICLR 2015. [S.l.]: ICLR, 2015: 38. [8] TOUVRON H, VEDALDI A, DOUZE M, et al. Fixing the train-test resolution discrepancy[C]//Advances in Neural Information Processing Systems. [S.l.]: NIPS, 2019: 8250-8260. [9] DUAN K, XIE L, QI H, et al. Corner proposal network for anchor-free, two-stage object detection[C]//European Conference on Computer Vision. Cham: Springer, 2020: 399-416. [10] HAN J, DING J, XUE N, et al. ReDet: A rotation-equivariant detector for aerial object detection[C]//2021 IEEE Conference on Computer Vision and Pattern Recognition. New York: IEEE, 2021: 2786-2795. [11] LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation[C]//2015 IEEE Conference on Computer Vision and Pattern Recognition. New York: IEEE, 2015: 3431-3440. [12] RONNEBERGER O, FISCHER P, BROX T. U-net: Convolutional networks for biomedical image segmentation[C]//Lecture Notes in Computer Science. Berlin: Springer, 2015: 234-241. [13] SAHINER B, PEZESHK A, HADJIISKI L M, et al. Deep learning in medical imaging and radiation therapy[J]. Medical Physics, 2019, 46(1): e1-e36. doi: 10.1002/mp.13264 [14] DU G, CAO X, LIANG J, et al. Medical image segmentation based on u-net: A review[J]. Journal of Imaging Science and Technology, 2020, 64(2): 20508-1-20508-12. [15] TAN M, LE Q. Efficientnet: Rethinking model scaling for convolutional neural networks[C]//International Conference on Machine Learning. [S.l.]: JMLR, 2019: 6105-6114. [16] KUZNETSOVA A, ROM H, ALLDRIN N, et al. The open images dataset V4: Unified image classification, object detection, and visual relationship detection at scale[J]. International Journal of Computer Vision, 2020, 128(7): 1956-1981. doi: 10.1007/s11263-020-01316-z [17] GIRSHICK R, DONAHUE J, DARRELL T, et al. Rich feature hierarchies for accurate object detection and semantic segmentation[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. New York: IEEE, 2014: 580-587. [18] UIJLINGS J R R, VAN DE SANDE K E A, GEVERS T, et al. Selective search for object recognition[J]. International Journal of Computer Vision, 2013, 104(2): 154-171. doi: 10.1007/s11263-013-0620-5 [19] REN S, HE K, GIRSHICK R, et al. Faster R-CNN: Towards real-time object detection with region proposal networks[C]//Advances in Neural Information Processing Systems. [S.l.]: NIPS, 2015: 91-99. [20] HE K, GKIOXARI G, DOLLÁR P, et al. Mask R-CNN[C]//Proceedings of the IEEE International Conference on Computer Vision. New York: IEEE, 2017: 2961-2969. [21] LU X, LI B, YUE Y, et al. Grid R-CNN[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. New York: IEEE, 2019: 7363-7372. [22] PANG J, CHEN K, SHI J, et al. Libra R-CNN: Towards balanced learning for object detection[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. New York: IEEE, 2019: 821-830. [23] REDMON J, DIVVALA S, GIRSHICK R, et al. You only look once: Unified, real-time object detection[C]// Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. [S.l.]: IEEE, 2016: 779-788. [24] LIU W, ANGUELOV D, ERHAN D, et al. SSD: Single shot multibox detector[C]//European Conference on Computer Vision. Cham: Springer, 2016: 21-37. [25] BROMLEY J, GUYON I, LECUN Y, et al. Signature verification using a “siamese” time delay neural network[J]. International Journal of Pattern Recognition and Artificial Intelligence, 1993, 7(4): 669-688. doi: 10.1142/S0218001493000339 [26] ZAGORUYKO S, KOMODAKIS N. Learning to compare image patches via convolutional neural networks[C]// Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. New York: IEEE, 2015: 4353-4361. [27] KOCH G, ZEMEL R, SALAKHUTDINOV R. Siamese neural networks for one-shot image recognition[C]// International Conference on Machine Learning. [S.l.]: IMLS, 2015: 2(1). [28] WU L, WANG Y, GAO J, et al. Where-and-when to look: Deep siamese attention networks for video-based person re-identification[J]. IEEE Transactions on Multimedia, 2018, 21(6): 1412-1424. [29] BERTINETTO L, VALMADRE J, HENRIQUES J F, et al. Fully-convolutional siamese networks for object tracking[C]//European Conference on Computer Vision. Cham: Springer, 2016: 850-865. [30] YU Y, XIONG Y, HUANG W, et al. Deformable Siamese attention networks for visual object tracking[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. New York: IEEE, 2020: 6728-6737. [31] SHUAI B, BERNESHAWI A, LI X, et al. Siammot: Siamese multi-object tracking[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. [S.l.]: IEEE, 2021: 12372-12382. [32] CIRESAN D, GIUSTI A, GAMBARDELLA L, et al. Deep neural networks segment neuronal membranes in electron microscopy images[C]//26th Annual Conference on Neural Information Processing Systems. [S.l.]: NIPS, 2012: 2843-2851. [33] PRASOON A, PETERSEN K, IGEL C, et al. Deep feature learning for knee cartilage segmentation using a triplanar convolutional neural network[C]//Lecture Notes in Computer Science, 16th International Conference on Medical Image Computing and Computer Assisted Intervention. Berlin: Springer, 2013: 246-253. [34] WANG S, ZHOU M, LIU Z, et al. Central focused convolutional neural networks: Developing a data-driven model for lung nodule segmentation[J]. Medical Image Analysis, 2017, 40: 172-183. doi: 10.1016/j.media.2017.06.014 [35] SHI Y, YANG W, GAO Y, et al. Does manual delineation only provide the side information in CT prostate segmentation?[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2017: 692-700. [36] YU Q, SHI Y, SUN J, et al. Crossbar-net: A novel convolutional neural network for kidney tumor segmentation in ct images[J]. IEEE Transactions on Image Processing, 2019, 28(8): 4060-4074. doi: 10.1109/TIP.2019.2905537 [37] HOU X, XIE C, LI F, et al. A triple-stage self-guided network for kidney tumor segmentation[C]//2020 IEEE 17th International Symposium on Biomedical Imaging (ISBI). New York: IEEE, 2020: 341-344. [38] 鲁浩达, 徐军, 刘利卉, 等. 基于深度卷积神经网络的肾透明细胞癌细胞核分割[J]. 生物医学工程研究, 2017, 36(4): 340-345. doi: 10.19529/j.cnki.1672-6278.2017.04.13 LU H, XU J, LIU L, et al. Nuclear segmentation of clear cell renal cell carcinoma based on deep convolutional neural networks[J]. Journal of Biomedical Engineering Research, 2017, 36(4): 340-345. doi: 10.19529/j.cnki.1672-6278.2017.04.13 [39] GIRUM K B, CRÉHANGE G, HUSSAIN R, et al. Fast interactive medical image segmentation with weakly supervised deep learning method[J]. International Journal of Computer Assisted Radiology and Surgery, 2020, 15(9): 1437-1444. doi: 10.1007/s11548-020-02223-x [40] WANG G, LI W, ZULUAGA M A, et al. Interactive medical image segmentation using deep learning with image-specific fine tuning[J]. IEEE Transactions on Medical Imaging, 2018, 37(7): 1562-1573. doi: 10.1109/TMI.2018.2791721 [41] JU M, LEE M, LEE J, et al. All you need is a few dots to label CT images for organ segmentation[J]. Applied Sciences, 2022, 12(3): 1328. doi: 10.3390/app12031328 [42] KINGMA D P, BA J. Adam: A method for stochastic optimization[C]//3rd International Conference on Learning Representations. [S.l.]: ICLR, 2015: 75. -

 ISSN
ISSN 



 下载:
下载: