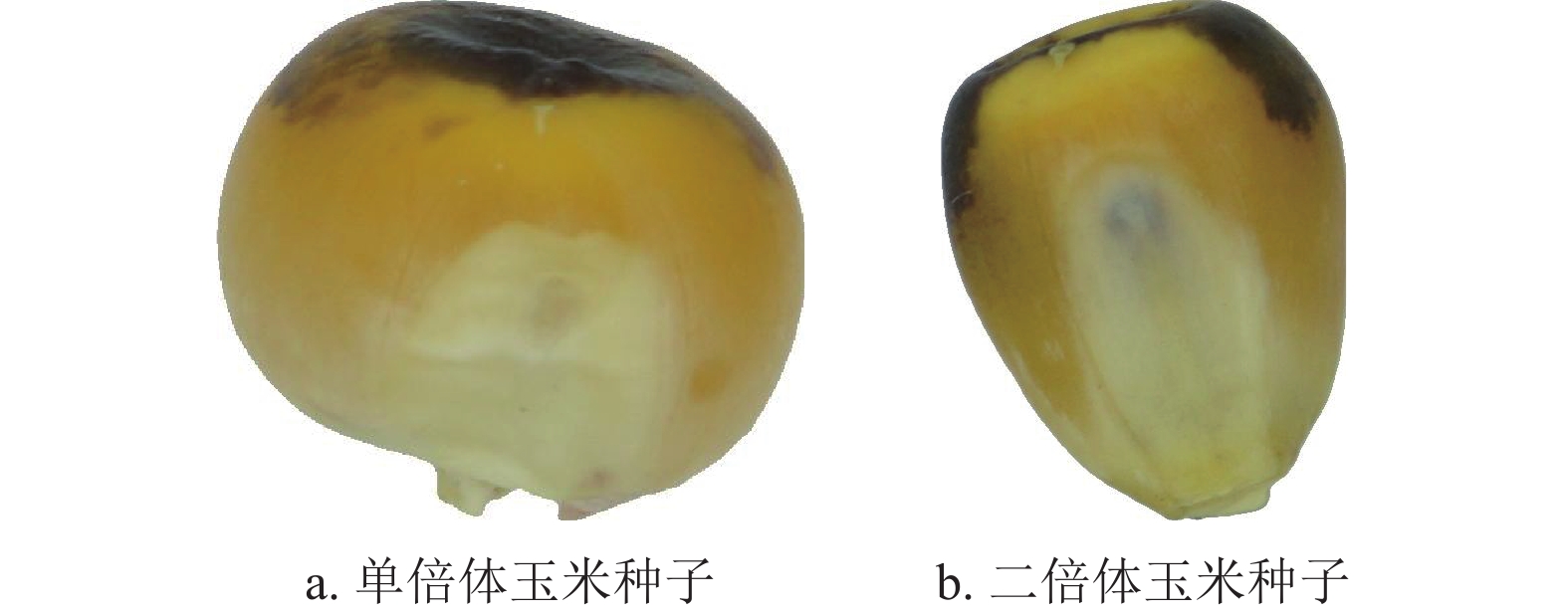
-
单倍体育种是一种植物育种方法,其原理是利用人工培育或自然发生的单倍体植株,通过加倍该植株染色体获得纯合的二倍体植株[1]。玉米单倍体育种[2]主要包括单倍体生产、单倍体鉴定、单倍体加倍产生单二倍体(diploid haploid, DH)系、DH系育种几个步骤[3],具有如下优点:1)获得纯系快,育种周期短;2)无基因互作效应,选择准确性高;3)所获DH系高度纯合和稳定[4];4)所得DH群体遗传类型丰富。单倍体自然发生的概率仅为0.1%[5],故实际单倍体育种均采用人工诱导单倍体技术。通过诱导剂进行人工诱导的DH率为8%以上[6],虽然人工诱导有如此高的DH率,但诱导后仍有问题待解决,即如何从众多单二倍体玉米种子中鉴别出单倍体种子。
文献[7]使用核磁共振测定玉米种子含油量,提出一种基于最小二乘误差的单倍体和二倍体的分离方法。根据模型结果,最小二乘误差法可在样本较少的情况下快速确定单倍体和二倍体含油量的阈值,识别率达90%以上。文献[8]设计了一套玉米单倍体种子的分选系统,该系统包括种子输送、图像采集与处理、分选和系统控制单元,系统根据玉米胚和玉米胚乳顶部的颜色特征分选单倍体,能够实现500粒/min的分选速度,对杂合种子的正确识别率为90%,分选成功率为80%。文献[9-10]使用从灰度共生矩阵中获得的文体特征识别单倍体和二倍体玉米种子。这些特征被用于决策树(decision tree, DT)、k-最近邻和人工神经网络分类器的输入,结果显示,最佳结果由DT获得,其分类成功率达84.48%。文献[11]使用红外光谱仪对玉米种子的红外光谱进行采集,然后将数据预处理后使用核变换提取特征,最后使用不同的特征提取算法建立鉴别模型。结果显示,采用核局部投影的特征提取算法在两组测试集的识别率分别达95.71%和96.43%。
现有研究中,使用传统图像处理[8]或机器学习进行图像分类[9-10]对玉米单倍体种子进行识别的成功率不高,未超过90%。使用核磁共振[7]或红外光谱[11]对玉米单倍体种子的识别率可达90%以上,但核磁共振仪和红外光谱仪设备昂贵,不具备普适性。当前深度学习技术发展迅速,卷积神经网络作为深度学习的一种网络结构,在图像、声音、语音等领域的研究均有很大进展。玉米种子R1-nj基因的表达会导致花青素沉积使籽粒显深紫色[12],单倍体只在胚乳顶端显示深紫色而二倍体会在胚乳顶端和胚胎都显示深紫色。使用卷积神经网络能够提取单倍体和二倍体玉米种子的不同颜色特征,本文以玉米单倍体和二倍体种子图像为数据集,建立卷积神经网络,进行玉米单倍体种子识别。本文的卷积神经网络将在内核选择性网络(selective kernel networks, SKNet)[13]的基础上加以改进,与DenseNet[14]、ResNet[15]和VGGNet[16]进行性能比较。
-
本研究所使用数据集为网络公开数据集,该数据集共有3000粒玉米种子,包括1230个单倍体和1770个二倍体。图像中玉米种子放置为4行5列共20粒,胚胎处于向上位置且彼此不接触。所有图像均使用Sony SLT-A58相机和Sony SAL 100 mm f/2.8微距镜头拍摄,图像均在“M”模式下拍摄,图像参数为100 ISO、1/125 s的快门速度、f/9的光圈、150 mm焦距和55 cm的相机距离,图像的最终分辨率为5459×3632。
由于每张图像包括20粒玉米种子,所以最终数据集对原始图像进行图像分割后获得。根据经验选定150作为阈值将原始图像进行二值化,通过使用玉米种子的轮廓线计算玉米种子的边界框。通过二值图和边界框裁剪每粒玉米种子的图像单独存储,如图1所示。
-
数据增强是一种正则化技术,用以增加数据量和数据的多样性[17]。常用数据增强方法包括旋转、翻转、缩放、裁剪、位移、高斯噪声等。原数据集包含3000粒玉米种子的图像,每粒种子单独保存为一幅图像,即一共3000幅图像。对原始数据集中的3000幅图像进行数据增强,数据增强方式选择旋转和翻转,其中旋转包括旋转90°、180°和270°。通过数据增强把数据集数量扩充至15000张。
-
卷积神经网络是一种深度学习网络架构,该网络由一些连续的层组成,不同的层具有不同的功能,如卷积层、池化层、Dropout和全连接层等。卷积层是卷积神经网络的最基本层,通过卷积核与输入特征图进行卷积运算,抽取特征图蕴藏的抽象特征。为降低运算复杂度和防止过拟合,卷积层后紧跟池化层,简化特征图的信息维度,常用池化方式包括平均池化、L2范数池化和最大池化,其中最大池化因其速度快和收敛性好而被广泛使用[18]。卷积神经网络通常设置全连接层,将图像空间分布的特征映射到样本标签空间,通过softmax输出类别概率并实现分类。
VGG是一个被广泛使用的卷积神经网络,主要研究卷积神经网络的深度对大规模图像识别的精度影响[16],该模型将卷积神经网络的深度加到16~19层,获得了显著的性能提升。
ResNet模型采用残差学习简化网络训练[15],模型的核心问题是网络的退化问题[19]。网络退化是指网络的训练精度随着网络深度的增加而趋于饱和,如果继续增加深度,训练精度反而会下降。网络退化的原因是随着网络深度的增加,梯度在传递的过程中逐渐消失。由于卷积神经网络中采用非线性激活函数,从输入到输出的过程几乎不可逆,造成许多信息损失。ResNet通过恒等映射,把输入的结果传递到输出让深层网络至少保持浅层网络的水平,使得网络的深度获得大幅提升。
DenseNet借鉴ResNet的思想将网络分为多块,每块中的网络均与后续每层连接,则拥有L层的块包括L(L+1)/2个连接。DenseNet网络减轻梯度消失问题,加强特征传播,鼓励特征重用,显著减少了参数数量[14]。
-
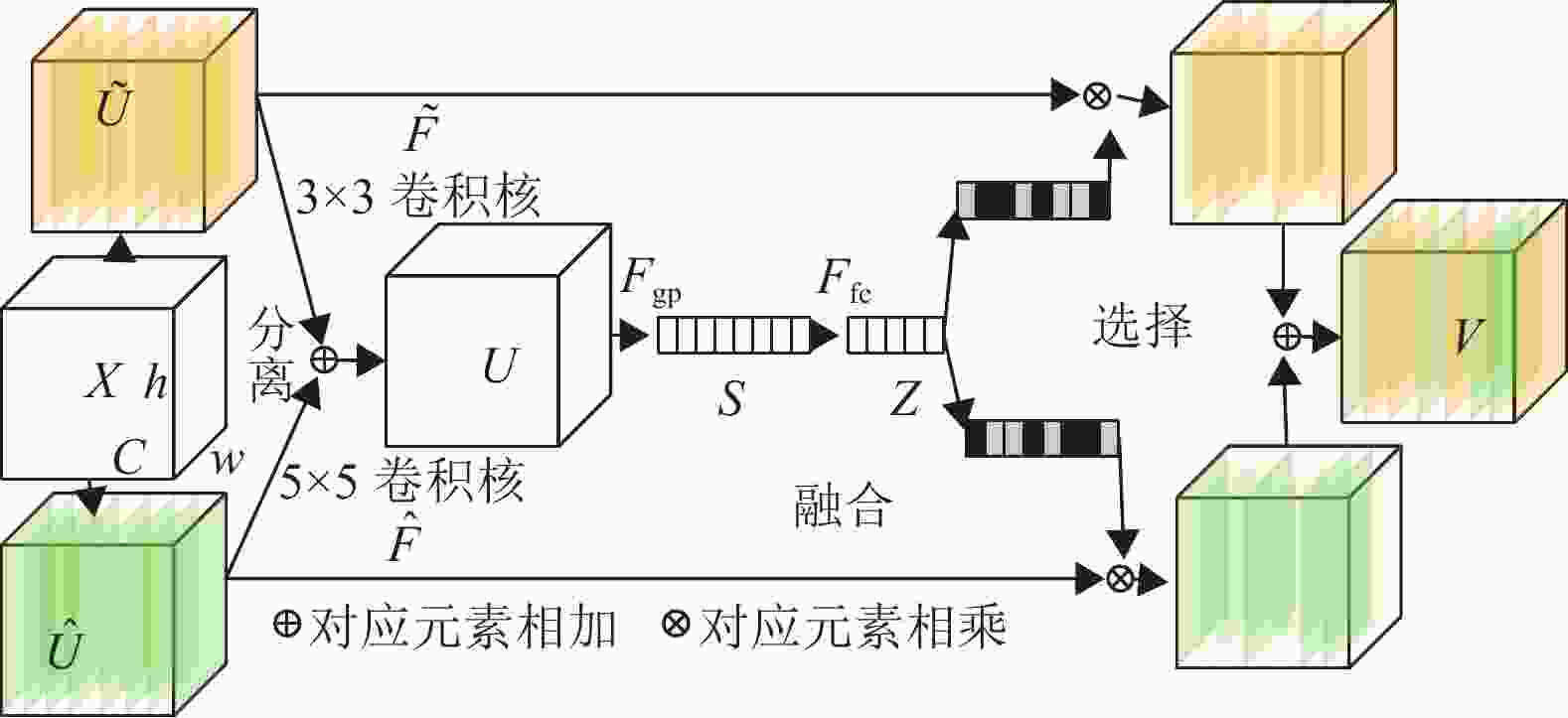
SKNet是一个轻量级的嵌入式模块,其关键是选择性内核单元(selective kernel, SK)的构建块,使用基于softmax的注意力机制融合具有不同卷积核大小的多个分支[13],对这些分支的不同注意力会产生不同大小的融合层神经元有效感受野。在SK卷积的前后分别加上一个1×1卷积,即可构成选择性内核单元,将多个SK构建块堆叠而构建的网络称为SKNet。在SK构建块中,需对3个重要超参数进行设置:SK构建块需融合的分支数M,每个分支的分组卷积的分组数G,融合操作中全连接的压缩系数r。图2为SKNet的核心模块,该模块分为3个部分:分离、融合和选择。
1)分离:输入特征X经过两个具有不同大小的卷积核分支,卷积获得特征图
$\tilde U$ 和特征图$ \hat{U} $ 。2)融合:不同分支的特征图对应元素相加,融合成特征图U,然后对特征图U使用全局平均池化[19],全局平均池化后特征图大小从h×w变为1×1,可理解为特征图从三维转换为一维。紧接着对一维向量进行全连接操作再进行降维,使一维向量的通道数量从C降低到d,d=max(C/r, dmin),其中dmin表示d的最小值,改变r即可改变d的大小。
3)选择:对降维的向量再进行升维,把通道数增加到特征图
$\tilde U$ 和特征图$ \hat{U} $ 的通道数量,然后对向量使用softmax获得a、b两个权重矩阵,使用权重矩阵对特征图$\tilde U$ 和特征图$ \hat{U} $ 进行加权操作可自适应地选取合适的特征图。 -
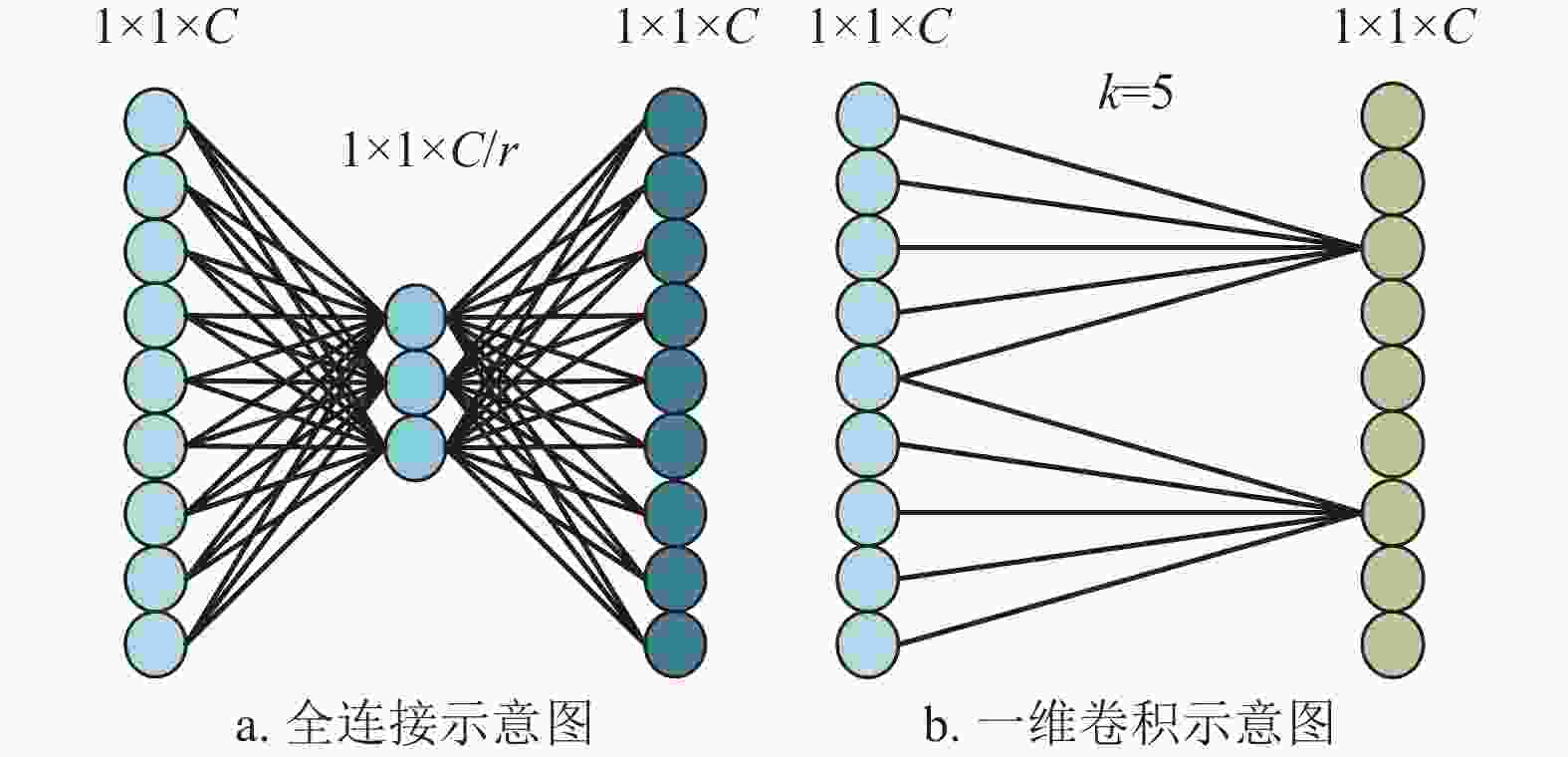
SKNet使用注意力机制改善网络性能,不可避免地会增加网络的计算复杂度。虽然在SKNet中使用先降维再升维的操作会降低复杂度,但由于该操作中使用全连接,网络的参数量依然庞大。此外,避免降维操作对学习通道注意力非常重要,适当的跨通道交互可显著降低模型复杂性的同时保持模型性能,ECA-Net采用一维卷积生成通道权重以避免捕获所有通道依赖关系导致的低效性[20]。
本文提出一个高效通道注意力(efficient channel attention, ECA)模块替换SKNet中的全连接层。在SKNet的基础上进行变更,对SKNet中先降维再升维的操作进行替换,实现一种不降维的局部跨通道交互策略。一维卷积能以极轻量级的方式捕获每个通道及其k个邻居的局部跨通道交互,故采用一维卷积替换SKNet的全连接层,如图3所示。ECA模块用于减少全连接层的计算参数量,通过ECA模块获得网络分支数对应个数的权重矩阵用于选择合适的特征图,可进一步降低网络参数数量,同时提升性能,将改进后的SKNet称为ECA-SKNet。
-
本实验的硬件环境为:Intel(R) Core(TM) i5-9300H 2.4 GHz处理器、16 GB内存、NVIDIA GeForce GTX 1660Ti 6 GB显存显卡。使用的系统为Windows 10,使用PyTorch深度学习框架进行卷积神经网络的构建和训练。实验采用VGG、ResNet、DenseNet、SKNet和ECA-SKNet进行训练,实验的超参数设置见表1。训练批量大小为16,训练周期为100,训练初始学习率为0.001,学习率每经过30个周期缩小到原来的0.1倍,优化器使用adam优化器,损失函数使用交叉熵损失函数。实验所使用的评价指标为准确率(accuracy)、精确率(precision)、召回率(recall)和F1分数(F1 Score),计算公式分别为:
$$ {\text{Accuracy }}= \frac{{{\rm{TP}} + {\rm{TN}}}}{{{\rm{TP}} + {\rm{TN}} + {\rm{FP}} + {\rm{FN}}}} $$ (1) $$ {\rm{Precision}} = \frac{{{\rm{TP}}}}{{{\rm{TP}} + {\rm{FP}}}} $$ (2) $$ {\rm{Recall}} = \frac{{{\rm{TP}}}}{{{\rm{TP}} + {\rm{FN}}}} $$ (3) $$ {\rm{F}}1{\text{ }}{\rm{Score}} = 2 \times \frac{{{\rm{Precision}} \times {\rm{Recall}}}}{{{\rm{Precision}} +{\rm{Recall}}}} $$ (4) 式中,TP表示正确识别单倍体的数量;TN表示正确识别二倍体的数量;FP表示不正确识别单倍体的数量;FN表示不正确识别二倍体的数量。
表 1 实验超参数设置
参数 值 批量大小/张 16 训练周期/次 100 初始学习率/% 0.1 学习率衰减率/% 10 学习率衰减周期/次 30 -
1)最优模型的确定
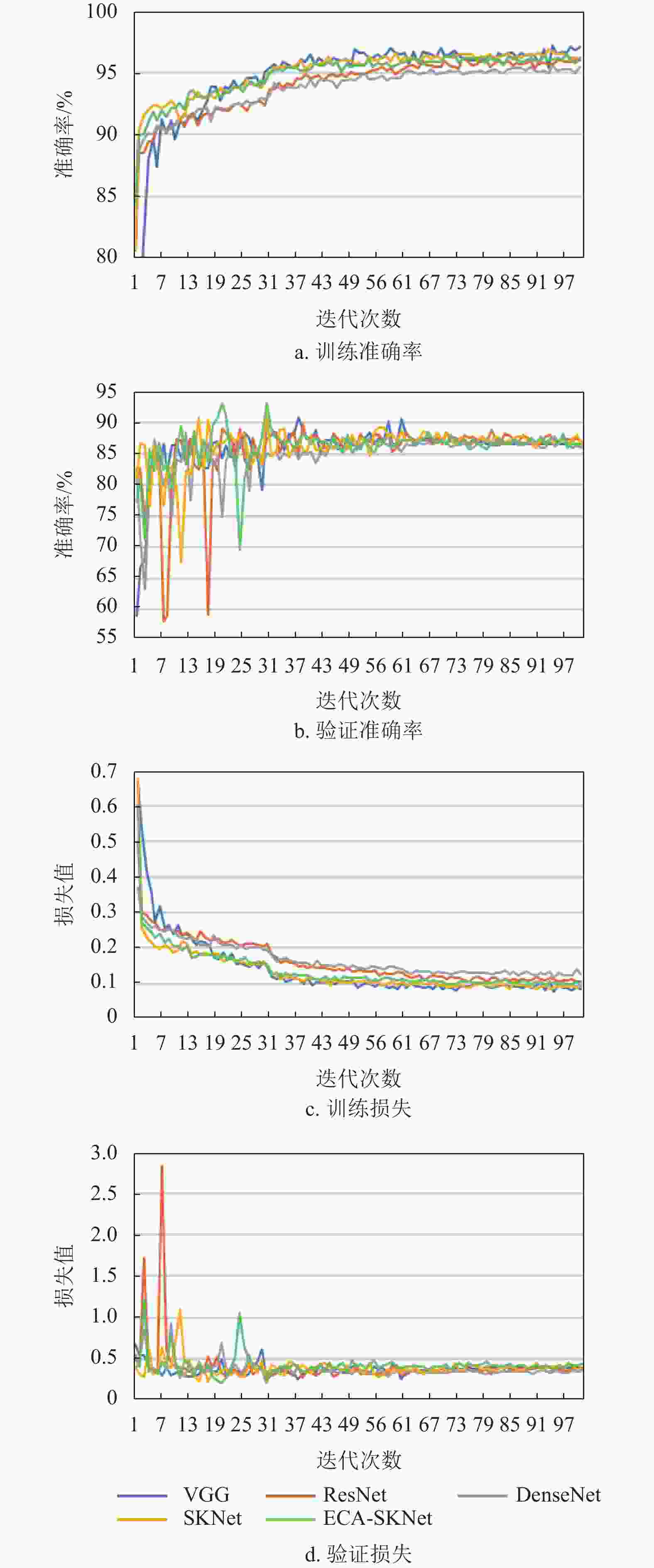
将玉米种子数据集按7:3的比例随机划分为训练集和验证集对VGG、ResNet、DenseNet、SKNet和ECA-SKNet进行训练,训练超参数使用表1中的设置,对于SKNet和ECA-SKNet的其他超参数,使用默认设置。各模型的训练准确率、训练损失、验证准确率、验证损失如图4所示,实验结果如表2所示。
由表2可知,实验模型对单倍体和二倍体玉米种子的分类精度达88%以上,其中VGG、SKNet和ECA_SKNet的分类精度达90%以上。表2的4个验证指标中,准确率为最重要指标,因为其表示该模型对单倍体玉米种子的筛选精度。可见,SKNet的实验结果为第二优结果,ECA-SKNet在准确率、精确率、和F1分数3项评价指标上均取得最好结果。
表 2 不同模型的训练结果
% 模型 准确率 精确率 召回率 F1分数 VGG 90.83 82.60 98.10 89.70 ResNet 89.88 81.40 97.60 88.80 DenseNet 88.50 82.20 91.80 86.70 SKNet 91.08 84.20 96.30 89.90 ECA-SKNet 91.38 85.10 95.70 90.10 实验结果表明,ECA-SKNet在对单倍体和二倍体玉米种子图像分类上具有较好效果,验证了ECA模块在避免降维和局部跨通道交互对学习通道注意的影响,即SKNet的改进能够进一步提升分类效果,故选定ECA-SKNet作为后续实验的模型。
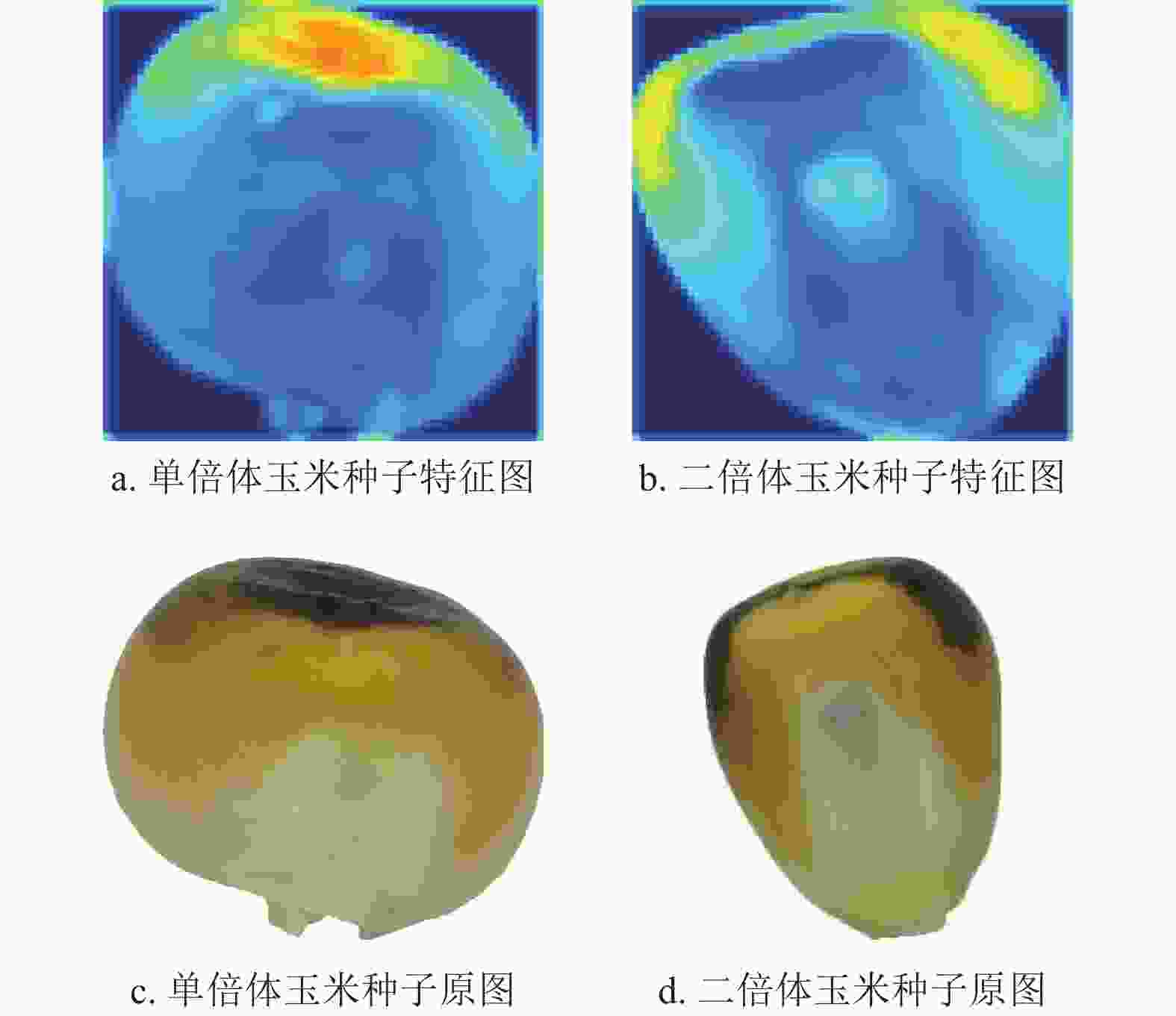
图5是ECA_SKNet模型在中间层提取出的特征图及其对应的原图。可见,ECA_SKNet关注玉米种子胚乳和胚胎的颜色特征。
2) ECA-SKNet的超参数调优
SKNet中使用分组卷积降低网络参数数量,ECA-SKNet也使用分组卷积。ECA-SKNet默认的分组卷积的组数为32,分别对组数为32、64和128的网络进行对比实验,实验结果如表3所示。
表 3 ECA-SKNet不同分组数的训练结果
% 分组数 准确率 精确率 召回率 F1分数 32 91.38 85.10 95.70 90.10 64 93.04 92.70 90.10 91.40 128 90.54 82.90 96.80 89.40 由表3可知,当分组数为64时,准确率为93.04%,精确率为92.70%,召回率为90.10%, F1分数为91.40%。分组卷积在相同的FLOPs下,分组数越大,通道上的密集卷积越稀疏,ECA模块可实现通道间信息的交互,促进模型精度增加。其中,分组数为64时在准确率、精确率和F1分数都获得最优结果,表明此模型对玉米种子的特征能进行更有效的提取和区分。
SKNet使用多分支进行自适应的特征选取,如图2所示为2分支的结构,在分组数为64的基础上,对分支数为2、3、4的ECA-SKNet进行对比试验,实验结果见表4。
表 4 ECA-SKNet不同分支数的训练结果
% 分支数 准确率 精确率 召回率 F1分数 2 93.04 92.70 90.10 91.40 3 92.88 88.70 94.70 91.60 4 89.46 80.20 98.70 88.50 由表4可知,当分支数为2时,准确率和精确率为最优结果。当分支数为3时准确率和精确率相较于分支数为2的模型略微下降,但F1分数为最优。当分支数为4时,准确率下降明显,模型效果与ResNet相当。
-
本文使用卷积神经网络对单倍体和二倍体玉米种子进行分类识别,对比了不同卷积神经网络对单倍体和二倍体玉米种子进行分类的效果,从中选出最优模型并进行超参数的调优,获得以下结论。
1)在VGG、ResNet、DenseNet、SKNet和本文改进的ECA-SKNet中,ECA-SKNet获得了最优结果。在未调优前ECA-SKNet获得91.38%的验证准确率。
2) ECA-SKNet使用分组数为64的模型能获得较大的性能提升,相比于默认32分组数量的模型准确率提高1.66%。分支数为2和3的模型性能相差不大,分支数为2的模型准确率和精确率较高,分支数为3的模型F1分数较高。
3)本文使用ECA-SKNet对单倍体和二倍体玉米种子进行分类的准确率达93.04%,为本研究中的最优结果。
ECA-SKNet: Convolutional Neural Network Identification Model for Corn Haploid Seeds
-
摘要: 采用3000张玉米种子图像进行基于卷积神经网络的玉米单倍体种子识别,包含1230张单倍体玉米种子图像和1770张二倍体玉米种子图像。为对比不同卷积神经网络模型在单倍体玉米种子识别的效果,使用VGG、ResNet、DenseNet和SKNet等经典模型,并对SKNet模型进行改进,将其降维升维全连接层设计为一维卷积以降低模型参数数量,改进后的SKNet称为ECA_SKNet。对5种模型使用相同优化器和训练周期进行实验,结果表明:实验模型均能对单倍体玉米种子达到较好的识别效果,最低准确率能达88.5%,ECA_SKNet模型准确率达93.04%。可见,卷积神经网络在玉米单倍体种子识别中能够发挥重要作用,为作物种子识别提供新思路。Abstract: In this paper, a study is conducted on corn haploid seeds recognition based on convolutional neural network using 3000 corn seed images with 1230 haploid corn seed images and 1770 diploid corn seed images. In order to compare the effect of different convolutional neural network models on haploid corn seeds recognition, classical models including VGG, ResNet, DenseNet and SKNet are adopted, and the SKNet model is improved by replacing the fully-connected layer in dimensionality reduction and dimensionality increase with one-dimensional convolution to further reduce the number of model parameters, and the improved SKNet is called ECA_SKNet. The experimental results show that aforementioned five models can achieve good recognition of haploid corn seeds with the lowest accuracy of 88.5% and the accuracy of ECA_SKNet can reach 93.04%. It is seen that convolutional neural networks can play an important role for the recognition of corn haploid seeds and provide a new way to recognize crop seeds.
-
Key words:
- convolutional neural network /
- corn seeds /
- deep learning /
- seed recognition
-
表 1 实验超参数设置
参数 值 批量大小/张 16 训练周期/次 100 初始学习率/% 0.1 学习率衰减率/% 10 学习率衰减周期/次 30 表 2 不同模型的训练结果
% 模型 准确率 精确率 召回率 F1分数 VGG 90.83 82.60 98.10 89.70 ResNet 89.88 81.40 97.60 88.80 DenseNet 88.50 82.20 91.80 86.70 SKNet 91.08 84.20 96.30 89.90 ECA-SKNet 91.38 85.10 95.70 90.10 表 3 ECA-SKNet不同分组数的训练结果
% 分组数 准确率 精确率 召回率 F1分数 32 91.38 85.10 95.70 90.10 64 93.04 92.70 90.10 91.40 128 90.54 82.90 96.80 89.40 表 4 ECA-SKNet不同分支数的训练结果
% 分支数 准确率 精确率 召回率 F1分数 2 93.04 92.70 90.10 91.40 3 92.88 88.70 94.70 91.60 4 89.46 80.20 98.70 88.50 -
[1] LIU J L, MA K J. Select inbred lines quickly for breeding by inducting monoploid[J]. Journal of Maize Sciences, 2003(z2): 70-72. [2] ROBER F K, GORDILLO G A, GEIGER H H. In vivo haploid induction in maize—performance of flew inducers and significance of doubled haploid lines in hybrid breeding[J]. Maydica, 2005, 50: 275-283. [3] SONG J Q, ZHANG P, LU D W, et al. Research progress of maize haploid breeding technology: Haploid induction and identification[J]. Bulletin of Agricultural Science and Technology, 2020(3): 4-9. [4] XING J F, ZHANG R Y, DUAN M X, et al. Discussion on problems for utilization of haploid breeding of maize[J]. Crops, 2012(2): 15-17. [5] GEIGER H H, ANDRÉS GORDILLO G, KOCH S. Genetic correlations among haploids, doubled haploids, and testcrosses in maize[J]. Crop Science, 2013, 53(6): 2313-2320. doi: 10.2135/cropsci2013.03.0163 [6] MELCHINGER A E, SCHIPPRACK W, FRIEDRICHUTZ H, et al. In vivo haploid induction in maize: Identification of haploid seeds by their oil content[J]. Crop Science, 2014, 54(4): 1497-1504. doi: 10.2135/cropsci2013.12.0851 [7] LI H G, LI W J, QIN H, et al. Classifying method of haploid and diploid based on least square error[J]. Transactions of the Chinese Society for Agricultural Machinery, 2016, 47(6): 259-264. [8] SONG P, WU K, ZHANG J X, et al. Sorting system of maize haploid kernels based on computer vision[J]. Transactions of the Chinese Society for Agricultural Machinery, 2010, 41(S1): 249-252. [9] ALTUNTAŞ Y, KOCAMAZ A F, CENGIZ R, et al. Classification of haploid and diploid maize seeds by using image processing techniques and support vector machines[C]//26th Signal Processing and Communications Applications Conference (SIU). Turkey: IEEE, 2018: 1-4. [10] ALTUNTAŞ Y, KOCAMAZ A F, CÖMERT Z, et al. Identification of haploid maize seeds using gray level co-occurrence matrix and machine learning techniques[C]//International Conference on Artificial Intelligence and Data Processing (IDAP). Turkey: IEEE, 2018: 1-5. [11] LIU W J, LI W J, QIN H, et al. Research on identifying maize haploid seeds using near infrared spectroscopy based on kernel locality preserving projection[J]. Spectroscopy and Spectral Analysis, 2019, 39(8): 2574-2577. [12] NANDA D K, CHASE S S. An embryo marker for detecting monoploids of maize (Zea Mays L.)[J]. Crop Science, 1966, 6(2): 213-215. doi: 10.2135/cropsci1966.0011183X000600020036x [13] LI X, WANG W, HU X, et al. Selective kernel networks[C]//Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. Long Beach: IEEE, 2019: 510-519. [14] HUANG G, LIU Z, VAN DER MAATEN L, et al. Densely connected convolutional networks[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. Honolulu: IEEE, 2017: 4700-4708. [15] HE K, ZHANG X, REN S, et al. Deep residual learning for image recognition[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016: 770-778. [16] SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[C]//International Conference on Learning Representations. San Diego: [s.n.], 2015. [17] PASCHALI M, SIMSON W, ROY A G, et al. Data augmentation with manifold exploring geometric transformations for increased performance and robustness[J]. Information Processing in Medical Imaging, 2019, 11492: 517-529. [18] TRAORE B B, KAMSU-FOGUEM B, TANGARA F. Deep convolution neural network for image recognition[J]. Ecological Informatics, 2018, 48: 257-268. doi: 10.1016/j.ecoinf.2018.10.002 [19] LIN M, CHEN Q, YAN S. Network in network[EB/OL]. [2022-10-22]. https://doi.org/10.48550/arXiv.1312.4400. [20] WANG Q, WU B, ZHU P, et al. ECA-Net: Efficient channel attention for deep convolutional neural networks[C]//IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Seattle: IEEE, 2020: 11531-11539. -

 ISSN
ISSN 




 下载:
下载: