-
乳腺癌已成为当前社会最常见的癌症之一,自20世纪70年代以来发病率一直处于上升趋势。在美国,约12%的女性会患乳腺癌疾病,仅在2017年就新增252 710个病例[1],由于乳腺癌的患病原因仍然未知,因此尽早发现是降低死亡率的关键[2]。乳腺癌的常用筛查方法包括影像检查和活组织检查(活检),活检花费高,并且痛苦,不太适用于早期筛查。在乳腺癌的医学影像诊断方法中,乳腺超声是无辐射、经济、高效的诊断方法[3]。乳腺超声成像旨在检测和分类良性或恶性肿瘤,乳腺超声的诊断,基于美国放射学会发布的乳腺影像报告和数据系统(BI-RADS)[4]。乳腺超声图像诊断的准确性取决于从业者的经验,需要训练有素且经验丰富的临床专业医生。此外,即使训练有素的专家也可能具有较高的观察者间差异。所以基于乳腺超声图像的计算机辅助诊断,一直是重要研究课题。
深度学习在很多领域都取得了卓越的成果,例如:汽车不安全行为识别[5]、无人机智能跟踪[6]、人体解析[7]等。这使我们能够重新思考基于乳腺超声图像的计算机辅助诊断方法。基于深度学习的方法要想达到较好的效果,需要大量准确标注的训练样本数据。然而,由于受到患者隐私和机构政策之类因素的限制,乳腺超声图像标注数据短缺是研究者面临的挑战之一。乳腺肿瘤诊断方法中只有活检才能最终确诊[2],并作为超声图像的准确标签。然而电子病历库中很多乳腺超声肿瘤图像没有活检结果[8-10],而只有基于BI-RADS的标签。BI-RADS评级分为0−6,不同评级按恶性概率区分:0级表示评估不完整,需要补充检查;1级表示无异常发现;2级表示良性,恶性概率为0%;3级表示恶性概率为0%~2%;4a级表示恶性概率为2%~10%;4b级表示恶性概率为10%~50%;4c级表示恶性率概率为50%~95%,5级表示恶性率为95%~100%;6级表示恶性概率为100%[4]。当医生为病人拍摄乳腺超声图像后,依据专业经验,给出每张图像的BI-RADS评级,当怀疑有恶性可能时,会推荐病人做活检进行确诊[11],这就导致很多低评级乳腺超声图像没有活检结果。另外评级高的部分病人可能会选择其他医院进行活检,而医院间的数据并不共享。这些原因导致了从一家医院很难收集大量有活检结论的乳腺超声图像。为了克服有准确标签乳腺肿瘤超声图像短缺的问题,一些先前的工作直接基于乳腺超声BI-RADS评级来生成乳腺肿瘤的良恶性标签。这些工作中直接将BI-RADS评级的2−4a(2、3、4a)作为良性肿瘤,BI-RADS评级的4b−6(4b、4c、5、6)作为恶性肿瘤[12-13]。然而BI-RADS评级只提供了乳腺肿瘤的恶性可能性,在机器学习领域,这个问题会导致噪音标签问题[14-15]。近年来,在机器学习领域,对训练带有噪声标签的深度模型为目标的弱监督分类方法进行了广泛的研究[16]。解决噪声标签的学习方法主要分为4类:基于损失的噪音鲁棒性方法[17-19]、噪音迁移预估方法[20-23]、样本选择方法[24-26]、标签修正方法[27-28]。
本文联合四川省人民医院进行基于BI-RADS评级的乳腺超声肿瘤图像分类研究,并收集标注了一个乳腺超声数据集。为了解决基于BI-RADS评级的乳腺超声肿瘤分类模型中的噪音标签问题,设计了一个协作标签修正网络(COLC-Net)。COLC-Net利用深度网络的“记忆能力”[29],即在训练初期优先记住特征明显或容易识别的样本特征,并结合乳腺超声图像的BI-RADS标签噪音率两端低、中间高的分布特点,为乳腺超声图像设计了软标签,在训练过程中进行标签修正,而不是将噪音标签视为固定值。为了通过网络参数和标签联合训练来优化网络,本文设计了双网络来协作修正标签。在训练过程中,从两个网络中蒸馏优秀知识修正软标签从而提升网络的泛化能力,随着软标签准确性的增加,可以降低噪音标签负作用,并增强准确标签知识的学习。
HTML
-
本文从四川省人民医院历史电子病历库,收集了一个乳腺超声图像数据集。这些乳腺超声图像是被GE-LOGIQ、Mindray和Philips IU-ELITE医学超声设备采集。原始超声图像包括一些无关信息,例如:医院名字、图像注释和相关参数,这些信息是无用的甚至会引入偏差。由于本研究主要关注肿瘤的良恶性分类,为了排除无关信息及脂肪等其他组织的干扰,对图像进行了手工预处理,即对图像进行裁剪,只裁剪出感兴趣的肿瘤区域,将其他无关信息删除。由于肿瘤区域不规则,本文选择的裁剪形状为长方形,长方形区域包含整个肿瘤区域。值得注意的是,所有图像的裁剪均由3位经验丰富的超声医生进行,并进行了交叉确认。
数据集依据是否有活检结论分为两部分:准确标签数据集和噪音标签数据集。准确数据集包括473个良性样本和462个恶性样本,准确标签数据集具有活检和BI-RADS两种标签。对于噪音标签数据集,参照以前的工作[12-13],使用BI-RADS评级来产生良恶性标签,将BI-RADS评级为2−4a(2、3、4a)的样本视为良性肿瘤,将BI-RADS评级为4b−6(4b、4c、5、6)的样本视为恶性肿瘤,噪音标签数据集包括577个良性样本和464个恶性样本。本文对样本进行随机划分,其中从准确标签数据集中随机取70张良性图像和66张恶性图像作为验证集,139张良性图像和133张恶性图像作为测试集。准确标签数据集的剩余部分按BI-RADS评级产生噪音标签,和噪音数据集一起组成训练集。所有训练集按BI-RADS定义的恶性概率平均值,生成软标签初始值。
-
定义
$X = \{ {x_1},{x_2}, \cdots ,{x_n}\} $ 表示具有n个样本的训练集,其中${x_n} \in {R^{W \times H \times 3}}$ 表示乳腺超声图像。定义向量${{l}} = [1,1]$ ,${{{l}}^{\rm T} }$ 为${{l}}$ 的转置,定义$H = \{ h:h \in {\{ 0,1\} ^2}, $ $h \times {{{l}}^{\rm T} } = 1\}$ 为基于BI-RADS评级的噪音标签空间,称为硬标签,标签${{{h}}_i}$ 可能和真实标签不一致。定义$S = \{ s:s \in {[0,1]^2},s \times {{{l}}^ {\rm T} } = 1\} $ 为基于BI-RADS评级的标签分布空间,称为软标签,它可以体现乳腺超声图像的类别相关性强弱。每个样本${x_i}$ 对应的软标签${s_i} \in S$ 不同于硬标签${{{h}}_i} \in H$ ,${{{h}}_i}$ 是一个可以表达真实类别标签的one-hot向量,软标签${s_i}$ 是标签分布,表示一个样本${x_i}$ 属于某个类别的概率。传统的监督学习方法基于硬标签进行学习,其优化问题如下:
深度学习训练过程会尽量拟合所有标签,在基于噪音标签的乳腺肿瘤超声图像良恶性分类问题中,深度学习网络模型拟合准确标签的同时也会拟合噪音标签。然而,噪音标签会引入偏差,对网络模型产生负作用。为了解决噪音问题产生的负作用,利用BI-RADS评级表达的是一张乳腺超声图像属于良恶性概率的特点,本文为每张超声图像生成噪音标签H的同时,生成一个软标签S。通过软标签和硬标签联合训练来优化网络,本文要解决的优化问题用公式描述如下:
软标签的初始值来自于BI-RADS评级。然而不同的BI-RADS评级给出的仅仅是一个概率范围,且评级边界较模糊,甚至存在由于医生经验而导致的误差。所以无法仅依据BI-RADS评级获得图像准确的软标签值。为了获得尽量可靠的软标签值,软标签在迭代过程中动态修正。所以本文的目标优化问题通过公式描述如下:
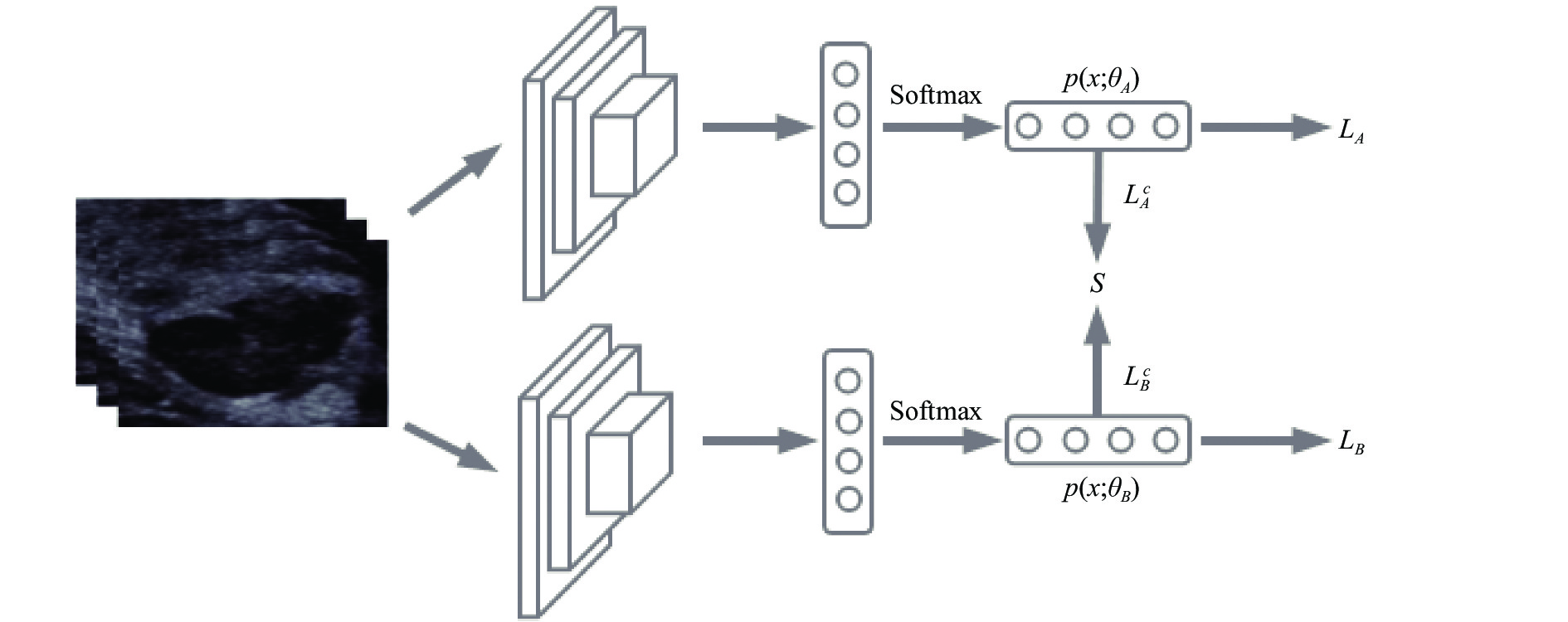
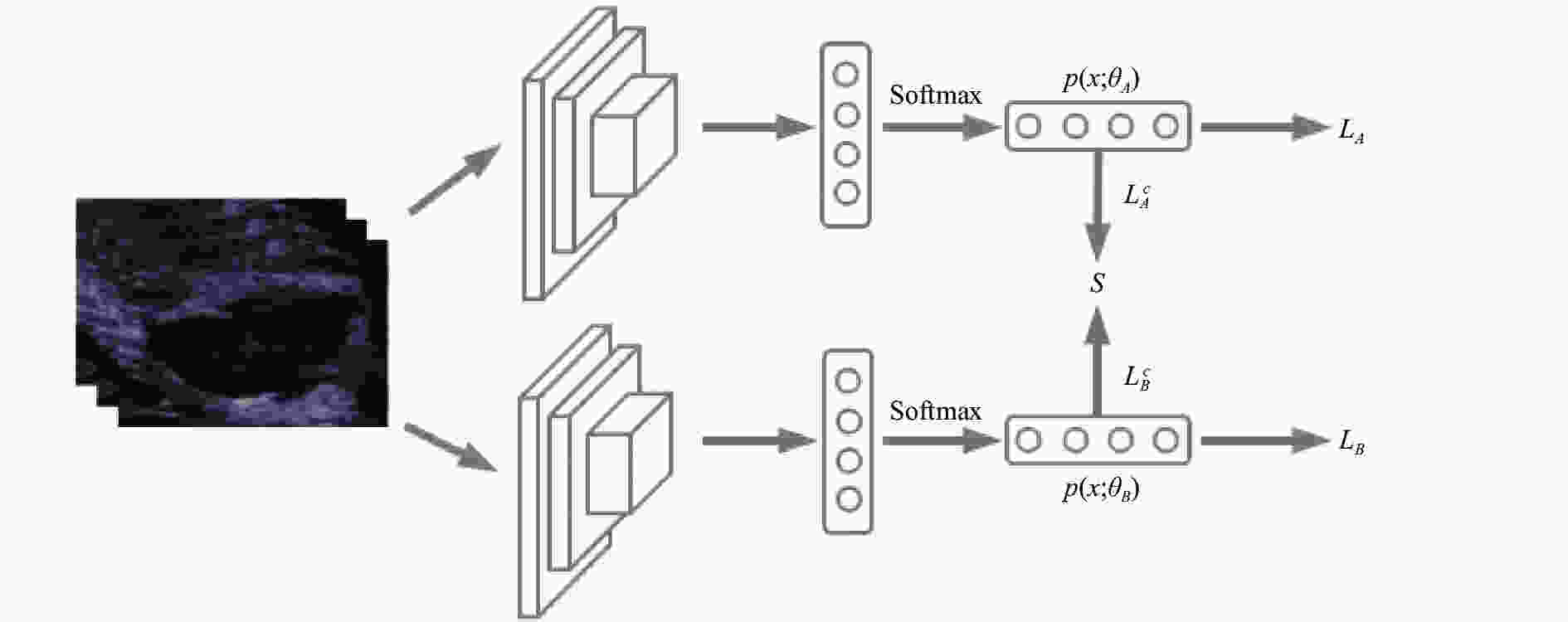
深度神经网络训练数据时,网络具有一定的随机性,对同一个训练批次的数据,两个分类器具有不同的学习能力,会产生不同的预测值。受此思想的启发,为了减少噪音标签的负作用,从而加强训练集中准确标签数据的学习,本文设计了一个针对标签修正的双网络协作学习框架,如图1所示。在此框架中,首先设计了基于硬标签的学习任务,定义网络学习参数为
$\theta $ ,分类器的预测输出为$p(x,\theta )$ ,用公式定义两个网络A和B的交叉熵损失如下:式中,
${h_{ij}}$ 是${h_i} \in H$ 的第$j$ 个元素;${\theta _A}$ 是A网络的学习参数;${\theta _B}$ 是B网络的学习参数。为了高效利用基于软标签S的特征知识,希望在训练过程中修正软标签,并让修正后的标签促进网络的整体学习能力。为了让软标签S的分布尽量准确,本文设计的学习策略为:软标签S从两个网络中蒸馏知识,随着S的能力增强,会约束训练数据集中标签的噪音,减弱噪音的负作用,并增强正确标签的学习能力。为了让软标签可以从两个网络中蒸馏出更优的知识,本文设计了基于软标签的协作损失函数,用Kullback-Leibler (KL)距离表示软标签的分类损失函数:
式中,
${s_{ij}}$ 表示样本${x_i}$ 对应于类别$j$ 的软标签分布;$L_A^c$ 表示网络A的软标签损失函数;$L_B^c$ 表示网络B的软标签损失函数。软标签修正方法中,定义大小为M的软标签容器。每个迭代基于硬标签分类器的预测输出被放入容器,当容器满后,最旧的值会被替换。软标签的更新规则如下:
式中,
$f(L_A^c,L_B^c)$ 表示训练过程中每个batch从$L_A^c$ 和$L_B^c$ 中取较小者的网络预测值放入软标签容器。综上所述,最终目标损失函数定义如下:
式中,α,β和γ是可调节超参数,用来平衡不同目标损失函数的权重。模型通过梯度下降算法来优化最终目标函数L。
1.1. 数据集
1.2. 方法
-
本文设计的所有实验是基于深度学习框架Pytorch,实验环境的GPU为NVIDIA GTX 1 080,操作系统为Ubuntu14.04。由于数据集较小,为了实验的公平性,所有对比实验中均采用AlexNet作为基础网络,并采用AlexNet基于ImageNet的预训练模型作为网络的初始值。实验过程中,训练阶段采用的优化器为Adam optimizer。为了适应Alexnet网络的输入,图片大小重置为227×227,并随机裁剪为224×224。超参数均按经验值设置,其中分类器初始学习率为0.005,网络其他参数的基础学习率为0.000 1,衰减周期为4。设置Momentum值为0.9,权重衰减值为0.001,批尺寸的值为16。最终目标损失函数的超参数α=0.5、β=0.5、γ=2。在标签更新过程中,软标签容器大小为2,前2个迭代使用初始软标签值,从第3个迭代开始使用修正后的软标签。在对比实验中,为了获得收敛结果,所有试验均进行了20个迭代。
-
为了验证COLC-Net算法的有效性,在收集的乳腺超声数据集上设计了充分的实验,并和最新的优秀方法进行了对比,对比方法的详情描述如下:
AlexNet:本实验使用的基础网络模型。
Co-teaching:文献[25]设计的有优异成绩的处理噪音标签的弱监督学习方法,此方法使用“最小损失”标准筛选的样本来交叉训练两个网络。
Decouple:文献[26]设计的一个非常简单的处理噪音标签的方法,此方法使用两个网络来进行训练,每个迭代只选择两个网络预测不一致的样本参与网络参数更新。
JOF:文献[28]设计的标签修正方,此方法通过最小化网络预测值和软标签值之间的KL距离来修正软标签值。
PENCL:文献[30]设计的标签修正方法,此方法是对JOF的改进,将软标签的更新融入反向传播和梯度下降迭代过程中。
COLC-Net:本文设计的协作标签分布修正方法。
为了实验比较的公平性,所有的比较方法均使用AlexNet作为基础模型。实验中恶性肿瘤是正类。表1为本文设计的COLC-Net和现有的最新优秀方法在准确率、精度、召回率和F1-Score上的对比。从实验结果的第一行可以看到由于数据有噪音,网络在学习过程中会拟合错误标签,AlexNet的表现并不能令人满意。第二行和第三行采用基于深度学习的样本选择方案来解决标签噪音问题,但是,与MINIST[31]等标准基准数据集不同,本文处理的乳腺超声图像类别之间的差异非常细微。因此,这两种比较的基线方法将遇到样本选择偏差的问题,样本选择偏差将导致误差在深度模型的训练过程中累积。第四行和第五行,采用基于深度学习的软标签修正方案来解决标签噪音问题,然而从结果来看,这两种方法的性能相比于AlexNet并没有明显提升。这是由于这两种方法的共同点是必须采用大学习率来训练,因为在大学习率下,网络不容易过拟合噪音标签。然而对于乳腺超声图像,图像类别区别度低,使用大学习率很容易陷入局部最优,影响其学习性能,所以JOF和PENCL并不适合处理此类问题。最后一行可以看到,本文方法的效果明显优于其他方法。
模型 准确率/% 精度/% 召回率/% F1-Score AlexNet 72.4 65.6 91.7 0.765 Co-teaching 74.6 69.8 85.0 0.766 Decouple 74.3 67.6 91.0 0.776 JOF 73.5 67.8 87.2 0.763 PENCL 71.7 65.1 91.0 0.759 COLC-Net 77.2 70.5 91.7 0.797
2.1. 实验细节
2.2. 结果和讨论
-
为了解决乳腺肿瘤超声图像标签不足的问题,本文针对依据BI-RADS评级生成的良性/恶性标签会引起标签噪音的问题进行研究,提出一种处理噪音标签的方法COLC-Net。由于乳腺超声图像BI-RADS评级表示的是肿瘤恶性可能性,天然适合改造为标签分布方式,进行噪音标签问题优化。COLC-Net利用深度网络的“记忆能力”,即在训练初期优先记住特征明显或容易识别的样本特征,并结合乳腺超声标签噪音率两端低、中间高的分布特点,为乳腺超声的BI-RADS评级设计了软标签。另外不同的分类器可以生成不同的决策边界,并具有不同的学习能力。为了对乳腺超声的软标签进行修正,COLC-Net采用网络协作框架来过滤标签噪音。训练中,每个批次对不同网络的预测值进行选择,采用类似软件工程中常用的“结对编程”方法,从两个网络中蒸馏优秀知识,并动态修正软标签来阻止噪音标签的拟合,强化准确标签的学习。本文方法主要针对乳腺超声图像的BI-RADS标签设计,在其他类似医学数据集也有潜在价值。然而已开源相关性较高的乳腺X光数据集[32],只给出了良恶性标签,未给出BI-RADS标签。其他开源医学图像,如结肠癌CT影像[33],和BI-RADS评级诊断方法明显不同。所以无法采用这些数据集对本文方法进行验证。基于此,在本文收集的数据集上对不同方法进行了对比实验,实验证实了本文方法在处理具有BI-RADS标签的乳腺超声图像分类问题上更加高效。

 ISSN
ISSN 



































 DownLoad:
DownLoad: