-
微阵列技术的快速发展积累了大量的基因表达数据。基因表达数据具有高维、小样本及不确定性的特点。用传统的统计分析方法与机器学习方法选择最佳基因时,往往陷入维数灾难的困境[1]。基因选择是从众多的基因中选择一个基因子集使得基因样本分类最优化。基因子集的评价依赖于具体的评价函数。根据评价函数的不同,基因选择方法主要分为两类:Filter方法(筛选器)[2]和Wrapper方法(封装器)[3]。Filter方法不依赖于具体的分类器,根据度量准则筛选出最优的基因子集。常用的度量方法有t检验[4]、信息增益[5]、距离度量[6]、相关性分析[7]等。依据上述度量方法评估每个基因或多个基因与类别的相关性,按照相关性从高到低排序,选择排在前面的少数基因作为最佳基因组。这类方法简单、时间复杂度低,但没有考虑基因的分类性能,使得选择后的基因子集冗余度高,分类精度不是特别理想。Wrapper方法以分类精度为评价标准,在所有的基因子集中搜索,以分类精度最高的基因子集作为基因选择的结果。按照搜索策略的不同,基因选择可分为前向选择[8]、后向删除[9]、启发式搜索[10]等算法。Wrapper方法获取的基因子集分类性能较好,冗余度低,但时间复杂度较高,存在过拟合的现象。
粒计算是智能信息处理的一种新方法,涵盖粗糙集[11]、邻域粗糙集[12]、模糊集[13]、商空间[14]、覆盖粗糙集[15]等理论,能够处理不同粒度层次上的不精确、不完整与不确定的数据。邻域粗糙集以δ邻域构造上下近似集来度量一个不确定性的集合。文献[16]提出了基于邻域粗糙集的邻域分类算法,并把该方法应用于特征选择领域[12]。文献[5]研究了模糊粗糙集的不确定性度量,并成功应用于癌症基因的选择。文献[17]研究了邻域粗糙集与神经网络模型,并用于基因表达数据的分类研究。文献[18]提出的鱼群算法具有并行性、跟踪性、随机性、简单性的特点,是一种解决全局优化问题的有效工具。这种方法模仿自然界鱼群觅食行为,采用自下而上的寻优模式,通过鱼群中各个体的局部寻优,使得全局最优值在群体中突现出来。
面对高维、冗余、不确定性的基因表达数据,需要降低基因数据的复杂性,建立具备并行计算能力的基因选择理论与方法。为此,针对基因数据分析系统存在的维数灾难与不确定性问题,提出了基于邻域粗糙集与鱼群智能的基因选择方法。采用邻域关系粒化连续型的基因表达数据,利用鱼群智能算法提高基因选择的并行处理能力与寻优能力,设计基于邻域粒化与鱼群智能的基因选择算法。在两个高维基因数据集上进行基因选择,并对选择的基因进行了分类实验。
HTML
-
对于广泛存在的连续型基因数据分析系统,引入邻域粗糙集模型[16]粒化连续型的基因数据,用于基因选择领域。
定义1 设五元组$ {\rm{IS}} = (U, A, V, f, \delta ) $为邻域基因表达数据系统,其中U为基因样本集,A表示有限个基因,$ V = { \cup _{a \in A}}{V_a}$ ,Va表示基因a的表达水平值域,$f:U \times A \to V$是一个信息映射函数,即对$\forall x \in U, a \in A$,有$f(x, a) \in {V_a}$,$\delta \in [0, 1]$为邻域粒化参数。
定义2 设五元组$ {\rm{IS}} = (U, A, V, f, \delta ) $为邻域基因表达数据系统,对于任一基因样本$x, y \in U$, 基因子集$B \subseteq A$,其中B={a1, a2,…, an},定义B上的距离函数${D_B}(x, y)$满足如下条件:
1) ${D_B}(x, y) \ge 0$,非负;2) ${D_B}(x, y) = 0$,当且仅当$x = y$;3) ${D_B}(x, y) = {D_B}(y, x)$,对称;4) $ {D_B}(x, y) + {D_B}(y, z) \ge {D_B}(x, z) $,三角不等式。
其中$ {D_B}(x, y) = {\left( {\sum\limits_{i = 1}^n {{{(|f(x, {a_i})-f(y, {a_i})|)}^p}} } \right)^{1/p}} $,当p=1时,称为曼哈顿距离,当p=2时,称为欧氏距离。
定义3 设五元组$ {\rm{IS}} = (U, A, V, f, \delta ) $为邻域基因表达数据系统,对于任一基因样本$x \in U$,基因子集$B \subseteq A$,定义x在B上的δ邻域$n_B^\delta (x)$为:
根据距离函数的定义,邻域$n_B^\delta (x)$满足性质:
1) $ n_B^\delta (x) \ne \phi $;2) $ x \in n_B^\delta (x) $;3) $y \in n_B^\delta (x) \Leftrightarrow x \in n_B^\delta (y)$;4) ${ \cup _{x \in U}}n_B^\delta (x) = U$。
定义4 设五元组$ {\rm{IS}} = (U, A, V, f, \delta ) $为邻域基因表达数据系统,任一基因子集$B \subseteq A$决定了一个邻域参数δ上的邻域关系${\rm{N}}{{\rm{R}}_\delta }(B)$:${\rm{N}}{{\rm{R}}_\delta }(B) = \{ (x, y) \in U \times U|{D_B}(x, y) \le \delta \} $。$U/N{R_\delta }(B)$构成了U的一个邻域划分,称其为U上的一簇邻域知识,其中邻域划分的子集称为一个邻域类或者邻域知识。上述邻域$n_B^\delta (x)$即为一个邻域类。
定义5 设$ {\rm{DT}} = (U, C \cup D, V, f, \delta ) $为邻域基因表达数据决策表,其中C为基因集合,其值为连续型的数据,邻域参数为δ,其邻域划分为$U/{\rm{N}}{{\rm{R}}_\delta }(C) = \{ {X_1}, {X_2}, \cdots, {X_m}\} $,D为决策分类信息,为离散型的数据,以等价关系划分为$U/D = \{ {Y_1}, {Y_2}, \cdots, {Y_n}\} $。
定义6 设$ {\rm{DT}} = (U, C \cup D, V, f, \delta ) $为邻域基因表达数据决策表,$\forall B \subseteq C$,$X \subseteq U$,记$U/{\rm{N}}{{\rm{R}}_\delta }(B) = \{ {B_1}, {B_2}, \cdots, {B_i}\} $,则称$ {B_*}{(X)_\delta } = \cup \{ {B_i}|{B_i} \in U/{\rm{N}}{{\rm{R}}_\delta }(B), $为X关于B的邻域下近似集,称$ {B^*}{(X)_\delta } = \cup \{ {B_i}|{B_i} \in U/{\rm{N}}{{\rm{R}}_\delta }(B), {B_i} \cap X \ne \emptyset \} $为X关于B的邻域上近似集。
定义7 设$ {\rm{DT}} = (U, C \cup D, V, f, \delta ) $为邻域基因表达数据决策表。定义D对C的邻域分类精度为${\gamma _C}{(D)_\delta } = \left| {{C_*}{{(D)}_\delta }} \right|/\left| U \right|$,其中|U|表示集合U的基数。
定义8 设$ {\rm{DT}} = (U, C \cup D, V, f, \delta ) $为邻域基因表达数据决策表,对$\forall b \in B \subseteq C$,若${\gamma _B}{(D)_\delta } \ne {\gamma _{B - \{ b\} }}{(D)_\delta }$,则称b为B中相对于D是必要的;否则称b为B中相对于D是不必要的。对$\forall B \subseteq C$,若B中任一元素相对于D都是必要的,则称B相对于D是独立的。
定义9 设$ {\rm{DT}} = (U, C \cup D, V, f, \delta ) $为邻域基因表达数据决策表,若$\forall B \subseteq C $,${\gamma _B}{(D)_\delta } = {\gamma _C}{(D)_\delta }$且B相对于D是独立的,则称B是选取的关键基因组,这一过程称为邻域基因选择。
性质1 设$ {\rm{DT}} = (U, C \cup D, V, f, \delta ) $为邻域基因表达数据决策表,若${B_1} \subseteq {B_2} \subseteq \cdots \subseteq C$,则$0 \le {\gamma _{{B_1}}}{(D)_\delta } \le {\gamma _{{B_2}}}{(D)_\delta } \le \cdots \le {\gamma _C}{(D)_\delta } \le 1$。
根据定义9可知,基因选择过程即是保持邻域分类精度不变的基因冗余降低过程,性质1说明邻域分类精度具有单调性的特点。关键基因组可能有多个,其中基数最小的为最优关键基因组,其冗余度最小。最优关键基因组的计算与搜索过程是一个典型的优化问题,可采用启发式搜索方式求解,但容易陷入局部最优。因此,下面引入鱼群智能优化原理,用于最优关键基因组的搜索过程。
-
基因表达数据集具有高维的特点,设基因表达数据集有n个基因,则基因的组合就达到2n种方式,搜索空间达到指数级别。采用穷举法搜索出最优的关键基因组,显然是不可行的。而启发式贪婪搜索方法却很容易陷入局部解。鱼群算法具有较好的全局寻优能力与优越的并行计算的特点[18],因此,有必要采用鱼群算法搜索出最佳的关键特征组。
鱼群算法是一种模拟鱼群觅食行为的群智能算法,主要涉及鱼群的3种行为:觅食行为、聚集行为与追尾行为[18]。
1) 觅食行为
鱼觅食时总是在自己可视的邻域范围内往食物浓度高的地方游动。觅食行为数学上表示如下:
式中,Xi表示一条鱼所处的i位置,代表目前的解;$ {X_{{\rm{next}}}} $表示鱼要选择的下一个位置,表示下一个更优的解;$ R(S) $表示随机移动步长;$ {\rm{F}}{{\rm{S}}_i} $表示位置i的食物浓度。如果满足$ F{S_j} > F{S_i} $,则鱼向食物浓度高的j位置的方向上游动一步,否则,向随机方向游动一步。
2) 聚集行为
鱼聚集时总是在自己可视的邻域范围内往鱼群的中心位置游动,条件是中心位置食物浓度高且并不拥挤。聚集行为数学上表示如下:
式中,$ {X_c} $表示鱼群的中心位置;$ {\rm{F}}{{\rm{S}}_c}$表示中心位置的食物浓度;η为拥挤因子;$ {n_s}/n < \eta $表示中心位置并不拥挤。
3) 追尾行为
鱼追尾时总是在自己可视的邻域范围内往最大食物浓度的鱼群追尾游去。追尾行为数学上表示如下:
根据以上描述的3种鱼群行为,每条人工鱼探索它当前所处的环境状况和伙伴的状况,从而选择一种更佳行为,人工鱼集结在几个局部极值的周围,最终,全局极值解突现出来。
-
将鱼群算法引入基因选择领域时,需要解决如何度量两个基因组集合之间的距离。为此,将基因组集合转化为二进制数,并引入汉明距离度量两个二进制数的距离,从而可以度量两个集合的距离。
1) 鱼群位置表示
基因表达数据分析系统有n个基因,则有2n种组合方式,每种组合用一个二进制数来表示,代表一条人工鱼的位置。因此,每条人工鱼的位置是一个n位的二进制数,当第i个基因被选中为关键基因时,则该二进制数第i位为1,否则为0。
2) 人工鱼之间的距离度量
每条人工鱼所处的位置用一个二进制数表示,则两个二进制数的汉明距离为人工鱼之间的距离。设X、Y为两个n位二进制数,代表两条人工鱼的位置,$ {x_i} \in X $表示X的第i位,$ {y_i} \in Y $表示X的第i位,$ \oplus $表示异或运算,则人工鱼之间的汉明距离定义如下:
设$ X = ({X_1}, {X_2}, \cdots, {X_m}) $表示m条人工鱼组成的鱼群,则该鱼群的中心位置定义如下:
3) 评价函数
基于鱼群优化的基因选择算法中,每条人工鱼分头并行去寻找最优基因子集。基因子集的评价采用邻域分类精度与基因子集长度的加权值作为评价函数,定义如下:
式中,|C|表示所有的基因个数;|R|表示选择的基因个数;$ \lambda \in [0, 1] $表示权重参数。
4) 搜索停止过程
最佳关键基因组的搜索过程是一个不断迭代的过程,每次迭代随机生成k条人工鱼,分头去寻找局部最优解,迭代一次完成后获得暂时的全局最优解,当迭代次数达到最大值或全局最优解连续3次迭代都不再进化时,搜索关键基因组过程停止,输出全局最优解。
-
根据邻域粗糙集理论和鱼群智能搜索原理,提出基于邻域粗糙集与鱼群智能的基因选择算法,具体描述如下:
算法1 NFSAGS (neighborhood and FSA based gene selection)
输入:基因表达数据集${\rm{DS}} = (U, C \cup D, V, f, \delta ) $,最大迭代次数${\rm{maxcycle}}$。
输出:最优关键基因组$R{\rm{min}}$及基因个数$ L{\min} $ 。
1) 初始化$R{\rm{min}} = C$, $L{\rm{min}} = {\rm{|C|}} $;
2) 对基因表达数据进行邻域粒化,形成粒域类,并计算邻域正域${\rm{PO}}{{\rm{S}}_C}{(D)_\delta }$;
3) 计算邻域分类精度${\gamma _C}{(D)_\delta } = \left| {{\rm{PO}}{{\rm{S}}_C}{{(D)}_\delta }} \right|/\left| U \right|$;
4) 若迭代次数t小于maxcycle或者未达到满意解,则循环执行如下操作:
① 产生k条人工鱼,${R_k} = \mathit{\Phi} $;
② 每条人工鱼分别随机选择一个基因${a_k}$,${R_k} = {R_k} \cup {a_k}$;
③ 每条人工鱼并行循环搜索下一个最佳基因:
a.每条人工鱼分别执行觅食行为${R_s} = {\rm{Search}}({R_k})$、聚集行为${R_w} = {\rm{Swarm}}({R_k})$和追尾行为${R_f} = {\rm{Follow}}({R_k})$;选择评价函数最大值${R_k} = {\rm{max}}({\rm{fitness}}({R_s}), {\rm{fitness}}({R_w}), {\rm{fitness}}({R_f}))$,并计算其邻域分类精度${\gamma _{{R_k}}}{(D)_\delta }$;
b.若${\gamma _{{R_k}}}{(D)_\delta } = {\gamma _{\rm{C}}}{(D)_\delta }$或者$|{R_k}| \ge L{\rm{min}}$,则第k条人工鱼结束其搜索过程;
④ 若${\gamma _{{R_k}}}{(D)_\delta } = {\gamma _{\rm{C}}}{(D)_\delta }$并且$|{R_k}| < L{\rm{min}}$,则更新全局解$R{\rm{min}} = {R_k}$,$L{\rm{min}} = {\rm{|}}{R_k}{\rm{|}}$;
⑤ 迭代数$t = t + 1$;
5) 输出最佳基因组Rmin和基因个数 $L{\min} $。
在算法NFSAGS中,主要涉及评价函数与邻域分类精度的计算,而这些计算与邻域类相关。文中采用文献[19]中Hash排序的方法计算邻域类,邻域类计算时间降为线性。除了邻域类的计算之外,外层循环还有迭代次数和人工鱼条数。因而,最坏情况下,NFSAGS算法的时间复杂度为$O(k * t * m * n)$,其中k为人工鱼的条数,t为迭代的次数,m为基因的个数,n为样本的个数。其中步骤3)过程可并行计算,因此,时间复杂度可降为$O(t * m * n)$。
2.1. 鱼群智能优化原理
2.2. 鱼群优化基因选择
2.3. 基于邻域粒化与鱼群智能的基因选择算法
-
为验证算法的有效性,实验分别采用文献[20]中的基于邻域粗糙集的基因选择方法(SGSA)和本文算法(NFSAGS)进行基因选择,并比较选择后基因的冗余度与分类效果。算法性能评估采用如下方法:1)冗余度的比较,评估算法的基因选择能力;2)分类精度的比较,评估选择基因的分类能力。基因数据集采用两个公开的基因表达数据集Colon和SRBCT。
1) 结肠癌数据集(Colon):该数据集共包含62例样本,其中40例为结肠癌组织样本、22例为正常组织样本,每例样本由2 000个基因表达数据组成。
2) 小圆蓝细胞肿瘤数据集(SRBCT):该数据集共包含63例样本,4种类别,其中EWS类23例,RMS类20例,NB类12例和BL类8例,每例样本由2 308个基因组成。
-
为了分析选择基因的冗余程度,定义冗余度来表示选择基因的精简程度,表示如下:
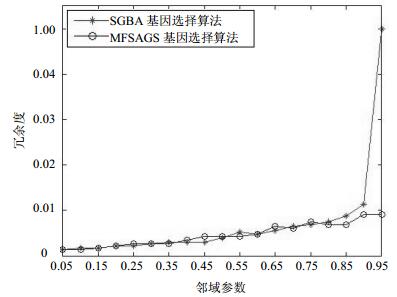
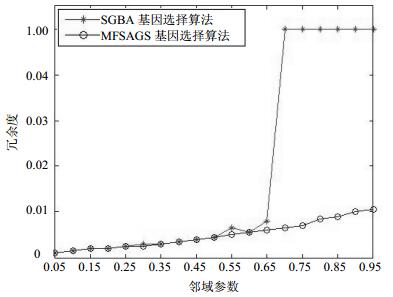
式中,|C|表示基因数据集的基因个数;|R|表示基因选择后的基因个数。冗余度越小,精简的效果越好。实验中,邻域粒化采用欧式距离,邻域粒化参数δ从0.05变化到0.95,每次变化的间隔是0.05。NFSAGS算法中人工鱼的个数为20,每条人工鱼向周围试探游动的次数为20,迭代次数为20。实验结果如图 1和图 2所示。
由图 1中的实验结果可知,在Colon基因表达数据集中,NFSAGS基因选择算法和SGSA基因选择算法都随邻域参数的增大,其选择基因组的冗余度逐步增大。NFSAGS算法在邻域参数δ从0.05变化到0.95区间,冗余度增加缓慢,而SGSA算法在邻域参数δ=0.7时,冗余度增大为1。由图 2中可知,在SRBCT基因表达数据集中,冗余度的变化也呈现类似图 1的特点,NFSAGS算法选择的基因组冗余度增加缓慢,而SGSA算法在邻域参数δ=0.95时,冗余度增大为1。冗余度越小,选择的基因个数越少,当冗余度为1时,全部基因是选择的基因。因此,实验表明基于鱼群智能的基因选择算法具有较好的基因精简效果和寻优能力。
-
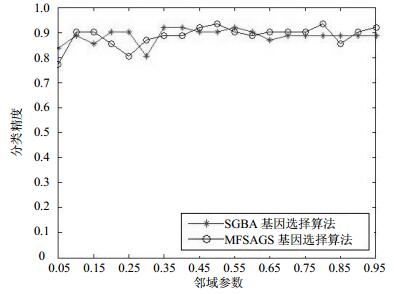
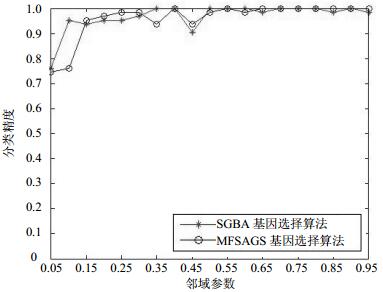
为了比较NFSAGS基因选择算法与SGSA基因选择算法中被选择基因的分类精度,采用SVM分类器进行测试。因基因数据具有样本少的特点,因此分类精度的计算采用留一交叉验证法。每次提取一个样本作为测试样本,将剩余的n-1个样本作为训练集,训练SVM分类器并用于测试提取的样本。然后,再轮流提取另一个样本,直到所有样本都测试一遍,最终的分类精度由分类正确的样本数与样本总数之比得到。实验结果如图 3和图 4所示,表示分类精度随邻域参数变化的曲线。
从图 3可知,在Colon数据集中,NFSAGS算法在邻域参数δ为0.5和0.8时分类精度达到最大值,为93.55%;而SGSA算法在邻域参数δ为0.35和0.4时分类精度达到最大值,为91.93%。在整个邻域参数变化期间,NFSAGS算法选择基因的分类精度有11次高于SGSA算法,8次低于SGSA算法。因此,在数据集Colon中,NFSAGS算法比SGSA算法能够获得更好的分类性能。
从图 4可知,在SRBCT数据集中,NFSAGS算法在邻域参数δ为0.4时分类精度达到最大值,为100%,这时选择出7个基因,这和文献[21]中的结果相同;SGSA算法在邻域参数δ为0.35时分类精度达到最大值,也为100%,这时选择出6个基因,略好于文献[21]中的结果。而在整个邻域参数变化期间,NFSAGS算法选择基因的分类精度有8次高于SGSA算法,5次低于SGSA算法,6次相同分类精度。从以上分析可知,NFSAGS算法和SGSA算法都具有较好的精简效果和分类性能,在整个邻域参数变化期间,NFSAGS算法的分类性能略好于SGSA算法。
3.1. 冗余度分析
3.2. 分类精度分析
-
本文将人工鱼群智能算法引入基因选择领域,并采用邻域粗糙集模型进行邻域粒化,在粒计算理论的框架中提出基于邻域粗糙集与鱼群智能的基因选择方法,并给出了适用于基因表达数据的基因选择算法。该算法充分利用邻域粗糙集粒化连续型数据的优势,发挥鱼群智能算法全局寻优及并行计算的特点,引入汉明距离度量人工鱼群之间的距离,给出了鱼群中心点的计算方法,并应用于基因选择研究。目前,采用鱼群算法的方法进行基因选择的研究还很少见,本文的研究拓展了粗糙集与软计算理论研究的应用范围,为基因选择研究提供了一条新的途径。理论分析及实验结果表明基于邻域粒化与鱼群智能的基因选择算法是有效可行的。

 ISSN
ISSN 









 DownLoad:
DownLoad: