Data Augmentation Algorithm for miRNA Omics-Based Classifications
-
摘要: 近年来,诸多研究揭示了miRNA的表达和疾病之间的关系,特别是其与肿瘤的发生、发展和治疗的密切关联。然而,传统的分子生物学测试方法既耗时又昂贵,患病样本获取困难,不平衡的数据集训练得到的分类器导致患病样本识别准确率低。面对以上挑战,提出了一种新的区分患病样本、健康样本以及挖掘疾病生物标志物的数据增强算法OCF,使用条件式生成对抗网络进行数据增强,然后用特征选择算法减少特征数量,最后再利用机器学习分类器进行分类识别并筛选出生物标志物进行分析。实验结果表明,该算法具有更好的分类性能,并验证了筛选出的生物标志物的准确性。Abstract: In recent years, many studies have revealed the relationship between microRNA expression and diseases, especially its close relationship with the occurrence, development and treatment of tumors. However, traditional molecular biology testing methods are time-consuming and expensive, and it is difficult to obtain disease samples. The classifier obtained from imbalanced data set training leads to low accuracy of disease sample recognition. In the face of the above challenges, we propose a new data augmentation algorithm OCF (original data-based conditional generative adversarial network for sample generation) to distinguish health samples from disease samples and mine disease biomarkers, by using conditional generative adversarial networks for data augmentation, followed by feature selection algorithms to reduce the number of features. Finally, the machine learning classifier is used for classification and recognition, and the biomarkers are selected for analysis. The experimental results show that our proposed algorithm has better classification performance, and verify the accuracy of the selected biomarkers.
-
微小RNA(microRNA, miRNA)是一种长度为20个左右核苷酸的非编码短RNA分子,是人类基因组编码的重要功能元件,在细胞发育和分化等过程中起重要调节作用。miRNA的发现揭示了一种新的基因调节机制的存在,并被验证在癌症发生、发育和转移等方面都发挥至关重要的作用。此外,miRNA被认为是识别各种不同癌症类型的潜在生物标志物。文献[1]研究表明miRNA可在血清中稳定存在,与组织标本miRNA表达谱相比,血清miRNA具有微创采样、稳定性强、灵敏度高及便于连续监测等优势,成为疾病诊断和预后评估标记物的研究热点。近年来,越来越多的研究揭示了miRNA的表达和疾病之间存在紧密关联。如miRNA与甲状腺乳头状癌的发生、发展和转移关系紧密,在对甲状腺乳头状癌的特异性诊断、治疗及预后评估等方面具有广泛应用前景[2]。另有研究表明miRNA在心血管疾病发生早期的诊断和治疗方面也具有重要价值,miRNA表达的抑制不仅与心脏病有关,与心脏的发育和生长也相关联。由于miRNA在特定的细胞通路上具有特异性靶标,因此将其作为诊断的标记物或通过对它的操控以获得治疗作用,都具有较好的生物学机制支持和临床应用前景[3]。miRNA在血脑屏障和脑神经方面的调节和干预功能也获得了广泛的研究成果,据此构建的miRNA调控血管性痴呆病的分子交互网络,为进一步研究该疾病临床诊断方案和靶向治疗药物提供了理论支撑[4]。
但研究仍面临以下挑战:1)样本获取困难,导致可使用的样本数量少,同时样本的特征数量多,存在部分不相关的特征和冗余特征会降低模型训练的速度、提高计算复杂度,也会影响模型的泛化精度与准确率;2)数据集存在类别不平衡的问题,健康样本远远多于患病样本,训练得到的分类器模型会更侧重学习多数类即健康样本的特征,从而更准确地识别多数类,而忽略了少数类即患病样本的识别,因此得到的整体结果是不准确的。在现实生活中,准确识别患病样本往往更有意义,所以训练一个分类模型,可以正确识别少数类样本,从而保证最终的分类性能具有多类别的识别平衡性。
1. OCF算法
针对以上挑战,本文提出了数据增强和特征选择结合的算法OCF (original data-based conditional generative adversarial network for sample generation),首先以生成对抗网络[5]的变种条件式生成对抗网络作为数据增强模型,学习原训练集的分布,生成新的少数类样本,加入原不平衡训练集中,降低其不平衡程度。然后对增强后的训练集分析特征重要性进行特征选择[6],选择方差最大的50个特征来减少样本冗余特征和不相关特征,从而提高分类器模型的训练速度和准确率,最后从选择出的特征中找出疾病相关的生物标志物,算法流程如图1所示。
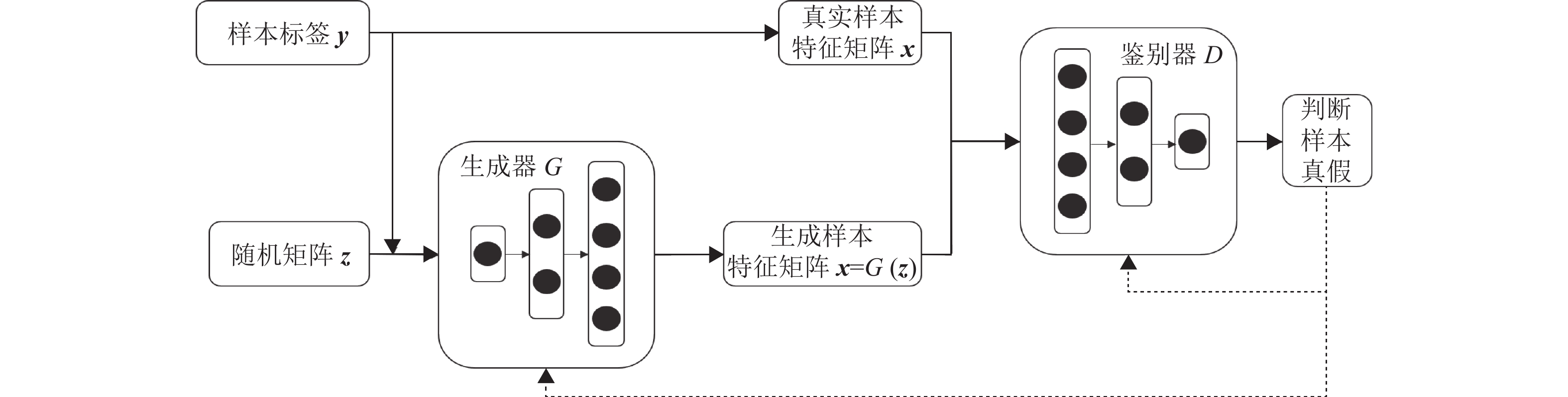
算法中的数据增强模块网络结构如图2所示,其中生成器和鉴别器均由3层全连接神经网络组成,模型参数如表1所示。将本文所用数据集按7∶3的比例划分为训练集和测试集,分别用每个数据集的训练集去训练鉴别器和生成器并保存训练得到的模型。样本标签y作为条件信息参与对生成器和鉴别器的训练,用真实样本和经生成器生成的样本去训练鉴别器。鉴别器的鉴别结果反馈给生成器,让生成器逐渐提升生成的样本质量,直到鉴别器无法区分传入的样本是真实样本还是生成样本时,结束训练。然后把需要的样本标签和数量传入已训练好的生成器,以生成所需的样本。本文提出的OCF算法及实验代码可以在如下网址下载:http://www.healthinformaticslab.org/supp/。
表 1 模型超参数设置超参数 取值 训练轮数/轮 200 生成器网络层数/层 3 生成器网络节点数/个 641, 1282, 2565 鉴别器网络层数/层 3 鉴别器网络节点数/个 2565, 1282, 641 2. 实验结果与分析
2.1 实验环境和模型参数
本文的实验环境使用了Python编程语言(版本3.6.13)、PyTorch框架(版本1.7.1)、numpy库函数(版本1.19.2)、pandas库函数(版本1.1.5)、sklearn库函数(版本0.24.2)。计算服务器的GPU加速显卡型号为TITAN RTX(24 GB显存),驱动程序版本455.45.01,CUDA 版本11.1。
实验数据增强模块的条件式生成对抗网络超参数的取值设置参见表1。
2.2 数据集和评价指标
本文使用3个结构化数据集,具体如表2所示,定义一个数据集的不平衡率为多数类样本数量与少数类样本数量的比值。
表 2 数据集信息数据集 样本数/个 特征数/个 不平衡率 GSE122497 5531 2565 8.772 GSE106817 3079 2565 8.622 GSE137140 3744 2565 1.391 数据集GSE122497是食管鳞状细胞癌的大规模血清miRNA谱组学数据,共有5531个样本。其中566个是食管鳞状细胞癌患病样本,剩下的4965个为非癌症对照样本。数据集GSE106817用于卵巢癌筛查的整合细胞外miRNA谱分析,包含 3079个样本,其中包括320个卵巢癌患病样本和2759个非癌症对照样本。数据集GSE137140使用血清miRNA的血液检测肺癌患者,包含3744个样本,其中包括1566个术前肺癌样本和2178个非癌症对照样本。
表2是本文使用的数据集,分别按7∶3的比例划分为训练集和测试集。本文所有实验的模型训练、数据增强和特征选择步骤均在训练集上进行。本文所获得的优化模型和特征子集,在没有变动的测试集上进行性能测试。
实验选择KNN[7]作为分类器,数据集划分为70%训练集和30%测试集,对于不平衡数据的分类问题,采用准确率(ACC)、ROC曲线下的面积(AUC)和 几何平均数(G-mean)作为评价指标。
2.3 消融实验结果展示和分析
为了验证本文算法模型OCF的有效性,以消融实验来验证各模块的必要性。原数据为未经处理的原始数据的训练集,原数据+特征选择为对未经处理的原始数据的训练集进行特征选择筛选出方差最大的前50个特征,原数据+数据增强为对未经处理的原始数据的训练集进行数据增强,使得训练集中多数类样本和少数类样本数量相同,实验结果均在独立测试集上取得。
数据集GSE122497和数据集GSE106817的消融实验结果如表3所示,本文提出的算法OCF在数据集GSE122497上的3个指标比原数据结果分别提升5.16%、5.21%和5.21%;在数据集GSE106817上时,3个指标也均取得了最佳结果,与原数据结果相比分别提升了6.71%、7.52%和7.52%。
表 3 数据集GSE122497和GSE106817消融实验结果方法 GSE122497 GSE106817 ACC AUC G-mean ACC AUC G-mean 原数据 0.9428 0.9435 0.9435 0.9156 0.9119 0.9119 原数据+特征选择 0.9307 0.9409 0.9408 0.9232 0.9362 0.9361 原数据+数据增强 0.9428 0.9435 0.9435 0.9156 0.9119 0.9119 本文方法OCF 0.9946 0.9956 0.9956 0.9827 0.9871 0.9871 数据集GSE137140的消融实验结果如表4所示,因为数据集GSE137140的不平衡率较低,为了验证本文算法的有效性和生成样本与真实世界数据的相似程度,随机选择原数据中20%少数类样本和100%多数类组成筛选数据,对其数据增强和特征选择后,再与原数据特征选择进行对比,实验结果表明,本文模型生成的新样本组成的数据集表现优于原数据集。
表 4 数据集GSE137140 消融实验结果方法 ACC AUC G-mean 原数据 0.8319 0.8532 0.8530 原数据+特征选择 0.9671 0.9591 0.9590 筛选数据 0.7109 0.8463 0.8354 筛选数据+特征选择 0.9484 0.9688 0.9686 筛选数据+数据增强 0.7109 0.8463 0.8354 本文方法OCF 0.9973 0.9987 0.9987 以上结果表明,数据增强和特征选择对分类器模型具有较好的性能提升,且二者联合使用会对原始模型的改进程度更大,从而证明了本文算法的有效性,可以更好地优化特征子集,筛选出针对目标问题更有意义的特征子集。
2.4 数据增强模块的对比实验结果
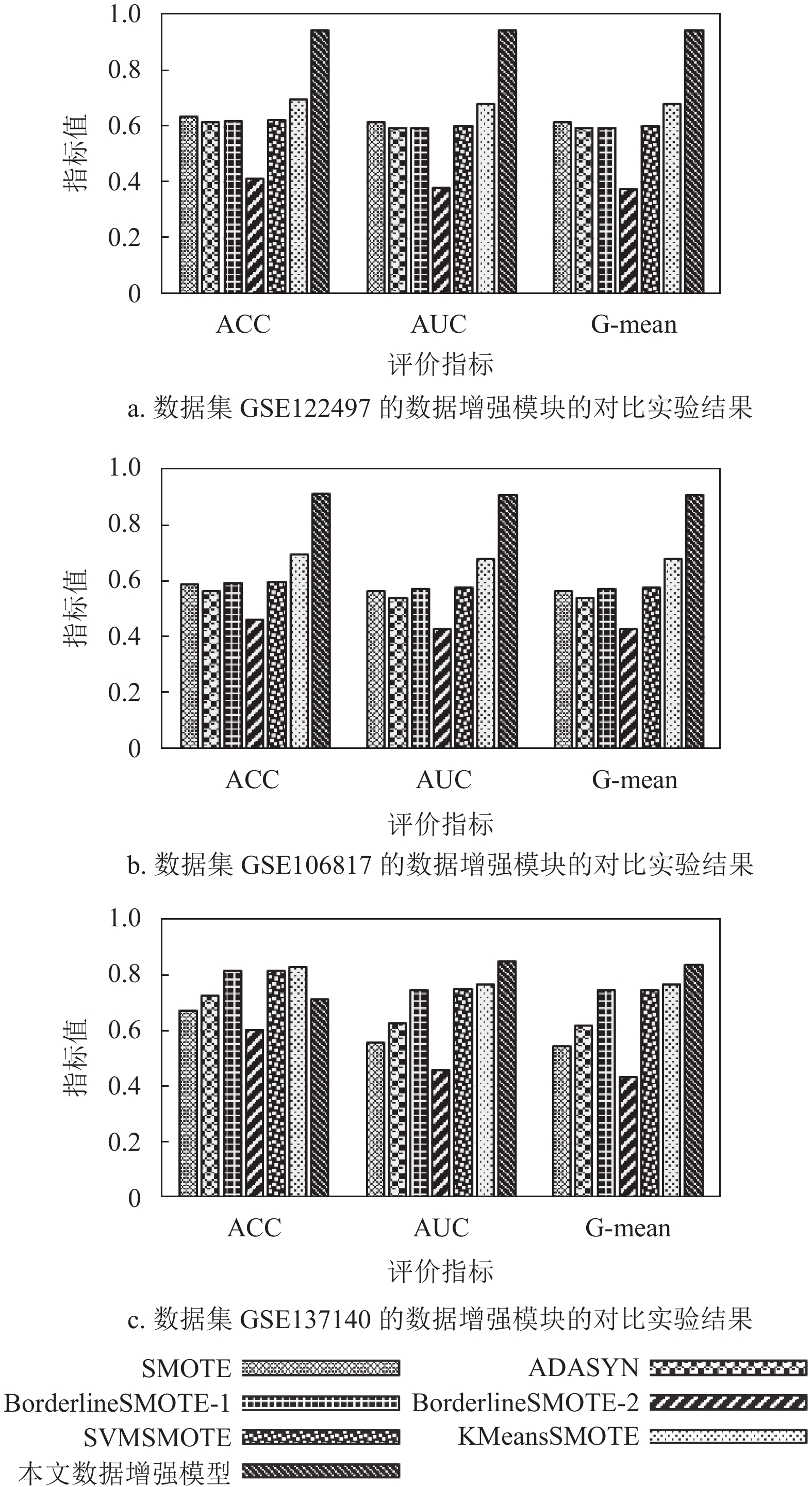
为了证明本文提出的OCF算法的有效性,和已有的其他面向不平衡数据分类问题的数据增强算法进行了对比。这些模型包括SMOTE[8]、ADASYN[9]、Borderline-SMOTE1[10]、Borderline-SMOTE2[10]、SVMSMOTE[11]及KMeansSMOTE[12]。实验结果都在独立测试集上取得,验证指标包括准确率(ACC)、ROC曲线下的面积(AUC)和几何平均数(G-mean)。图3为数据集GSE122497、数据集GSE106817和数据集GSE137140的数据增强模块的对比实验结果。
与6种流行的数据增强算法对比结果表明,除了数据集GSE137140的评价指标ACC未取得最大值,在其余数据集的各指标上本文算法均取得了最好的结果,提升明显。数据集GSE122497和数据集GSE106817的指标值都能达到90%以上,数据集GSE137140随机选择原数据中20%少数类样本和100%多数类组成筛选数据,本文算法在指标AUC和G-mean上都取得最大值,也可以证明本文算法训练出了高质量的分类器模型。
2.5 结合特征选择算法的对比实验结果
表5为数据集GSE122497、数据集GSE106817和数据集GSE137140的数据增强和特征选择结合算法的对比实验结果。6个数据增强算法结合特征选择算法之后指标值也均有所提升,本文算法在数据集GSE122497和数据集GSE137140上的3个指标的值均达到99%以上,在数据集GSE106817上的3个指标的值均达到98%以上,表现出了明显的优势。
以上两组对比实验的结果表明,本文提出的OCF算法中的数据增强模块在3个指标上都比其他数据增强模型表现好,结合了数据增强和特征选择的OCF算法均取得了指标的最大值,这足以证明本文算法的有效性。
2.6 生物标志物分析
用梯度提升决策树算法XGBoost[13]选出各个数据集重要性排名前20的特征取交集,对候选关键特征进行排序[14],取排名前5名的miRNA进行生物标志物分析,miRNA信息如表6所示。
文献[15]验证了hsa-miR-1228-5p作为生物标志物为肝癌诊断的高准确性,也可用于区分HCC患者与健康和肝硬化患者。文献[16]验证了hsa-miR-1228-5p为具有抗黑素瘤分化相关蛋白5抗体阳性亚群的皮肌炎相关间质性肺病的新型生物标志物。文献[17]确定了hsa-miR-4532为早期和快速识别COVID-19患者疾病进展的预测标志物之一。文献[18]的结论证明了hsa-miR-4532与糖尿病肾病相关,影响KCNJ11的表达和磺酰脲刺激的胰岛素分泌。文献[19]证明了hsa-miR-4532是在卵巢血清癌样本中受到调控的miRNA之一,为证明血清miRNA谱是卵巢癌的一个有前途的诊断生物标志物提供了重要证据。文献[20]表明hsa-miR-2861作为检测宫颈癌的新型非侵入性生物标志物。
表 5 数据增强和特征选择结合算法的对比实验结果算法 GSE122497 GSE106817 GSE137140 ACC AUC G-mean ACC AUC G-mean ACC AUC G-mean SMOTE 0.7964 0.7854 0.7854 0.7857 0.7751 0.7750 0.9528 0.9413 0.9412 ADASYN 0.8331 0.8246 0.8246 0.7641 0.7504 0.7503 0.9475 0.9309 0.9308 Borderline-SMOTE1 0.8392 0.8337 0.8337 0.8149 0.8121 0.8121 0.9555 0.9426 0.9425 Borderline-SMOTE2 0.7500 0.7371 0.7370 0.7435 0.7298 0.7297 0.8594 0.8089 0.8073 SVMSMOTE 0.8458 0.8407 0.8407 0.8268 0.8186 0.8186 0.9528 0.9413 0.9412 KMeansSMOTE 0.8108 0.8007 0.8007 0.8539 0.8472 0.8472 0.9609 0.9498 0.9498 本文方法OCF 0.9946 0.9956 0.9956 0.9827 0.9871 0.9871 0.9973 0.9987 0.9987 表 6 miRNA信息排名 特征 miRNA名称 1 MIMAT0005582 hsa-miR-1228-5p 2 MIMAT0019071 hsa-miR-4532 3 MIMAT0022946 hsa-miR-1237-5p 4 MIMAT0023712 hsa-miR-6087 5 MIMAT0013802 hsa-miR-2861 3. 结 束 语
本文提出了一种基于条件式生成对抗网络的数据增强和特征选择结合的算法,生成现实生活中难获取的少数类样本,降低了数据集的不平衡程度,减少了数据集的特征数量,去掉了冗余特征,筛选出对于目标问题更具备意义的特征子集,训练出优秀的分类器模型。实验结果表明,本文提出的OCF算法在各评价指标上都表现最好,可以准确地区分疾病样本和健康样本,并找出疾病相关的生物标志物。期望本文算法可以为早期的疾病诊断、预测和预防提供有价值的信息。
-
表 1 模型超参数设置
超参数 取值 训练轮数/轮 200 生成器网络层数/层 3 生成器网络节点数/个 641, 1282, 2565 鉴别器网络层数/层 3 鉴别器网络节点数/个 2565, 1282, 641 表 2 数据集信息
数据集 样本数/个 特征数/个 不平衡率 GSE122497 5531 2565 8.772 GSE106817 3079 2565 8.622 GSE137140 3744 2565 1.391 表 3 数据集GSE122497和GSE106817消融实验结果
方法 GSE122497 GSE106817 ACC AUC G-mean ACC AUC G-mean 原数据 0.9428 0.9435 0.9435 0.9156 0.9119 0.9119 原数据+特征选择 0.9307 0.9409 0.9408 0.9232 0.9362 0.9361 原数据+数据增强 0.9428 0.9435 0.9435 0.9156 0.9119 0.9119 本文方法OCF 0.9946 0.9956 0.9956 0.9827 0.9871 0.9871 表 4 数据集GSE137140 消融实验结果
方法 ACC AUC G-mean 原数据 0.8319 0.8532 0.8530 原数据+特征选择 0.9671 0.9591 0.9590 筛选数据 0.7109 0.8463 0.8354 筛选数据+特征选择 0.9484 0.9688 0.9686 筛选数据+数据增强 0.7109 0.8463 0.8354 本文方法OCF 0.9973 0.9987 0.9987 表 5 数据增强和特征选择结合算法的对比实验结果
算法 GSE122497 GSE106817 GSE137140 ACC AUC G-mean ACC AUC G-mean ACC AUC G-mean SMOTE 0.7964 0.7854 0.7854 0.7857 0.7751 0.7750 0.9528 0.9413 0.9412 ADASYN 0.8331 0.8246 0.8246 0.7641 0.7504 0.7503 0.9475 0.9309 0.9308 Borderline-SMOTE1 0.8392 0.8337 0.8337 0.8149 0.8121 0.8121 0.9555 0.9426 0.9425 Borderline-SMOTE2 0.7500 0.7371 0.7370 0.7435 0.7298 0.7297 0.8594 0.8089 0.8073 SVMSMOTE 0.8458 0.8407 0.8407 0.8268 0.8186 0.8186 0.9528 0.9413 0.9412 KMeansSMOTE 0.8108 0.8007 0.8007 0.8539 0.8472 0.8472 0.9609 0.9498 0.9498 本文方法OCF 0.9946 0.9956 0.9956 0.9827 0.9871 0.9871 0.9973 0.9987 0.9987 表 6 miRNA信息
排名 特征 miRNA名称 1 MIMAT0005582 hsa-miR-1228-5p 2 MIMAT0019071 hsa-miR-4532 3 MIMAT0022946 hsa-miR-1237-5p 4 MIMAT0023712 hsa-miR-6087 5 MIMAT0013802 hsa-miR-2861 -
[1] 王火强, 王奕然. miRNA标志物在临床检测的应用[J]. 中国医药导刊, 2022, 24(2): 127-130. DOI: 10.3969/j.issn.1009-0959.2022.02.005 WANG H Q, WANG Y R. Perspective of clinical diagnosis by miRNAs Biomarker[J]. Chinese Journal of Medicinal Guide, 2022, 24(2): 127-130. DOI: 10.3969/j.issn.1009-0959.2022.02.005
[2] 胡仿玲, 张思林, 余杰情, 等. microRNA作为甲状腺乳头状癌生物标记物的研究进展[J]. 临床耳鼻咽喉头颈外科杂志, 2018, 32(15): 1199-1202. HU F L, ZHANG S L, YU J Q, et al. Research progress of microRNA as a biomarker of papillary thyroid carcinoma[J]. J Clin Otorhinolaryngol Head Neck Surg(China), 2018, 32(15): 1199-1202.
[3] 齐秀丽, 徐莉, 韩丽丽, 等. microRNA-疾病诊断的潜在生物标记物[J]. 生物化工, 2016, 2(6): 72-74. DOI: 10.3969/j.issn.2096-0387.2016.06.024 QI X L, XU L, HAN L L, et al. microRNAs: Potential biomarkers for disease diagnosis[J]. Biological Chemical Engineering, 2016, 2(6): 72-74. DOI: 10.3969/j.issn.2096-0387.2016.06.024
[4] 孙孟艳, 秦合伟, 牛雨晴, 等. microRNA与其在血管性痴呆中作用的研究进展[J]. 解放军医学院学报, 2022, 11: 1198-1203. SUN M Y, QIN H W, NIU Y Q, et al. Research advances in microRNA and its role in vascular dementia[J]. Acad J Chin PLA Med Sch, 2022, 11: 1198-1203.
[5] GOODFELLOW I J, POUGET-ABADIE J, MIRZA M, et al. Generative adversarial nets[C]//The 2014 Neural Information Processing Systems. Montreal: NIPS Press, 2014: 139-144.
[6] 黄娜, 何泾沙, 吴亚飈. 恶意PDF检测中的特征工程研究与改进[J]. 电子科技大学学报, 2022, 51(5): 766-773. DOI: 10.12178/1001-0548.2021403 HUANG N, HE J S, WU Y B. Research and improvement of feature engineering for malicious PDF detection[J]. Journal of University of Electronic Science and Technology of China, 2022, 51(5): 766-773. DOI: 10.12178/1001-0548.2021403
[7] MAMUN A M, KAWSAR A, MINHTHANG B F, et al. Machine learning-based statistical analysis for early stage detection of cervical cancer[J]. Computers in Biology and Medicine, 2021, 139: 104985.
[8] CHAWLA N V, BOWYER K W, HALL L O, et al. SMOTE: Synthetic minority over-sampling technique[J]. Journal of Artificial Intelligence Research, 2002, 16: 321-357. DOI: 10.1613/jair.953
[9] HE H B, BAI Y, GARCIA E A, et al. ADASYN: Adaptive synthetic sampling approach for imbalanced learning[C]//The 2008 IEEE International Joint Conference on Neural Networks (IJCNN 2008). Hong Kong, China: IEEE Press, 2008: 1322-1328.
[10] HAN H, WANG W Y, MAO B H. Borderline-SMOTE: A new over-sampling method in imbalanced data sets learning[C]//Advances in Intelligent Computing, ICIC 2005. Berlin, Heidelberg: Springer-Verlag, 2005: 878-887.
[11] NGUYEN H M, COOPER E W, KAMEI K. Borderline over-sampling for imbalanced data classification[J]. Int J Knowl Eng Soft Data Paradigms, 2009, 3: 4-21.
[12] LAST F, DOUZAS G, BACAO F. Oversampling for imbalanced learning based on K-means and SMOTE[EB/OL]. (2017-11-02) https://arXiv.org/abs/1711.00837.
[13] LYU H, ZHANG Y, WANG J S, et al. iRice-MS: An integrated XGBoost model for detecting multitype post-translational modification sites in rice[J]. Briefings in Bioinformatics, 2021, 23(1): bbab486.
[14] 姚旭, 詹秀秀, 刘闯, 等. 基于复杂网络控制理论的肿瘤关键基因预测研究[J]. 电子科技大学学报, 2022, 51(1): 138-147. DOI: 10.12178/1001-0548.2021173 YAO X, ZHAN X X, LIU C, et al. Predicting the critical tumor genes based on complex network control theory[J]. Journal of University of Electronic Science and Technology of China, 2022, 51(1): 138-147. DOI: 10.12178/1001-0548.2021173
[15] GUAN X Y, TAN Y W, GE G H, et al. A serum microRNA panel as potential biomarkers for hepatocellular carcinoma related with hepatitis B virus[J]. PLoS ONE, 2014, 9(9): e107986.
[16] ZHONG D L, WU C Y, XU D, et al. Plasma-Derived exosomal hsa-miR-4488 and hsa-miR-1228-5p: Novel biomarkers for dermatomyositis-associated interstitial lung disease with anti-melanoma differentiation-associated protein 5 antibody-positive subset[EB/OL]. (2021-07-29). https://www.hindawi.com/journals/bmri/2021/6676107/.
[17] PARRAY A, MIR F A, DOUDIN A, et al. SnoRNAs and miRNAs networks underlying COVID-19 disease severity[J]. Vaccines, 2021, 9(10): 1056.
[18] CHEN Z R, HE F Z, LIU M Z, et al. MIR4532 gene variant rs60432575 influences the expression of KCNJ11 and the sulfonylureas-stimulated insulin secretion[J]. Endocrine, 2019, 63(3): 489-496.
[19] HAMIDI F, GILANI N, BELAGHI R A, et al. Exploration of potential miRNA biomarkers and prediction for ovarian cancer using artificial intelligence[J]. Front Genet, 2021, 12: 724785.
[20] ZHANG Y, ZHANG D, WANG F, et al. Serum miRNAs panel (miR-16-2*, miR-195, miR-2861, miR-497) as novel non-invasive biomarkers for detection of cervical cancer[J]. Sci Rep, 2015, 5: 17942.
-
期刊类型引用(0)
其他类型引用(1)

 ISSN
ISSN 



 下载:
下载: